Les médias se font l’écho des conséquences dues à la révolution technologique engendrée par l’évolution du séquençage haut-débit.
Qu’est ce que la biotechnologie peut faire pour vous ? Comment peut-elle repousser la date de péremption de vos artères ?
En effet, Arte (pour TF1, il va falloir attendre un épisode de Dr House traitant le sujet) diffuse un documentaire américain de moins d’une heure dont l’objectif est de présenter les évolutions technologiques dans le champ de la médecine personnalisée. Il s’agit tout d’abord de développer le concept pour le grand public (en s’appuyant sur des exemples où le mélodrame l’emporte un peu sur le débat bioéthique).
Ce document présente les acteurs majeurs de cette révolution biotechnologique qui sont tous américains (alors que, rappelons le, la plus grande plateforme de séquençage au monde est chinoise). Malgré quelques défauts, il s’agit d’un documentaire faisant tout d’abord oeuvre de pédagogie et développant des exemples plutôt précis comme autant d’arguments poussant à la large utilisation du séquençage haut-débit. Le documentaire vaut surtout pour l’accent mis sur la manière d’interpréter les résultats, sur la pertinence de ceux-ci mais tait les notions économiques liées à un futur marché qui s’annonce éléphantesque.
N’est ce pas un problème que votre génome soit, dans un avenir proche, séquencé, « interprété » et stocké par une firme privée ?
Le documentaire dont il est question est disponible ici-même (vous trouverez le synopsis dans les quelques lignes ci-dessous)
« LE DÉCRYPTAGE DU GÉNOME HUMAIN
D’ici peu, en échange de quelques centaines d’euros, chacun d’entre nous pourra obtenir un séquençage complet de son ADN. Des informations qu’il sera possible de lire, de stocker et de soumettre à l’analyse. Cette révolution est déjà en marche, ainsi que le montrent deux exemples spectaculaires : un patient cancéreux qui semble défier la mort et un malade de la mucoviscidose à présent capable de respirer normalement. Dans ces deux cas, les scientifiques sont parvenus à détecter et à neutraliser les anomalies génétiques à l’origine des maladies. Demain, grâce au décryptage du génome, il sera possible de faire un diagnostic personnalisé pour chaque individu et de mettre en place des traitements ultra personnalisés. Mais quelles sont les conséquences d’une telle révolution ? Connaître les maladies dont nous aurons à souffrir dans l’avenir, est-ce une bénédiction ou un fardeau ? Que se passerait-il si de telles informations tombaient entre les mains de compagnies d’assurance, d’employeurs ou de futurs conjoints ? Une chose est sûre : cette nouvelle ère, marquée par une médecine personnalisée s’appuyant sur nos gènes, est une réalité qui nous concerne tous. »
(Etats-Unis, 2012, 53mn)
ARTE F
Date de première diffusion : Jeu., 25 avr. 2013, 22h50
Date(s) de rediffusion : Dimanche, 5 mai 2013, 10h50
Pendant ce temps, de l’autre côté de l’Atlantique, Laurent Alexandre intervient dans l’émission grand public : ce soir (ou jamais !). Le débat avait pour thème l’obsolescence programmée. Ainsi au bout de 1h20 d’émission Frédéric Taddeï lance le débat sur l’obsolescence du corps humain. Après un extrait de l’assez mauvais film Repo Men de Miguel Sapochnik (réalisateur de certains épisodes de Dr House… c’est dire s’il doit s’y connaître en médecine personnalisée…), Laurent Alexandre, l’auteur du livre « la mort de la mort » et président de la société DNAVision, intervient ensuite. Son discours est clair et d’une grande pédagogie, il est peut être un peu dommage que l’exemple mis en avant fut le diagnostic prénatal non invasif de la trisomie 21 par séquençage haut-débit.
Evidemment très rapidement l’argument eugéniste se fait entendre. Finalement, l’intervention de Laurent Alexandre fait écho au documentaire américain.
Si les questionnements liés à la bioéthique sont au cœur du débat, rarement sont abordées les questions économiques et de confidentialité des données, associées à l’exploitation de nos données les plus intimes : la connaissance quasi-exhaustive de notre génome (et donc d’une partie de celui de nos descendants qui n’auraient rien souscrit).
Car depuis la publication de Gymrek et al., dans Science de janvier 2013 : Identifying personal genomes by surname inference, il semble difficile de soutenir l’anonymisation des données présentes dans les banques publiques. Il semble difficile d’imaginer que déposer son génome dans les banques d’une firme privée ne constitue pas un problème dont il faut d’ores et déjà prendre la mesure.
Car si tout le monde s’accorde sur les bienfaits potentiels de la médecine qui exploite la connaissance approfondie des bases nucléiques d’un patient ou d’un bien portant-futur mourant, actuellement le pouvoir est aux mains des firmes privées (Knome, 23andMe), qui se constituent des bases de données colossales. Ces bases de données hébergeant des quantités exponentielles de génomes humains (dans l’immédiat il s’agit plutôt de génotypes) permettront à leurs détenteurs de dominer un marché (voire plus !) qui s’annonce colossal…
 DNA-Vision est devenue en 2011, le prestataire européen en génomique avec la plus grande puissance de séquençage (quatre systèmes Solid, deux HiSeq2000, un GAIIx, tous deux d’Illumina, et deux 454-de chez Roche). La petite société issue de l’Université Libre de Bruxelles (ULB) et fondée en 2004 a bien grandi et est actuellement dirigée par Laurent Alexandre. Ce dernier est le créateur du site e-toubib, Doctissimo, aujourd’hui détenteur de 80 % de DNA-Vision, suite à la vente de son site internet à Lagardère qui a permis à l’ex-chirurgien de devenir riche… engrangeant plus de 139 millions d’euros suite à la transaction. Il faut dire que le docteur en médecine, chirurgien urologue, énarque et diplômé en sciences politiques a été à l’origine de plusieurs sociétés dans le domaine médical : Benefit, Medcost, Clinics et enfin le site préféré des hypocondriaques, Doctissimo. C’est avec pour idée principale d’appliquer le séquençage, en profitant de sa vertigineuse baisse de coûts, au champ de la médecine personnalisée que le médecin essayiste, auteur de « la mort de la mort » est devenu président de la société DNAvision. Même s’il est vrai que, l’encore assez nouveau président de DNAVision, préparant l’entrée en bourse de son entreprise, parle de médecine personnalisée, la société belge est connue pour sa maîtrise des technologies haut-débit et à titre d’exemple, pour son expertise du séquençage appliqué aux études métagénomiques (vous pouvez lire : Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa, PNAS, avril 2010).
DNA-Vision est devenue en 2011, le prestataire européen en génomique avec la plus grande puissance de séquençage (quatre systèmes Solid, deux HiSeq2000, un GAIIx, tous deux d’Illumina, et deux 454-de chez Roche). La petite société issue de l’Université Libre de Bruxelles (ULB) et fondée en 2004 a bien grandi et est actuellement dirigée par Laurent Alexandre. Ce dernier est le créateur du site e-toubib, Doctissimo, aujourd’hui détenteur de 80 % de DNA-Vision, suite à la vente de son site internet à Lagardère qui a permis à l’ex-chirurgien de devenir riche… engrangeant plus de 139 millions d’euros suite à la transaction. Il faut dire que le docteur en médecine, chirurgien urologue, énarque et diplômé en sciences politiques a été à l’origine de plusieurs sociétés dans le domaine médical : Benefit, Medcost, Clinics et enfin le site préféré des hypocondriaques, Doctissimo. C’est avec pour idée principale d’appliquer le séquençage, en profitant de sa vertigineuse baisse de coûts, au champ de la médecine personnalisée que le médecin essayiste, auteur de « la mort de la mort » est devenu président de la société DNAvision. Même s’il est vrai que, l’encore assez nouveau président de DNAVision, préparant l’entrée en bourse de son entreprise, parle de médecine personnalisée, la société belge est connue pour sa maîtrise des technologies haut-débit et à titre d’exemple, pour son expertise du séquençage appliqué aux études métagénomiques (vous pouvez lire : Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa, PNAS, avril 2010).
Revenons au moins un instant sur le séquençage haut-débit appliqué à la médecine personnalisée, vu comme le fait de rendre accessible au soignant l’information génomique de son patient.
A une époque où le Dossier Médical Unique (DMP) a grand mal à devenir une évidence, à une période où l’accès universel aux soins est de plus en plus remis en question pour des raisons de disette financière, l’accès à une archive de plusieurs Gigaoctets de données par patient semble peu crédible à court terme. Ajoutés à cela les problèmes d’éthique, de confidentialité de données et de formation du personnel soignant à l’interprétation de ces données et vous comprendrez que le rêve d’un Laurent Alexandre aura du mal à toucher la médecine de masse mais pourra séduire les cliniques privées qui échappent à la problématique d’un accès universalisé aux soins.
On voit qu’à l’instar du tourisme spatial, la cible ne semble pas vraiment être le chômeur en fin de droit. La médecine pour pas grand monde… puisque si un séquençage coûte de moins en moins cher, l’interprétation des données générées et l’intégration de ces données dans un réel parcours médical est encore inaccessible aux 99 % des personnes les plus pauvres des 10 % des pays les plus riches.
Malgré tout, la médecine personnalisée dépasse cette vision de l’ADN. En effet, la médecine personnalisée comporte plusieurs approches :
– la détermination du risque (un génotypage haut-débit chez 23andme ou un séquençage chez un autre quidam pour vous dire que vous avez 2,03 % de chance de plus que la moyenne d’être atteinte de la maladie d’Alzheimer)
– la médecine stratifiée où seuls les patients répondant positivement à l’administration d’une solution curative, minimisant les effets secondaires de celle-ci, seront traités.
– la thérapie génique (un peu passée de mode) pour laquelle il existe trois types de thérapies : la transfection permanente d’ADN, la transfection transitoire d’ADN, les thérapies portant sur l’ARN.
 Si le séquençage multiparallélisé de l’ADN a tendance à se démocratiser au sein des laboratoires de recherche et de certains laboratoires en usant comme diagnostic, il semble qu’un pas de géant reste à franchir pour que celui-ci transforme complètement notre vision de la médecine. La médecine personnalisée n’est cependant pas à remettre en question… il s’agit juste de lui donner un horizon réaliste et réalisable, sans quoi cette notion sera à ranger, comme beaucoup d’autres avant, aux rayons des mots clés usés par la mode.
Si le séquençage multiparallélisé de l’ADN a tendance à se démocratiser au sein des laboratoires de recherche et de certains laboratoires en usant comme diagnostic, il semble qu’un pas de géant reste à franchir pour que celui-ci transforme complètement notre vision de la médecine. La médecine personnalisée n’est cependant pas à remettre en question… il s’agit juste de lui donner un horizon réaliste et réalisable, sans quoi cette notion sera à ranger, comme beaucoup d’autres avant, aux rayons des mots clés usés par la mode.
 Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
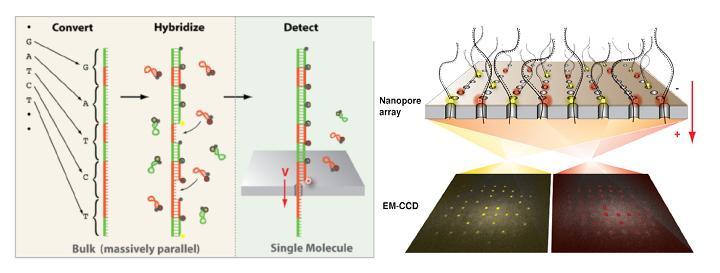
A l’instar du PGM Ion torrent, une puce en silicium constitue le coeur de la machine et renferme des centaines de nanopores.  La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
L’ensemble des études et preuves de faisabilité sur lesquelles repose la technologie de séquençage « optipore » sont décrites et détaillées au travers de l’article libre d’accès ci dessous: A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
Un soutien de 4,2 millions de dollars en septembre dernier de la part du National Human Genome Research Institute devrait permettre à la société américaine de conforter cette avancée prometteuse.
 L’exome humain, l’ensemble des exons ou encore l’ensemble de ce qui, encodé sous forme d’ADN, peut être traduit en protéines représente environ 1,2% genome humain, 50 Mb et inclus 85 % des mutations référencées.
L’exome humain, l’ensemble des exons ou encore l’ensemble de ce qui, encodé sous forme d’ADN, peut être traduit en protéines représente environ 1,2% genome humain, 50 Mb et inclus 85 % des mutations référencées.
Emilie Lalonde et al., montre l’efficacité du séquençage d’exomes dans un article paru dans Human Mutation de 2010. La démonstration est faite au niveau de la découverte rapide des mutations associées au syndrome de Fowler, impliqué dans la prolifération anarchique des vaisseaux du cerveau qui empêche son développement. Etudiant les exons de deux patients sans lien de parenté cette équipe de Montréal a démontré l’efficacité de la capture d’exons (SureSelect, Agilent) suivi d’un séquençage haut-débit sur plateforme Illumina GAIIx (en moyenne pour les 2 expériences ont été générés 70 millions reads de 76 pb, environ 5 Gbases de séquences brutes).
Techniquement le séquençage d’exons est relativement simple : il implique une capture par hybridation spécifique (NimbleGen, SureSelect de Agilent sont les plus employés), une purification de ces exons ou parties d’exons capturés et le séquençage haut-débit de ceux-ci… L’article de Majewski et al., disponible ici (J. Med. Genet. 2011) cerne avec brio le formidable potentiel de ces nouvelles études, avec son « qu’est ce que le séquençage d’exons peut faire pour vous ? ». Il est assez aisé de comprendre que le séquençage des régions codantes est plus accessible au niveau budget et souvent suffisant pour répondre à beaucoup de questions de cliniciens travaillant à la recherche des causalités de maladies rares. Le schéma ci-dessous, tiré de Leslie G Biesecker, Nature Genetics 42, 13–14 (2010), synthétise la procédure permettant d’aboutir rapidement à la notion de « gènes candidats » (ou plutôt de mutations causales). Les mutations témoins sont accessibles sur les bases de données mondiales dbSNP ou bien encore par l’intermédiaire du projet 1000 Génomes… dont nous avons parlé dans de précédents articles.

L’article montre à quel point l’investigation visant à la découverte des causalités d’une maladie rare, par exemple, a été totalement bouleversée. Ces technologies couplant capture d’exons et séquençage des régions codantes capturées ont d’ores et déjà montré la preuve de leur efficacité. L’article nous rappelle que le génome humain réalisé en 2001 a coûté plus de 2,7 milliards USD, qu’en 2008 un séquençage humain complet coûtait 1,5 millions de USD et qu’aujourd’hui ce même séquençage approche les 10 000 USD…
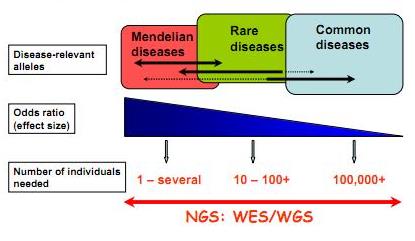
Feuille de route pour l'utilisation du séquençage haut-débit appliqué à l'identification des variations génomiques à l'origine de maladies "génétiques"
Ainsi que le montre Majewski et al. par le schéma ci-dessus plus la maladie que l’on cherche à élucider (si l’on peut s’exprimer ainsi) est la résultante d’une mutation qui a un effet fort, moins la cohorte à séquencer sera importante. En d’autres termes, pour ce qui concerne les maladies multi-factorielles (Alzheimer par exemple), le nombre d’individus dont les exons sont à séquencer sont importants… pour ce type de problématique les GWAS (Genome Wide Association Studies) par l’intermédiaire de scan de régions polymorphes anonymes sur la base de supports tels que les puces Illumina (puces permettant de scanner jusqu’à 4,3 millions de SNPs humains) restent compétitives et d’une puissance comparable. Dans le cas des maladies complexes d’autres stratégies sont développées telles qu’un premier crible sur une grande quantité de cas/témoins sur la base de puce haute densité de SNPs et un focus-séquencing des régions génomiques montrant une association potentielle avec le phénotype étudié…
Le séquençage haut-débit devenant de plus en plus accessible financièrement et par les méthodologies (pipeline d’analyses) de plus en plus disponibles permet d’envisager de nouvelles approches dans la découverte de cibles thérapeutiques potentielles. Ceci étant, l’accessibilité à des quantités de génomes de plus en plus disponibles (sans réel contrôle des organismes d’état) n’est pas sans soulever des problèmes d’éthique profonds et quelque peu négligé dans le débat scientifique actuel.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages