Pour rien vous cacher, il s’agit ici d’un article avec un petit goût d’auto-promotion. En effet, les 3 membres de Biorigami participent aussi activement à une plateforme d’expertise nouvellement créée, cette entité a pour ambition la réalisation de quelques prestations, principalement en génomique haut-débit, mais aussi la participation à des projets nécessitant conseils, expertises et analyses… pour répondre au mieux aux questions biologiques posées. Enchaînons en citant les pages actualités du site internet de Gènes Diffusion : « cette alliance entre les deux structures (ndlr : Gènes Diffusion et l’Institut Pasteur de Lille) a pris le nom de PEGASE (Plateforme d’Expertises Génomiques Appliquées aux Sciences Expérimentales). Elle permet de mutualiser les moyens de la branche R&D génomique de Gènes Diffusion et du laboratoire TAG (Transcriptomic analysis and Applied Genomics) de l’Institut Pasteur de Lille.
La création de cette unité devrait permettre le développement de nouvelles activités, collaborations scientifiques et prestations, dès les prochains mois
Issue de la collaboration entre Gènes Diffusion et l’Institut Pasteur de Lille, PEGASE (Plateforme d’Expertises Génomiques Appliquées aux Sciences Expérimentales) vient de lancer son site web. Cette présence sur le web permettra à la nouvelle structure de faire l’interface avec les différents acteurs de la génomique.
Par ailleurs, le site permettra également de faire partager à la communauté scientifique les différentes expertises en biologie moléculaire et bio-informatique issues des travaux de recherche de l’unité. »
Vous pourrez obtenir plus de précision concernant cette nouvelle plateforme en suivant le lien suivant : www.pegase-biosciences.com.
Pour les plus joueurs d’entre vous, un concours est lancé pour fêter cette naissance, le projet exécutable sur puce 318 de Ion Torrent, sera réalisé par nos soins, gratuitement… pour plus de précisions, nous vous invitons à suivre le lien suivant : concours PEGASE.
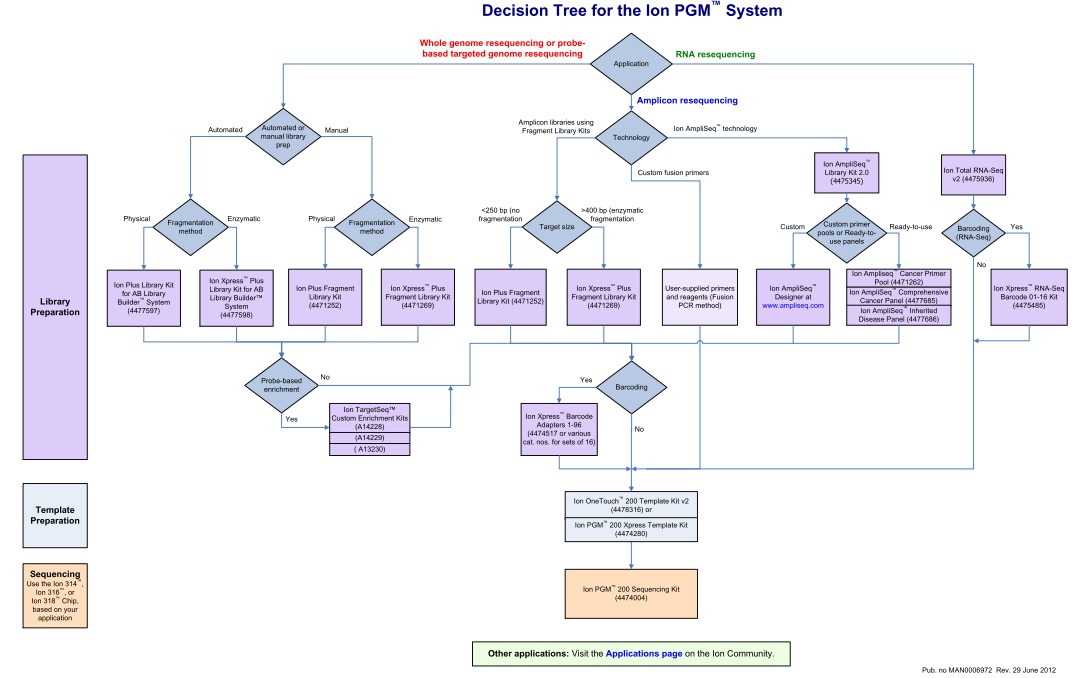
Un document qui n’est pas sans rappeler celui publié en novembre 2011, en intégrant les dernières mises à jour.
Cet arbre de décision est l’occasion de retrouver l’ensemble des modalités de séquençage qu’offre la technologie Ion Torrent via le PGM. En attendant l’arrivée du kit permettant de séquencer 400b, l’essentiel des modifications résident dans la préparation des échantillons avec notamment de nouvelles indexations (évolution du kit à 96 barcodes en DNA-seq et apparition du kit à 16 barcodes en RNA-seq), un élargissement du panel de primers dédiés à la recherche sur le cancer, etc…
Pour les privilégiées de la Ion Community, ce document est également l’occasion de retrouver aisément la liste des protocoles Ion Torrent selon l’ application d’intérêt en cliquant sur le lien en bas de page.
 Les outils d’assemblage, de mapping sur référence deviennent relativement pléthoriques… il est de ce fait relativement aisé de choisir l’outil permettant de gérer au mieux les défauts de votre séquenceur, vous n’avez que l’embarras du choix en somme. Même si cette étape n’est pas à négliger, il n’en reste pas moins que le rôle du biologiste restera toujours d’apporter du sens à une ou plusieurs millions de séquences, informations qui viendront tenter d’élucider une question biologique. Les outils permettant de faire parler un ensemble de reads, que vous pratiquiez le de novo RNA-seq ou séquençage génomique de novo sont plutôt restreints, peu diffusés, limités ou tout simplement absents… Ces grands absents constituent le point noir de l’analyse secondaire de vos reads. De nombreux biologistes recherchent un financement (séquencer massivement coûte encore un peu cher !), font séquencer l’objet de leur étude -hors humain, rat, souris- repartent quand tout va bien avec de beaux contigs, et se retrouvent face à un livre écrit dans une langue inconnue. Ce livre est censé renfermer la réponse à beaucoup de leurs questions… on comprend leur frustration d’autant qu’ils ne possèdent pas de pierre de Rosette.
Les outils d’assemblage, de mapping sur référence deviennent relativement pléthoriques… il est de ce fait relativement aisé de choisir l’outil permettant de gérer au mieux les défauts de votre séquenceur, vous n’avez que l’embarras du choix en somme. Même si cette étape n’est pas à négliger, il n’en reste pas moins que le rôle du biologiste restera toujours d’apporter du sens à une ou plusieurs millions de séquences, informations qui viendront tenter d’élucider une question biologique. Les outils permettant de faire parler un ensemble de reads, que vous pratiquiez le de novo RNA-seq ou séquençage génomique de novo sont plutôt restreints, peu diffusés, limités ou tout simplement absents… Ces grands absents constituent le point noir de l’analyse secondaire de vos reads. De nombreux biologistes recherchent un financement (séquencer massivement coûte encore un peu cher !), font séquencer l’objet de leur étude -hors humain, rat, souris- repartent quand tout va bien avec de beaux contigs, et se retrouvent face à un livre écrit dans une langue inconnue. Ce livre est censé renfermer la réponse à beaucoup de leurs questions… on comprend leur frustration d’autant qu’ils ne possèdent pas de pierre de Rosette.
Cet article part d’un constat, l’important est moins de savoir si votre séquenceur fournit les reads les plus propres, les plus longs, si votre assembleur est le plus adapté… car tous ces efforts resteront vains si vous écrivez un livre dont personne ne peut comprendre le sens. J’arrête là de filer la métaphore.

Blast2GO -ici dans sa version gratuite- est un outil qui trouvera vite (trop vite) ses limites mais qui permettra d’apporter du sens à des séquences générées à haut-débit. La publication de Conesa et al. (Bioinformatics – 2005), dévoile le pipeline d’analyse. Grossièrement, il s’agit de blaster des séquences (en 2005 – on envisageait par séquences des reads et non des reads assemblés) automatiquement sur le serveur du NCBI, les résultats de blast sont récupérés, les autres phases du pipeline sont réalisées en interrogeant les bases de données de Blast2GO, in fine des termes GO permettent d’annoter les séquences dont on souhaite décoder le sens. La principale qualité de Blast2GO réside dans son ergonomie, l’interface Java6 permet une prise en main directe (aucune ligne de code). Ses défauts sont multiples… tout d’abord Blast2GO plante… très régulièrement, ensuite pour une séquence d’entrée (dont la taille sera nécessairement inférieure à 7000 bases) vous ne disposerez que d’une annotation pour le meilleur des hits.
 Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le principe assez simple du séquençage SBS (Sequencing By Synthesis) est en train de se décliner au sein de machines haut-débit plus accessibles en terme de coût… il s’agit là de l’arrivée sur le marché d’une nouvelle concurrence : des séquenceurs génériques tels que ceux que proposent le distributeur Azco Biotec, Inc. Ces séquenceurs viennent directement concurrencer ceux d’Illumina accompagnés de leurs réactifs « génériques » tels que ceux distribués par la société Proteigene…
Ces séquenceurs « génériques » ne révolutionnent rien, leur part de marché est actuellement marginale… les laboratoires académiques préfèrent souvent l’original à la copie, malgré tout la concrétisation de la « promesse » –qui l’a formulée au juste ?– d’un génome humain pour 100 $ d’ici 2015 passe par la large diffusion de machines dont la technologie a fait ses preuves… Le retard pris par les séquenceurs de 3ème génération laisse le champ libre à la 2ème. Le marché risque de se saturer en séquenceurs de paillasse (séquenceur de 2ème génération tels que le MiSeq d’Illumina et les PGM et Ion Proton de Life Technologies).

Il est douloureusement fatigant de se rendre compte de l’absence de mémoire Humaine! Cette amnésie collective provoque les mêmes écueils, les mêmes cercueils. « L’Histoire est un éternel recommencement » cette sentence attribuée à Thucydide a beau être un cliché des sujets de philosophie, une partie de la Hongrie a voté pour l’extrême droite (troisième force politique du pays, troisième force politique de notre pays aussi, à en croire les résultats du premier tour de l’élection présidentielle) -cette même extrême droite hongroise se sert de test génétique humain en guise de néo-certificat d’aryanité. C’est ce que relate l’article de Charlotte Chabas du Monde (15 juin 2012) – à lire le test de « pureté génétique » qui choque la Hongrie. Des sociétés privées surfent sur la mode du test génétique, devenu désormais largement accessible pour qui veut (peut) débourser quelques centaines d’euros ou de dollars pour connaître ses origines ethniques (test de 23andme) pour le moins pire ou pour s’assurer, comme c’est le cas ici, de sa non appartenance à un groupe ethnique rejeté par une politique clairement xénophobe et antisémite. Ce certificat hongrois qui émeut une majorité de la classe politique de Budapest est exploité à des fins nauséabondes par sa franche la plus extrême. Un test dont le résultat annonce « Aucune trace génétique d’ancêtres juifs ou roms » peut faire frémir n’importe quel démocrate.
L’Histoire se répète donc, et au gré de l’avancée des connaissances et des technologies, des hommes éditent des certificats d’aryanités phylogénétiques ou encore (à l’inverse) marquent d’une étoile jaune génomique des personnes pour lesquels l’Humain n’est que le fruit de son hérédité biologique. La science raciste qui a justifié par le passé la colonisation, les pogroms, refait surface (avait elle seulement disparu) et on ne peut s’empêcher, en tant que scientifique biologiste, d’exhumer quelques tristes mots de James Watson (co-élaborateur du modèle en double hélice de l’ADN). Le scientifique alors âgé de 79 ans a déclaré dans les colonnes du Sunday Times qu’il était «fondamentalement triste au sujet du devenir de l’Afrique» parce «toute nos politiques sociales sont fondées sur le fait que leur intelligence (celles des Africains, ndlr) est identique aux nôtres (occidentaux, ndlr) alors que tous les tests ne le disent pas vraiment». La science et l’accès à la connaissance n’éloigne pas nécessairement les idées les plus abjectes, à chaque humain sa morale, à chaque scientifique son éthique… mais les partisans de la science avec conscience, celle censée élevée l’âme ne peuvent que s’indigner.
D’un 23andMe, présentant de manière positive l’accès démocratisé à la connaissance de ses origines par le biais d’études GWS (Genome Wide Scan), à un prestataire hongrois cynique s’abritant derrière des notions d’éthique pour diffuser des résultats de tests génétiques de « pureté raciale », l’accès à ce type d’informations peut être sujet à interrogations quant à leurs utilisations (personnelle ou politique). La diffusion de tels tests ouvre une boîte de Pandore; ces tests génétiques (interdits sur notre territoire national) offrent une arme aux pires propagandistes et une information sujette à interprétation et pouvant être manipulée pour le pire rarement pour le meilleur… l’Histoire est un éternel recommencement.

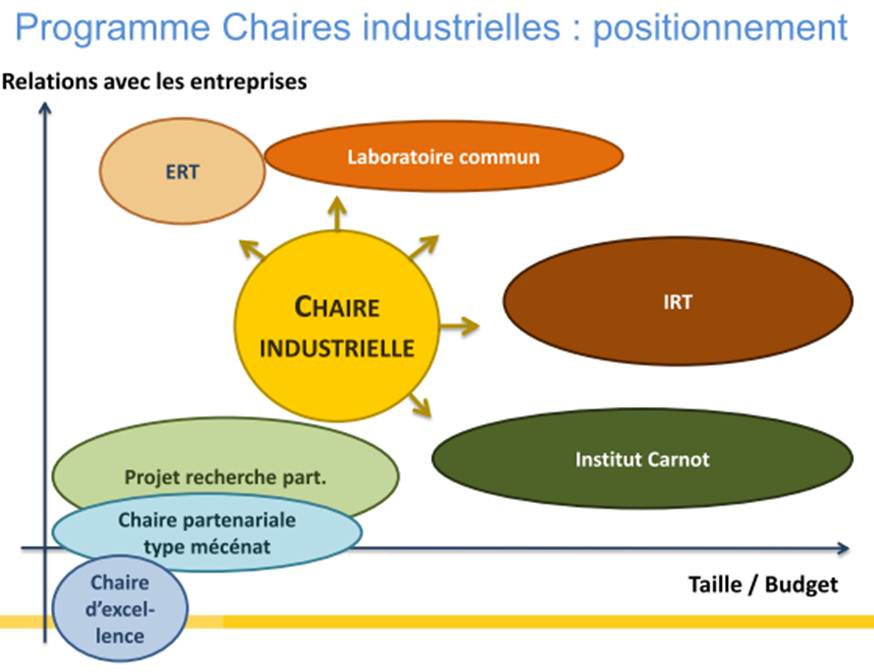
Lors d’une réunion le 21 mai dernier à Lille, Lionel Choplin, responsable du programme chaires industrielles venait présenter les ambitions de l’ANR pour associer le secteur privé –le co-financier et bénéficiaire des fruits de la valorisation de la recherche- et le secteur public –le pourvoyeur de cerveaux (de préférence provenant d’une grande université étrangère) et de structure d’accueil. Vous trouverez la présentation en suivant ce lien.. Ce programme est très largement inspiré du PCI canadien (Professeurs-Chercheurs Industriels) du CRSNG (Conseil de Recherches en Sciences Naturelles et en Génie du Canada) qui « offre » trois types de subventions :
- professeur-chercheur industriel principal pour les chercheurs principaux distingués (subvention renouvelable d’une durée de cinq ans);
- professeur-chercheur industriel agrégé pour les chercheurs en début de carrière qui démontrent un potentiel exceptionnel (subvention renouvelable une fois d’une durée de cinq ans);
- professeur-chercheur industriel cadre pour les professionnels exceptionnels de la recherche et développement (subvention non renouvelable d’une durée de cinq ans).
Dans sa version française, la subvention est clairement destinée aux deux premiers cas mentionnés ci-dessus. Ainsi la relation industriels-organismes de recherche devient plus monodirectionnelle. L’industriel lève des fonds (le ticket d’entrée étant de 600 k€ minimum sur 4 ans), l’ANR abonde d’autant (la stratégie du quitte ou double), la structure d’accueil hébergeant le titulaire de la chaire assure son bon fonctionnement (sachant que l’investissement en gros équipement est limité à 20 % de l’assiette). Le titulaire de la chaire ainsi que la structure d’accueil bénéficient de ce financement, l’industriel, quant à lui, aura un retour en terme d’image, de retombées sous forme de Propriété Intellectuelle et surtout en terme de connaissances sur un sujet qui est censé impacter sa croissance.
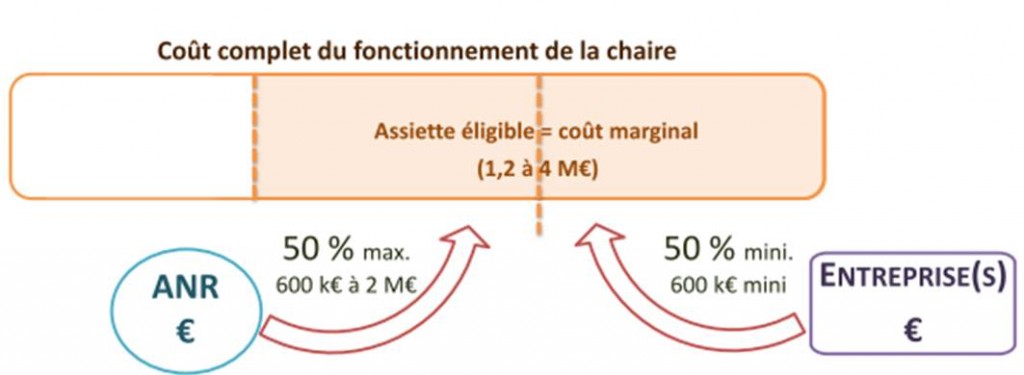
Le schéma ci-dessous reprend le positionnement relatif de la chaire industrielle par rapport aux autres programmes de l’ANR.
Par ce type de programmes, l’ANR vise plusieurs objectifs :
– Créer une dynamique à partir d’une offre de compétences et d’expertise en adéquation avec les besoins des acteurs du monde socio-économique
– Organiser les recherches dans une logique d’exploitation des résultats de recherche
– Insuffler au sein de l’établissement d’accueil une dynamique de recrutement de qualité dans un cadre de développement concerté fort
– Permettre un échange en temps réel de technologies et de savoir-faire entre les acteurs publics et privés de la recherche
– Assurer des retombées en termes d’image, résultant de l’accueil d’éminents enseignants-chercheurs au plan international
– Participation des acteurs publics au développement de produits innovants
– Mise en place de modèles économiques construits sur l’économie de la connaissance
Ce programme est comme beaucoup, critiquable, certainement imparfait mais il a le mérite d’afficher la volonté étatique de concilier secteur privé et secteur public, les industriels et les structures d’accueil… dans une relation plus équilibrée que de coutume afin que ces deux acteurs tirent bénéfice de cette relation. Rappelons ici que même aux États-Unis, souvent cités en exemple, les partenariats universités-entreprises restent modestes : 6 % des dépenses réalisées en 1999, au plus fort de la bulle Internet, depuis, cette part est redescendue à 4 %.
 Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Galaxy est en fait une brique d’une collection d’outils dédiés à l’analyse et au stockage de données biologiques : GMOD ( Generic Model Organism Database )
Le lundi 14 mai a été l’occasion pour nous d’assister à une conférence sur l’utilisation de certains des outils GMOD, dont voici les principaux enseignements :
> Le projet GMOD a pour objectif de fournir à l’utilisateur biologiste un ensemble d’outils interconnectés, libre de droit (open-source), générique (pour tous types de données biologiques) et facile d’utilisation (à travers des services Web principalement)
> Les outils GMOD sont développés (et donc installés) par des bioinformaticiens pour une utilisation par des biologistes.
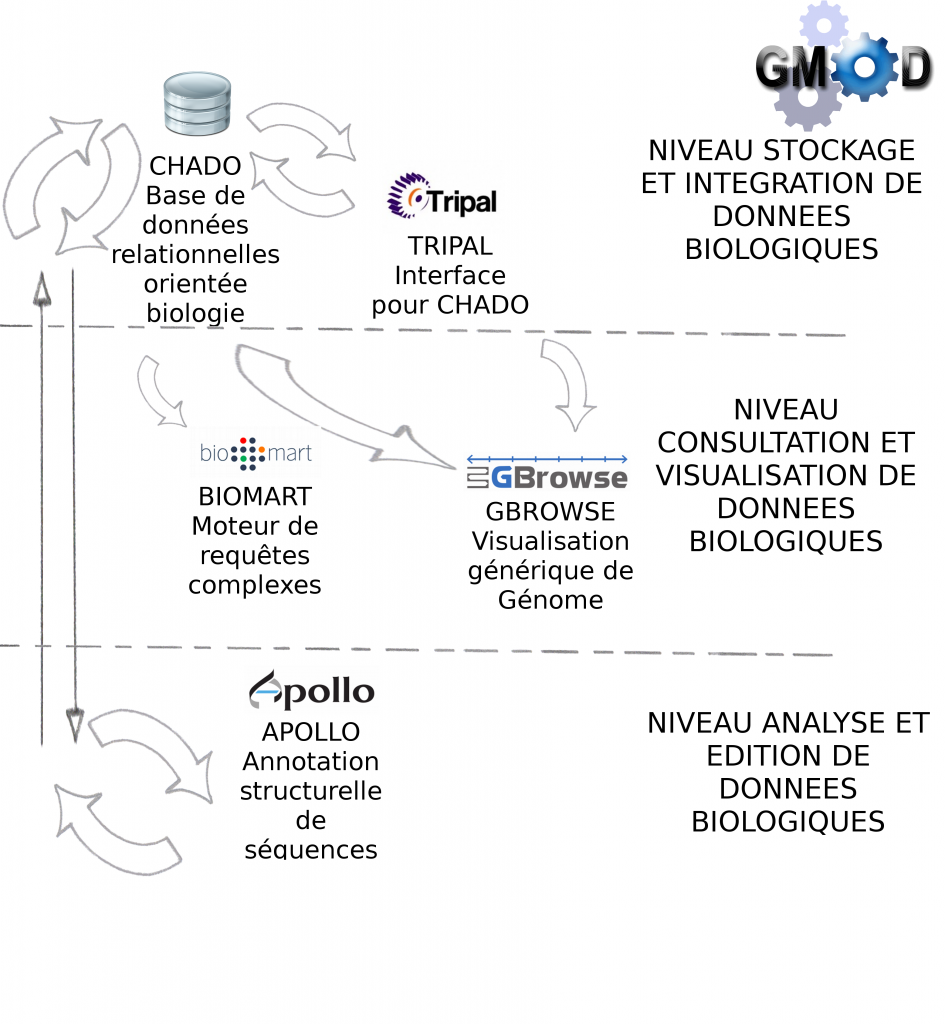
> Certains outils sont indispensables à GMOD, pour la manipulation de données génomiques, c’est le cas des outils Chado et Gbrowse qui sont respectivement les squelettes pour la manipulation et pour la visualisation des données biologiques.
Le schéma ci-dessous décrit les modules et interactions présentés lors de cette journée thématique :
> GBrowse est un outil de visualisation de données biologiques très puissant et certainement l’outil le plus populaire de la suite GMOD
Description détaillée : GMOD-GBROWSE
> Biomart est un outil de recherches avancées (ou requêtes complexes) pour la base de donnée relationnelle Chado
> Apollo est un module pour la correction manuelle d’annotation structurelle
Description détaillée : GMOD-APOLLO
> Tripal est une interface web développée en PHP pour interrogation de Chado
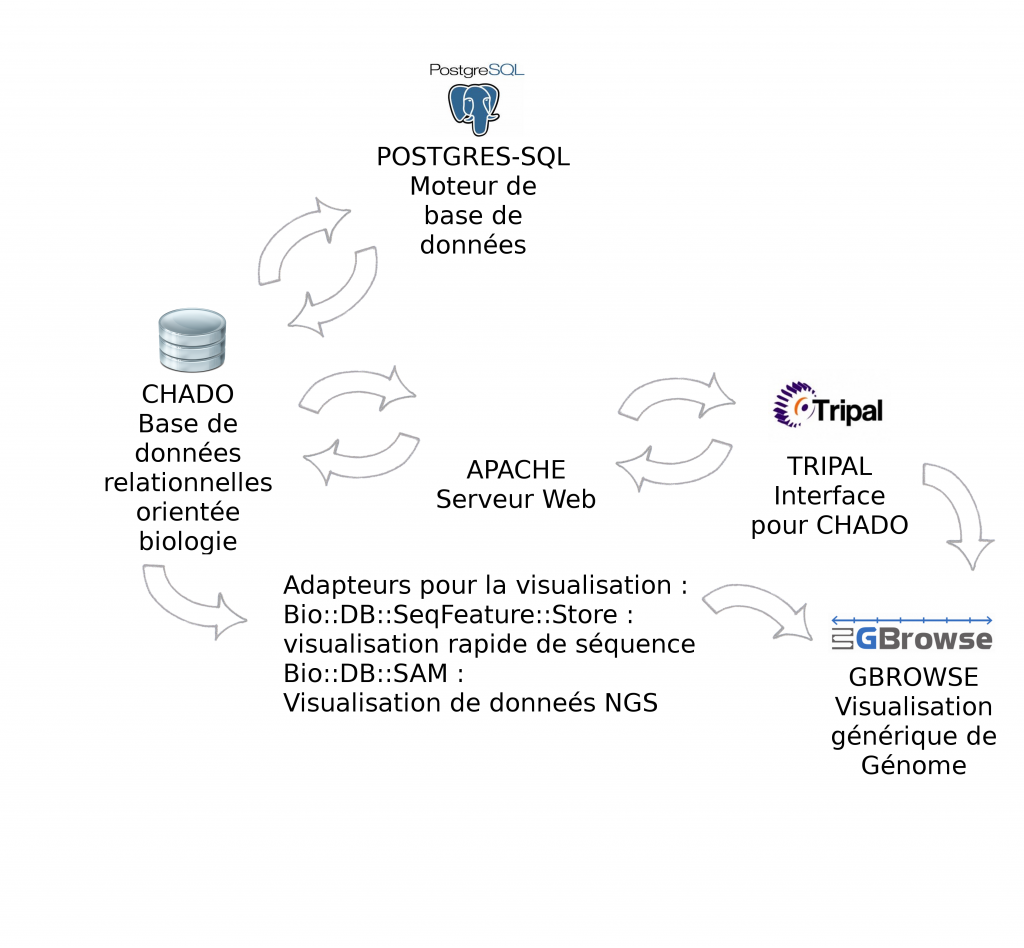
Environnement informatique : CHADO fonctionne par défaut avec Postgres-Sql. Pour l'interfaçage avec Tripal, la solution étant développée avec PHP, il est nécessaire d'installer un serveur Web Apache. Gbrowse peut s'utiliser à partir de l’interface Tripal mais également en "stand-alone". Afin d'accélérer la visualisation des données il est vivement recommandé d'utiliser les adapteurs Bio::DB::*, soit en relais entre Chado et Gbrowse (présentés dans cet article), soit en dupliquant les informations dans les deux bases de données (la visualisation ne se connecte qu'à Bio::DB sans passer par CHADO). Intuitivement, nous privilégions la première solution qui n’entraîne pas de duplication.
Pour conclure, GMOD propose un ensemble de modules pour la standardisation des processus bioinformatique : stockage et manipulation de données biologiques, visualisation et analyses avancées (assemblage, annotation…).
L’utilisation de tels outils (open-source) va dans le bon sens pour le partage scientifique et la standardisation des processus utilisés lors de l’analyse bioinformatique. Cela n’était pas le but de la conférence à laquelle nous avons assistée mais il serait également intéressant de connaître les conditions pour intégrer ces propres outils bioinformatiques en tant que brique GMOD.
GMOD est donc intéressant si :
> vous souhaitez stocker et gérer vos données biologiques
> vous cherchez des solutions d’analyses bioinformatiques déjà développées et robustes
Les biologistes sont les utilisateurs des outils GMOD, en revanche l’installation, l’administration et la formation des utilisateurs ne peuvent échapper à l’intervention, au moins ponctuelle, d’un bioinformaticien.
Si vous projetez d’utiliser ces outils, nous vous conseillons donc dans un premier temps, de regrouper l’ensemble des acteurs, installateurs comme utilisateurs de GMOD, afin de présenter les solutions offertes par l’outil et déterminer les besoins et objectifs pour votre propre utilisation.
Cet article est l’occasion de revenir sur un changement notable dans la politique d’évaluation de la recherche scientifique française, une politique légèrement moins ancrée sur la productivité bibliométrique, replacée dans l’écosystème économique et social. Une première réponse aux différentes critiques et notamment celle de Yehezkel Ben-Ari formulée dans sa tribune du Monde datée du 16 décembre 2011 : « Shanghai et l’évaluation technocratique tuent la recherche biomédicale ! ». L’évaluation étant le nœud gordien du problème un début de réponse peut être contenu dans cette reprise d’une dépêche de l’AEF. Celle-ci mérite d’être diffusée… et mérite aussi la faveur d’une opinion…
Dépêche n°166899, Paris, mardi 22 mai 2012
L’Aeres adopte six critères d’évaluation des entités de recherche, au lieu de quatre.
L’Aeres (Agence d’évaluation de la recherche et de l’enseignement supérieur) « a récemment étendu à six le nombre de ses critères » d’évaluation des entités de recherche, indique-t-elle dans un document intitulé « Critères d’évaluation des entités de recherche : le référentiel de l’Aeres », approuvé par le conseil de l’agence le 12 avril 2012 et rendu public mardi 22 mai 2012. Les nouveaux critères retenus sont les suivants : « production et qualité scientifiques ; rayonnement et attractivité académiques ; interactions avec l’environnement social, économique et culturel ; organisation et vie de l’entité de recherche ; implication dans la formation par la recherche ; stratégie et projet à cinq ans ». Ces critères seront mis en oeuvre « lors de la campagne d’évaluation 2012-2013 ». Ils se substituent aux quatre critères employés depuis 2008, qui étaient « la qualité scientifique et la production ; le rayonnement et l’attractivité ; la gouvernance et la vie de l’entité de recherche ; la stratégie et le projet ». Cette évolution vient en complément de l’abandon de la note globale, en décembre 2011 (AEF n°159690). L’agence avait alors indiqué vouloir « maintenir les notes associées aux différents critères », tout en affichant son intention de « réviser » ces critères, « pour les améliorer ».
Cette extension des critères d’évaluation répond à des améliorations qui ont été « suggérées dans deux domaines », explique l’Aeres. Il en va ainsi des « activités qui relèvent de la recherche finalisée » et des « activités d’expertise venant en appui des politiques publiques » : « Celles-ci n’étaient pas reconnues et valorisées par l’agence dans la déclinaison de son référentiel en quatre critères », indique-t-elle. « L’introduction du critère ‘interactions avec l’environnement social, économique et culturel’ (…) vise à les intégrer pleinement parmi les activités évaluées. » « Le second domaine est celui de la formation par la recherche qui, dans le référentiel à quatre critères, n’était qu’un élément parmi d’autres du critère ‘gouvernance et vie de l’entité de recherche’ », poursuit l’agence. « Pour donner à la formation toute sa place dans les missions assignées aux entités de recherche, il a paru nécessaire, là encore, de traiter celle-ci comme un critère à part entière. »
CHAMP D’APPLICATION, FAITS OBSERVABLES ET INDICES DE QUALITÉ
L’Aeres souligne aussi que ce nouveau référentiel « répond plus nettement aux attendus de la loi de programme pour la recherche de 2006 », qui définit la « mission d’intérêt national » des personnels de la recherche comme comprenant : « le développement des connaissances ; leur transfert et leur application dans les entreprises, et dans tous les domaines contribuant au progrès de la société ; la diffusion de l’information et de la culture scientifique et technique dans toute la population, et parmi les jeunes ; la participation à la formation initiale et continue ; l’administration de la recherche ; l’expertise scientifique. »
Chacun des six critères est lui-même décomposé en trois aspects : son « champ d’application », les « faits observables » et les « indices de qualité ». Le « champ d’application » d’un critère « résume les aspects que l’évaluateur doit apprécier, en des termes généraux pour tous les types d’entités de recherche et pour tous les domaines ». Par exemple, le critère « production et qualité scientifiques », tel qu’il est défini dans le référentiel, « apprécie, par rapport aux standards de la discipline et du domaine de recherche, les découvertes, les résultats, les problématiques, les faits expérimentaux conduisant à des réalisations scientifiques ». Le document précise qu’ « il apprécie également l’originalité, la qualité et la portée de la recherche ». Les « faits observables » désignent pour leur part « des données factuelles qui peuvent être observées par les évaluateurs pour étayer leur appréciation ». Quant aux « indices de qualité », il s’agit de « la valeur à accorder à ces données pour élaborer l’appréciation proprement dite ».
UN DOCUMENT COMPLÉMENTAIRE SUR LA NOTATION VA ÊTRE PRÉPARÉ
Le « référentiel » de l’Aeres rappelle que ces critères « s’appliquent non seulement aux entités de recherche », mais aussi à leurs « composantes » : les équipes internes et les thèmes. En conséquence, « chaque critère est donc noté non seulement au niveau de l’entité de recherche, mais aussi, lorsque celle-ci est structurée en équipes internes, au niveau de ces équipes ». En revanche, les thèmes ne sont pas notés, « dans la mesure où ils ont en général un caractère transversal et où ils réunissent des personnels appartenant à plusieurs équipes internes d’une même entité de recherche ». L’agence indique qu’il reste à « lier étroitement la définition des critères et l’échelle de notation », autrement dit qu’ « à chaque note doit correspondre un niveau de qualité, dont la définition doit être précisée ». « Ces éléments feront l’objet d’un document méthodologique complémentaire » qui sera rendu public « avant le lancement de la campagne d’évaluation 2013-2014 », annonce-t-elle.
L’évolution des critères d’évaluation « a intégré les propositions recueillies auprès des responsables d’entités de recherche évaluées et de leurs évaluateurs », précise l’Aeres. « Elle a également pris en considération les réflexions méthodologiques conduites par plusieurs groupes de travail (…) et par d’autres agences européennes », tout en ayant « tiré parti des recommandations de dernier rapport de l’Opecst (Office parlementaire d’évaluation des choix scientifiques et technologies) » (AEF n°161145). L’agence précise qu’ « avant d’être rendu public, ce document a été soumis à la direction d’un grand nombre d’organismes de recherche et à la CPU (Conférence des présidents d’université) ». En outre, ajoute-t-elle, « à l’issue de chaque campagne d’évaluation, l’agence procédera à un retour d’expérience qui permettra aux évalués d’apporter les contributions qu’ils jugeront utiles à l’amélioration de la réflexion en cours ».
ÉVALUER L’INTERDISCIPLINARITÉ ET LES SHS
Le document comporte aussi les résultats d’un « travail sur la méthodologie d’évaluation de l’interdisciplinarité et sur les critères adéquats pour apprécier équitablement ce type de recherche », selon que l’on parle de « pluridisciplinarité » (« juxtaposition de perspectives disciplinaires »), d’ « interdisciplinarité » (« coopération de plusieurs disciplines autour de projets communs ») ou de « transdisciplinarité » (« approche scientifique qui dépasse les points de vue disciplinaires par l’approche globale d’une question »). Pour l’Aeres, si « les critères d’évaluation des entités de recherche pluri, inter ou transdisciplinaires ne sont pas différents de ceux auxquels on recourt pour évaluer des entités monodisciplinaires », il s’avère néanmoins « nécessaire de dégager les faits observables propres aux différentes formes d’interaction des disciplines et les indices de qualité correspondants ». Un « groupe de réflexion » travaille à ces questions, mais d’ores et déjà, l’agence considère que « deux marqueurs » peuvent nourrir l’évaluation de l’interdisciplinarité : « le type d’interaction et la proximité entre les disciplines qui interagissent ».
Enfin, le document de l’Aeres présente « une note complémentaire sur la production et la qualité scientifiques en sciences humaines et sociales ». Il ne s’agit pas d’ « un autre référentiel », avertit l’agence, dans la mesure où les six critères élaborés offrent déjà « un référentiel commun qui intègre les perspectives des sciences humaines et sociales, au même titre que les autres, et peut, le cas échéant, s’y adapter ». Il s’agit plutôt de « quelques précisions aux faits observables et aux indices de qualité » relatifs au seul critère de « production scientifique », « pour intégrer au mieux la variété des formes de publication et des autres productions scientifiques en sciences humaines et sociales, mais aussi la diversité relative des langues qui sont utilisées pour la recherche dans ce domaine ». L’Aeres propose ainsi des repères pour « la caractérisation des revues » et « la caractérisation des ouvrages scientifiques », accompagnés des « principes de révision des listes de revues en sciences humaines et sociales » et d’une définition de ce que l’agence considère comme relevant d’un « véritable travail d’édition scientifique » des actes de colloques et des ouvrages collectifs.
Face aux arguments commerciaux développés en faveur de tels ou tels séquenceurs de paillasse à haut débit, l’étude menée par Loman et al., Nature Biotechnology propose une comparaison des performances du 454 GS Junior (Roche), du MiSeq (Illumina) et du PGM Ion torrent (Life Technologies), à partir du séquençage de novo d’un isolat d’E.coli O104 :H4 responsable de l’intoxication alimentaire en Allemagne (Mai 2011). Pour cela, le génome de référence a été généré à l’aide du GS FLX (Roche) et permettra d’évaluer l’efficacité des séquenceurs et algorithmes d’assemblages associés.
Sur la base des caractéristiques techniques établies en 2011, les trois plateformes ont permis de générer un draft exploitable du génome bactérien sans pour autant parvenir à le reconstituer à 100%. Par conséquent, il est impossible de désigner un séquenceur comme étant celui de choix, chaque plateforme étant un compromis entre avantages et inconvénients.
Deux « runs » de 454 GS Junior (associés au bénéfice de reads de 600 bases) ont permis d’atteindre 96,28% de couverture par rapport au génome de référence (Contre 96,05% pour le MiSeq et 95,4% pour le PGM Ion torrent).
Malgré une capacité de séquençage du PGM Ion torrent supérieure à celle du 454 GS Junior (100Mb contre 70Mb) et une précision de séquençage voisine (Q20, soit 1 erreur toutes les 100 bases), la technologie reposant sur les semiconducteurs fait face à ses limites au niveau de la gestion des homopolymères et accessoirement d’une longueur de reads de 100 bases.
Vingt fois supérieur au 454 GS Junior en terme de quantité de bases décodées, un « run » de MiSeq ne parvient qu’à talonner les performances d’un double « run » de la technologie Roche. Bien qu’étant la seule à gérer les homopolymères et la seule doter d’une précision à Q30 (soit 1 erreur toutes les 1000 bases), la technologie Illumina faillit sur ses tailles de « reads » qui ne permettent pas une gestion optimum des séquences répétées lors de l’assemblage.
L’approche analytique est également évoquée et selon le système d’assemblage utilisé (Velvet, CLC, MIRA), de nombreuses différences sont observées.
Enfin, ce document est l’occasion de rassembler des notions assez pragmatiques comme le tarif des plateformes (et annexes associées), la durée d’un « run » de séquençage ou encore la quantité de données générées.
Ceci étant, on est en droit d’émettre quelques réticences quant à la pertinence de cette étude qui base son comparatif sur une application (séquençage de novo) qui n’est pas la vocation première de cette catégorie de séquenceurs. Par ailleurs, dans le laps de temps où cette étude a été réalisée et la parution des résultats, la technologique du PGM Ion torrent a évolué. Testé sur les bases d’une puce 316 (100Mb) et d’une technologie de 100 bases en longueur de « reads », le PGM bénéficie désormais de la puce 318 (1 Gb) associée à une technologie de reads de 200 bases. Sans tarder, Life technologies réagit (“Loman et al reflects the past, not the present”) et propose une mise à jour au travers de ce document.
Voici un lien sur le site Molecular Biology Freeware for Windows… un lien destiné aux biologistes un peu allergiques à Linux et qui ne souhaitent pas débourser des milliers d’euros dans des logiciels qui ne peuvent s’avérer utiles que sporadiquement (pour certains).
Le site Molecular Biology Freeware for Windows est particulièrement laid mais il permet de référencer ces logiciels utiles pour bon nombre d’applications : phylogénie, alignement de séquences, etc.
L’alliance un peu paradoxale du logiciel gratuit, fiable et utile sous Windows !
Ce site-référence est pour quelques uns d’entre vous… : il classe les logiciels outils de biologie moléculaire en fonction des catégories suivantes :
A. DNA, RNA and genomic analysis
B. Plasmid graphic packages
C. Primer design
D. Protein analysis
E. Viewing three dimensional structures
F. Alignments
G. Phylogeny
H. Miscellaneous
I. Graphic packages
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages