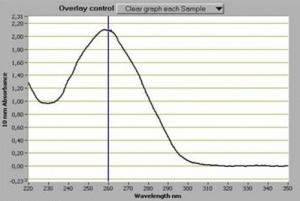
Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
En matière de dosage des acides nucléiques, il apparaît fondamental (si vous utilisez un spectrophotomètre) de connaître la fameuse loi de Beer où A=Ɛ.l.C
A étant l’absorbance (sans unité) et Ɛ est le coefficient d’absorption moléculaire (unité : L.mol -1 .cm-1)
l : la longueur du trajet optique (valeur fixe, donnée par l’épaisseur d’une cuve ou d’un ménisque sur un Nanodrop) (unité : cm)
C, souvent ce que l’on souhaite déterminer: la concentration d’acides nucléiques (unité : mol.L-1)
Cette formule permet d’estimer assez aisément une concentration en acides nucléiques, elle est vérifiée lorsque la solution est de concentration inférieure à : c < 0,1 mol.L-1
| A260 nm | ||
| acides nucléiques | Ɛ (M-1.cm-1) | pour 1 unité absorbance (µg.mL-1) |
| ADN db | 6200 | 50 |
| ADN ss | 8350 | 37 |
| ARN | 7700 | 40 |
| standard : température ambiante, l=1cm, A260/A280 =1,80 | ||
Assez rapidement, la concentration, le plus souvent en ng.µL-1, est déduite. Le point fort de la méthode spectrophotométrique (comparée à la méthode fluorométrique, plus sensible, quant à elle) est fournie par les informations que l’on tire des ratios suivants:
Ratio A260/A280 : un ratio de 1,80 (+/- 0,1) permet de qualifier une extraction d’ADN comme pure, ce ratio se situe autour de 2,0 pour une extraction d’ARN (+/- 0,1). Si ce ratio est inférieur cela signifie que l’extraction est polluée par des protéines et/ou des composés phénoliques
Ratio A260/A230 : la valeur attendue de ce ratio se situe entre 2,0 et 2,20. Si ce ratio est plus faible cela indique nécessairement qu’un composé absorbe à 230 nm. Ces composés peuvent le plus souvent être de l’EDTA, des sucres, et encore une fois le phénol et particulièrement le TRIzol reagent.
Ces valeurs de ratios peuvent parfois être négligées, cependant elles donnent une indication quant à la présence d’éventuels inhibiteurs qui viendraient perturber le devenir de vos acides nucléiques (PCR et autres réactions enzymatiques). Il est important de réaliser une valeur de blanc sur la solution qui vous aura servi à éluer vos acides nucléiques (car comme il a été précédemment mentionné l’EDTA absorbe à 230 nm… et bien souvent le TE 1X sert à reprendre et conserver des acides nucléiques)
vous trouverez ci-contre le mode d’emploi du Nanodrop 1000.
Une autre méthode alternative, permettant une meilleure sensibilité : méthode picogreen. Cette méthode, bien que très sensible, donne des résultats non valides (car très largement sous-estimés) dès lors que votre solution d’acides nucléiques est polluée par des traces d’agents détergents (type SDS). En effet, ces « adjuvants » quenchent la fluorescence émise par l’agent qui s’intercale au sein des acides nucléiques que l’on souhaite doser.
Note : bien souvent il vous est utile de calculer la quantité de molécules (quantité de copies).
Sachant que la masse molaire moyenne d’une base azotée => Masse Molaire (MM) : 309 g.mol-1
&
avec le nombre d’Avogadro 6,02214129.1023 molécules.mol-1
La formule devient : nombre de copies pour 1 µL = [C (concentration mesurée en ng/µL) x V (volume, ici 1 µL) x 10-9 ]/ [(MM environ 309 g.mol-1) x la taille de votre génome en pb x 2] le tout que l’on multiplie par le nombre d’Avogadro… le tour est joué !
plus simplement : 978 Mb pèsent 1 pg (en avant le produit en croix).
 Plusieurs de nos articles évoquent la technologie d’Oxford Nanopore, entre impatience et curiosité.
Plusieurs de nos articles évoquent la technologie d’Oxford Nanopore, entre impatience et curiosité.
Le principe de cette technologie : séquencer sans pré-amplification (il s’agit d’un séquenceur de 3ème génération) une molécule d’ADN en en hydrolysant les nucléotides élémentaires, qui passeront dans des pores. Ces pores seront couplés à des capteurs saisissant la signature singulière de chaque base constitutive de la molécule à séquencer. Jusqu’à présent le problème majeur sur lequel ont buté les ingénieurs d’Oxford Nanopore : accélérer la célérité d’acquisition des capteurs ou ralentir le passage des nucléotides au travers des pores -nucléotides qui, au départ, s’engouffraient à plus de 1000 nucléotides par seconde ce qui provoquait des problèmes d’acquisitions des signaux et donc des erreurs, sachant que l’objectif étant de ne pas dépasser les 400 bases / seconde, seuil au-delà duquel le capture devient fou…
Le 3 mai 2012, Oxford Nanopore annonce une collecte de fonds de 31,4 millions de livres (environ 50,8 millions de dollars). Encore plus de fond pour un séquenceur qui deviendrait pour un peu, un gouffre financier… « Ce tour de table a permis une levée de fonds dont la quasi-totalité provient d’investisseurs existants, ces fonds permettront un développement de l’entreprise, comprenant le développement de notre infrastructure, un renforcement de nos forces de vente, une optimisation de la fabrication et la poursuite de la R & D » a déclaré le Dr Gordon Sanghera, chef de la direction d’Oxford Nanopore. Oxford Nanopore se préparerait donc (enfin) à vendre ses produits qui sont attendus pour la fin d’année 2012. Phénomène plutôt rassurant pour la société anglaise, elle bénéficie de la confiance renouvelée de ces investisseurs, plus informés que nous sur ce que renferment (vraiment) ses tiroirs.
Eviter l’écueil d’un PacBio, trop cher, commettant trop d’erreurs, arrivé pour occuper le marché balbutiant du séquençage troisième génération, semble être la marque d’Oxford Nanopore -une société qui prend son temps. Après la possibilité de séquencer des molécules d’ADN à l’état natif, de taille très longue, en un temps record, il semble qu’Oxford Nanopore, avec ce nouvel appel de fonds, ficelle sa stratégie commerciale pour un lancement en grande pompe de leurs 2 références : le GridION & le MinION, la clé USB qui séquence dont vous pouvez voir la photographie ci-contre
Voici un documentaire diffusé sur Arte qui aborde en un peu plus de 50 minutes la biologie synthétique. Laetita Ohnona, la réalisatrice du film, pondère les interventions des Craig Venter et autres ingénieurs du MIT, technoscientistes par plusieurs interventions d’un lanceur d’alerte, Jim Thomas, filmé devant une campagne rassurante qui contraste avec l’univers d’un laboratoire. Ce documentaire très pédagogique (à mes yeux, il s’agit de sa principale qualité), consacre les ingénieurs face aux chercheurs -de la recherche appliquée vers la recherche fondamentale, en quelque sorte. Ainsi des parallèles sont réalisés entre des Steve Jobs & Bill Gates, deux personnages ayant révolutionné les applications liées à l’informatique et des Randy Rettberg & Tom Knight (co-fondateurs du prix Igem), des ingénieurs-chercheurs ayant œuvré en informatique pour terminer leur carrière dans les biotechnologies.
L’expérience de Philippe Marlière est développée en fin de documentaire pour apporter un début de solution au contrôle des organismes synthétiques comme outils de production.
Ce documentaire avivera la curiosité des scientifiques en herbe attiré par l’image d’Épinal de biogeek construisant un biofermenteur dans le garage de papa, effraiera les sceptiques, et très certainement produira un peu ces deux effets sur les initiés. On pourra simplement regretter l’absence d’un regard bioéthique dans ce documentaire qui permettrait de prendre un certain recul, évitant les allers-retours entre fascination technologique et peur de fin du monde, un monde ravagé par des bactéries tueuses luminescentes…
Ci-dessous : le synopsis d’Arte concernant ce documentaire
Les spécialistes sont unanimes : la biologie connaît une révolution aussi décisive que la découverte de l’ADN ou le séquençage complet du génome humain. La biologie de synthèse doit permettre à moyen terme à l’homme de se faire démiurge, pour écrire des séquences partielles ou entières d’ADN – et donc créer ainsi artificiellement la vie. Une perspective tellement nouvelle qu’elle suscite des vocations en dehors des laboratoires eux-mêmes. Dans de simples garages ou des hangars, les « biohackers » sont aujourd’hui capables d’assembler des séquences d’ADN synthétiques (via des DNA cassettes ou biobricks disponibles sur Internet). Peut-être donneront-ils vie à de nouvelles cellules, voire à des organismes entiers. Certains experts posent la question sans détour : l’homme va-t-il, pour la première fois, prendre la main sur l’évolution naturelle des espèces ? Et si oui, quelles seront les conséquences pour les écosystèmes les plus fragiles, et l’équilibre des espèces vivantes ? De nombreux acteurs, dont les plus gros laboratoires, investissent en tout cas dans la biologie de synthèse, jusqu’à Bill Gates, qui finance un programme colossal contre la malaria reposant sur elle. C’est cette révolution en marche que ce film souhaite montrer pour la première fois, en partant à la rencontre de ceux qui la portent.
(France, 2012, 52mn)
ARTE
Date de première diffusion : Jeudi 26 avr. 2012, 22h25
Date(s) de rediffusion : Samedi 5 mai 2012, 10h45
Vidéo disponible sur : http://videos.arte.tv/fr/videos/fabriquer_le_vivant-6609804.html
Vous aurez noté que le tour de France de Galaxy ne visite qu’un axe Perpignan-Lyon-Paris -loin de faire le tour de notre hexagone- on peut se réjouir de ce type d’initiatives. Galaxy en tournée un évènement dont vous trouverez quelques détails dans la suite de ce petit article…
 Lors de la visite de trois villes françaises des ateliers seront tenus à Perpignan, Lyon et Paris. L’inscription est gratuite, mais l’espace est limité et l’inscription pour chaque atelier sera arrêtée dès que les salles accueillant la tournée de Galaxy seront bondées.
Lors de la visite de trois villes françaises des ateliers seront tenus à Perpignan, Lyon et Paris. L’inscription est gratuite, mais l’espace est limité et l’inscription pour chaque atelier sera arrêtée dès que les salles accueillant la tournée de Galaxy seront bondées.
Le programme est disponible en suivant le lien ci-dessous :
http://wiki.g2.bx.psu.edu/Events/GalaxyTourDeFrance2012
28 mai: Perpignan
30 Mai: Lyon
1 juin: Institut Curie Amphithéatre Constant Burg, 12 rue Lhomond, 75005 Paris, France
Galaxy pour les biologistes :
9:00-12:00
Concepts de base: interface, historique, workflow
L’analyse des données NGS : découverte de polymorphismes, ChIP-seq. ARN-seq
En utilisant votre propre instance sur Cloud
Publication de « résultats supplémentaires » à l’aide des pages de Galaxy
Galaxy pour les développeurs :
14:00-17:00
Galaxy l’architecture
Outils pour le système Galaxy, Galaxy « entrepôt d’outils »
API
Exécution des sites de production Galaxy: infrastructure, planification de « jobs », de données et de gestion des utilisateurs
Cette tournée est à l’image de Galaxy : satisfaisante pour l’utilisateur biologiste qui se sert de cette « plateforme multimodale » comme outil d’analyse au bio-informaticien et informaticien qui intègre ses propres outils à Galaxy, cet outil chéri de Gaël tient, par la communauté des personnes qui l’anime toutes ses promesses.
Voici l’arrivée et la mise en exploitation du premier Ion Proton (Life Technologie) au Baylor College of Medicine à Houston.
36 heures entre la réception et la génération des premières données (avec des préparations de librairies réalisées en amont de la réception de la machine…).
Une présentation assez sympathique et ludique de la machine de Jonathan Rothberg… en avant première.
Le séquençage massif parallélisé palliant les limites des anciennes méthodes, a depuis ces dernières années amorcé de nouvelles perspectives, comme l’étude d’altérations génomiques pour la compréhension de nombreuses maladies rares (Plan National Maladies Rares). De plus, la mise sur le marché des derniers séquenceurs de paillasse aux visées hospitalières (positionnement de marché de Ion torrent avec son PGM) contribue également au développement de nombreux diagnostics cliniques.
Face à cette révolution, il convient de faire face au goulot d’étranglement de l’analyse et de l’interprétation, en faisant appel à une stratégie plus appropriée que le séquençage de génome complet et qui consiste à focaliser les efforts de décodage sur les régions codantes enrichies (Exome, gènes cibles, etc…) par une étape préliminaire de d’enrichissement d’exons. Tout en contribuant à une diminution des coûts (on ne séquence que ce qui semble pertinent…), elle permet entre autre d’augmenter la profondeur de séquençage des régions d’intérêts, paramètre indispensable pour l’identification d’altérations génomiques.
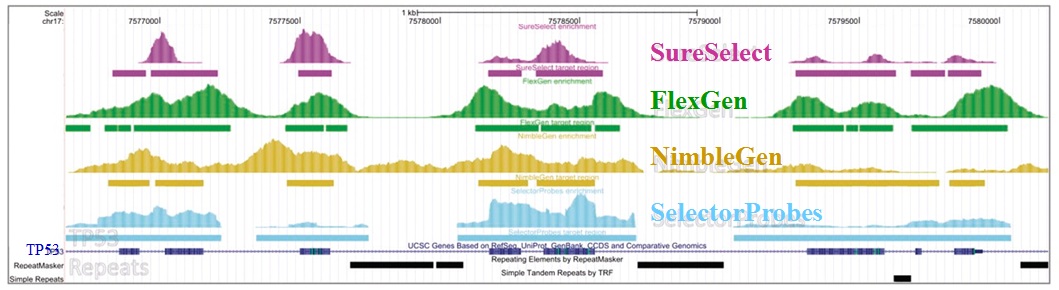
Succinctement, les techniques d’enrichissement sont multiples et peuvent être regroupées en trois catégories sur la base de leur principe technique:
– Enrichissement par capture d’hybrides : La capture par hybridation des régions cibles est initiée à partir de la librairie de séquençage. Elle peut être effectuée soit en solution (billes magnétiques) soit sur support solide (microarrays). Récemment, de nombreuses solutions (commerciales ou « à façon ») ont été développées: SureSelect, Agilent – SeqCap EZ, Roche NimbleGen – Sequence Capture Arrays, Roche Nimblegen – FleXelect, FlexGen – MYselect, MYcroarray – TargetSeq, Life Technologies .
– Enrichissement par circularisation de fragments d’ADN : Cette méthode est supérieure à la précédente en terme de spécificité. En amont de la préparation de la librairie, les fragments d’ADNg (fragmentation enzymatique ou mécanique selon la méthode) sont enrichis via une sonde constituée d’une séquence universelle flanquée aux extrémités de séquences spécifiques de la région cible. HaloPlex, Agilent.
– Enrichissement par PCR : Cette approche intervient avant la préparation de la librairie et consiste grossièrement en une PCR multipléxée ciblant les régions d’intérêts. Une étape préliminaire de design des amorces est requise. SequalPrep, Life technologies. Tout juste disponible, Ion AmpliSeq Designer, Life Technologies propose un PCR Ultra-Plex (jusqu’à 1536 amplicons) avec un étape de digestion des amorces permettant de ne conserver que les régions d’intérêts lors du séquençage.
Une technologie récente et élégante faisant appel à la microfluidique et l’émulsion PCR permet l’amplification multipléxée (jusqu’à 20000) en micro-gouttelettes (une paire d’amorces par microréacteur) en un seul tube. Multiplicom, Fluidigm – RainDance.
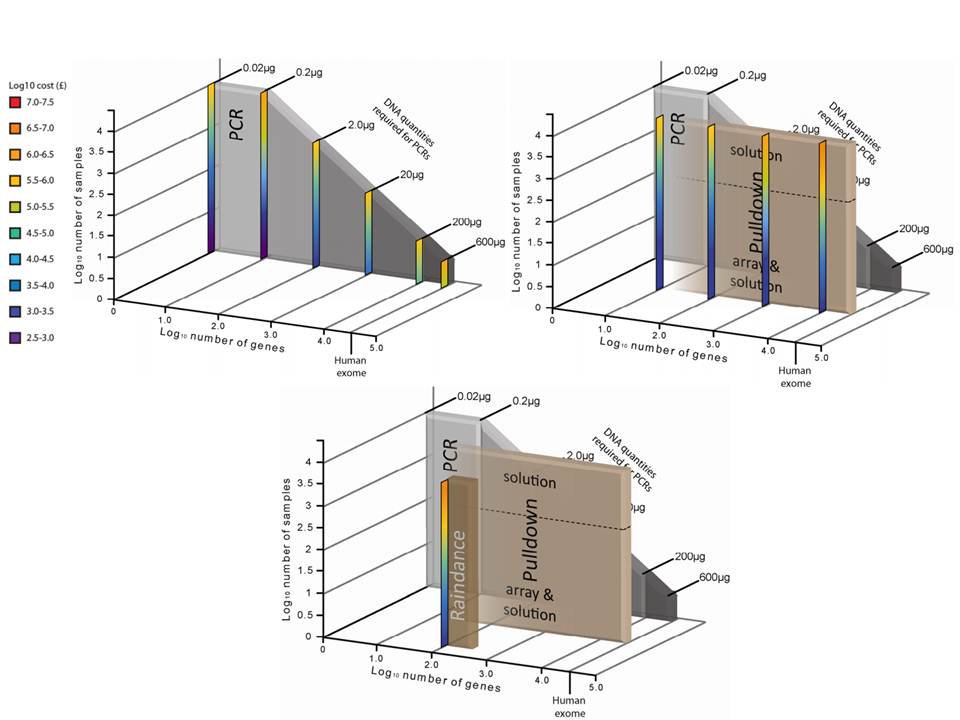
Ceci étant, les considérations fondamentales dans l’analyse de variants résident notamment dans:
– la profondeur de séquençage
– l’homogénéité de couverture des régions d’intérêt
– la reproductibilité de la méthode
– la quantité d’ADN « Input » requis
– le nombre d’échantillons à traiter
– le coût global
L’étude récemment menée par Florian Mertes et al. (nov. 2011) souligne l’importance de ces notions et fait le postulat de performances de captures de régions cibles significativement différentes selon la méthode d’enrichissement employée.
Il est impossible d’identifier une méthode d’enrichissement comme étant la meilleure notamment parce que ces approches sont en continuelle évolution et amélioration. Chacune d’elles répondent à des caractéristiques particulières et à des applications distinctes.
Si l’enrichissement par circularisation offre davantage de spécificité mais moins d’uniformité, l’inverse est aussi valable pour l’enrichissement par capture d’hybrides. Parmi les considérations dans le choix d’une méthode, le nombre d’échantillons et la taille des régions cibles sont essentiels. Si la capture d’hybrides est privilégiée pour l’analyse de mégabases (étude de l’exome) avec un nombre limité de cas, cette méthode sera délaissée au profit d’une approche PCR multipléxée pour l’étude d’un nombre restreint de régions cibles de petite taille appliquée à un grand nombre d’échantillons (Diagnostic).
L’association des méthodes d’enrichissement au séquençage haut débit offre des capacités technologiques ouvrant la voie vers de nombreuses perspectives. Toutefois, si l’émergence des séquenceurs de troisième génération s’accompagne à nouveau d’une diminution des coûts de séquençage, on est en droit de se demander si cette option restera aussi attractive.
Le réseau régional d’ingénieurs en bioinformatique de Lille et le PPF bioinformatique organisent lundi 14 Mai 2012 une conférence sur l’utilisation des outils GMOD (Generic Model Organism Database). Cette conférence se déroulera de 13h30 à 17h30 dans l’amphithéâtre de l’Institut de Biologie de Lille.
« Le projet GMOD a pour objectif de fournir une série d’outils génériques, clé en main, pour gérer et visualiser différents types de données biologiques. »
Deux interventions sont prévues :
Olivier Arnaiz du CGM Centre de Génétique Moléculaire (UPR 3404 Gif-sur-Yvette) introduira GMOD tout en se focalisant plus particulierement sur deux outils : Chado et Gbrowse. Chado est un schéma de base de données relationnelles qui permet d’intégrer des données « omiques » (génomiques, transcriptomices, séquençages…), tandis que GBrowse est un outil permettant de visualiser des annotations sur un génome.
Joelle Amselem de l’INRA – URGI (BIOGER Versailles) présentera un outil d’annotation développé à l’URGI à partir de modules GMOD. Deux exemples concrets d’utilisation seront développés.
Pour en savoir plus, vous pouvez télécharger le programme ici.
Si vous souhaitez des informations sur GMOD, je vous invite à consulter la présentation de GMOD.
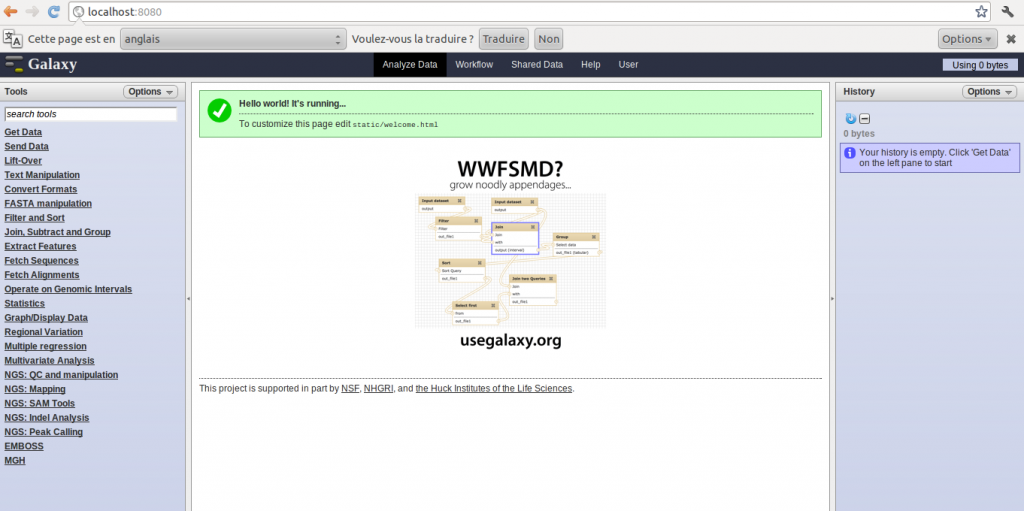
Cet article a pour but d’aider à la mise en place d’un serveur Galaxy en local. Il décrira également les avantages et inconvénients d’une telle approche par rapport à d’autres utilisations (Galaxy en ligne et Galaxy sur cloud).
Pour une définition de ce qu’est le logiciel de workflow Galaxy vous pouvez lire l’article précédemment posté sur ce blog ou accéder à la description PLUME du logiciel.
Préambule :
Il existe trois méthodes pour utiliser Galaxy :
> En local : c’est la méthode que nous décrirons ci-dessous
> En ligne : outre l’instance principale de Galaxy, de multiples organismes ouvrent l’utilisation de leurs serveurs Galaxy à l’extérieur ( pour une liste exhaustive cliquer ici)
> Sur un cloud : On utilise notre propre instance (virtuelle) de Galaxy mais au lieu d’être déployés sur une machine locale, les « jobs » sont envoyés sur le cloud de Amazon
Chaque approche a ses avantages et ses inconvénients, nous les détaillerons plus bas.
Installation :
Un grand nombre d’indications sont extraites du tutorial d’installation de Galaxy en local, disponible, en anglais, à cette adresse – http://wiki.g2.bx.psu.edu/Admin/Get%20Galaxy.
Ce qu’il faut retenir de l’installation de Galaxy en local est qu’il est possible de mettre en place un serveur en moins de 10 minutes!
Brève Explication du processus d’installation (sous un serveur Debian – les étapes seront les même pour Ubuntu):
Ouvrir une invite de commande
– Vérifier sa version de python :
python --version
La version 2.5 (et au-delà) est suffisante pour un bon fonctionnement de Galaxy.
– Installer le système de gestion de version mercurial : la version 1.6 est suffisante. S’ il est déjà installé, vérifier votre version :
hg --version
– Télécharger l’archive dans un répertoire (par exemple « workflow ») approprié :
hg clone http://bitbucket.org/galaxy/galaxy-dist
– Lancer Galaxy! Dans le répertoire galaxy-dist :
sh run.sh --reload
Note : reload permet de fermer correctement l’application sous debian (et ubuntu)
A ce stade, le programme devrait initialiser l’application.
Vous pouvez maintenant accéder à votre serveur Galaxy à l’adresse suivante : http://localhost:8080
Evidemment ce tutoriel est court et ne prend pas en compte la configuration plus fine de l’application. Pour plus de détails sur l’installation mais aussi sur l’utilisation je vous invite à lire (et à tester) les excellentes explications consultables sur le site de Galaxy :
http://wiki.g2.bx.psu.edu/Admin/Get%20Galaxy
http://wiki.g2.bx.psu.edu/Admin/Training/
Pour les (heureux) propriétaires d’un serveur de calcul :
http://wiki.g2.bx.psu.edu/Admin/Config/Performance/Cluster
Pour l’utilisation de Galaxy dans un environnement de production – ou si vous voulez directement installer votre serveur Galaxy en partant sur des bases solides :
http://wiki.g2.bx.psu.edu/Admin/Config/Performance/Production%20Server
Utilisation :
Il faut savoir que Galaxy en local fait souvent un lien vers les logiciels sans que ces derniers ne soient présents « physiquement » sur votre serveur. Ainsi le mapping avec BWA ne marchera pas, à moins que vous installiez au préalable bwa sur le serveur. C’est le cas de pas mal d’outils, donc si vous rencontrez parfois des erreurs lors de l’exécution, il est fort probable qu’il vous faudra d’abord installer les outils utilisés par Galaxy.
Un des inconvénients majeurs de la version en ligne concerne la limitation de chargement de jeux de données à 2 Gb. En séquencage haut-débit cette limite est très souvent atteinte. En ligne il est possible de contourner cette limitation en chargeant ses fichiers par FTP. Mais on ne peut éviter le problème du débit (temps de transfert long) et de la confidentialité des données.
En local, cette la limitation est également fixée à 2 Gb, et il y a également la possibilité de charger ces jeux de données directement dans le logiciel, sans passer par une phase de chargement (« upload ») en installant son propre FTP. Évidement en local, le débit est fortement accéléré.
Pour ce faire, vous pouvez lire le tutoriel suivant en anglais ou suivre les indications ci-dessous :
http://wiki.g2.bx.psu.edu/Admin/Config/Upload%20via%20FTP
ou regarder la video tutoriel .
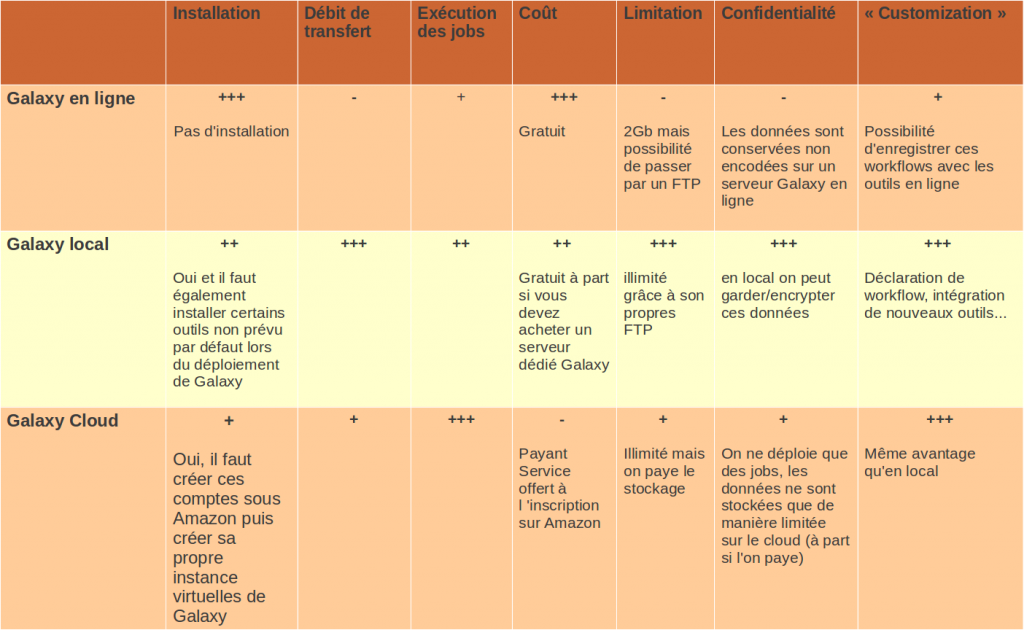
Comparaison entre les méthodes :
Pour résumer, l’installation en local offre plusieurs avantages par rapport à la version en ligne, en terme de confidentialité, de taille de données, de transfert et de temps d’analyses. Quelques inconvénients subsistent notamment la contrainte d’investir dans une machine correcte si l’on veut obtenir un temps d’analyse correct ou la nécessité d’installer certains outils extérieurement à Galaxy.
Si l’on supprime l’aspect coût et les contraintes lors de la première installation, la solution Cloud semble la plus pertinente si l’on a pas accès à des environnements matériels conséquents.
L’aspect « customisation » et particulièrement l’intégration de ses propres outils fera l’objet d’un prochain article.
 A quelques jours du premier tour, cet article vous propose d’aborder les propositions d’orientations de la recherche scientifique des 5 courants majeurs participant aux présidentielles de 2012.
A quelques jours du premier tour, cet article vous propose d’aborder les propositions d’orientations de la recherche scientifique des 5 courants majeurs participant aux présidentielles de 2012.
Nous allons procéder en partant de la gauche à la droite frontistes en passant par des nuances de rose, orange et bleue : du « pour en finir avec les Bidulex » de J.L.Mélenchon, à la préférence de la langue française dans les communications scientifiques prônée par M. Le Pen.
– Front de Gauche – le candidat J.L. Mélenchon par l’intermédiaire du site Enseignement Supérieur & Recherche – appels Front de Gauche, remet la notion de service public au centre de la politique de la recherche française. Le Front de Gauche dresse un triste bilan de la politique gouvernementale en mettant à mal la notion d’excellence : « Jamais le potentiel universitaire n’a été ainsi saccagé sous le prétexte mystificateur d’une “excellence” qui n’avoue pas ses véritables critères. » La multiplication des sources de financements et la structuration des organismes en poupées gigognes sont dénoncées : « Les Idex, Labex et autres « Bidulex » visent aujourd’hui à créer des territoires d’exception au sein de la République pour mieux conforter la concurrence libre et non faussée internationale. » L’ex-ministre de l’Enseignement professionnel taille un costard à l’ANR qu’il souhaite « dissoudre » (pour le remplacer par quoi ?). J.L. Mélenchon propose de transformer les IDEX qui lui paraissent participer d’une concurrence entre les intervenants de la recherche scientifique, en pôles de coopération scientifique. Le projet inclus le recrutement de 5000 emplois statutaires par an sur la durée du quinquennat.
– Parti Socialiste – Le programme du PS concernant la recherche scientifique reste quelque peu flou. Peu de mots dans les 60 engagements de François Hollande sont associés à la recherche scientifique (le programme du PS est délibérément tourné vers l’enseignement supérieur plus que vers l’aspect recherche de ce portefeuille ministériel). Malgré tout, quelques propositions peuvent être associées à la recherche scientifique : vers un plus d’autonomie et vers une recherche compétitive au niveau international- très proche sur ce sujet de la vision d’un UMP de début de mandat présidentiel de 2007 : « nous donnerons les moyens pérennes nécessaires à l’autonomie et au rayonnement des équipes, des laboratoires et des Universités ». Contrairement au parti du tribun du front de gauche, F. Hollande n’oublie pas de satisfaire les velléités scientifiques des industriels, ainsi, le projet du P.S. prévoit la création d’une Banque publique d’investissement, qui investira dans la recherche et l’innovation, soutiendra les PME-PMI, prendra des participations dans les activités stratégiques et les filières industrielles d’avenir (quelles sont elles précisément ?), et dont les moyens seront mobilisés sous la forme de fonds régionaux en co-pilotage avec les Régions. Les modalités d’obtention du crédit impôt recherche seront rendus plus simples (comment ?). Par l’intermédiaire de Vincent Peillon, le PS propose des solutions pour les étudiants et la modification de la loi LRU. Un programme du PS centré sur le financement de la recherche privée et de l’amélioration des conditions pour dispenser un enseignement supérieur de qualité, ouvert au plus grand nombre.
– MoDem- Nous arrivons ici au point de symétrie de notre tour d’horizon avec les (la?) propositions du Modem, le parti de F. Bayrou pour ce qui concerne la recherche scientifique dont voici la mesure phare : la création d’un crédit d’impôt innovation, parallèle au crédit impôt recherche… des fonds destinés à la recherche privée. Le programme de l’hyper-centre s’attarde (ceci n’est pas une critique) sur la problématique de l’enseignement mais oublie de parler de politique de la recherche scientifique… Mais en cherchant, on trouve quelques éléments : « veillons à ne pas axer nos politiques de recherche sur la base d’indicateurs d’origine asiatique qui certes favorise la diffusion des nouveaux savoirs par la publication scientifique, mais qui en pratique sont utilisés localement pour exacerber les rivalités entre équipes de recherche« … tiré du programme officiel du Modem pour l’enseignement et la recherche scientifique dont le chantre est Denis Badré, personnalité qui semble être l’interlocuteur des sujets « enseignement supérieur et recherche » du parti centriste.
– UMP – Au tour du programme du parti au pouvoir actuellement. Là aussi l’accent est mis sur l’innovation, aux talents et aux marchés, partant du principe qu’une recherche forte, dans une France forte vient surtout en aide aux PME et grandes industries pour améliorer leur compétitivité internationale, exemple :
– Le premier dépôt de brevet doit être gratuit pour les PME. Une partie des financements européens pour la recherche (Programme-cadre de recherche et de développement) seraient conditionnés à la participation de PME aux projets de recherche.
– Le parti présidentiel souhaite donc maintenir le crédit impôt-recherche. Par ailleurs, une meilleure valorisation de la recherche publique est invoquée, en favorisant les liens entre les universités et les entreprises et en intéressant les chercheurs publics aux retombées financières de leurs découvertes.
Enfin, la volonté de soutenir l’ambition (comment ?) de notre recherche et de notre industrie pour proposer de nouvelles thérapies, générer de la croissance économique au service d’une médecine humaine et contribuer au rayonnement de la médecine française…
Note : l’article que vous êtes en train de lire a été réalisé avant la parution officielle du programme de N. Sarkozy (programme qui paraitra jeudi prochain – 15 jours avant le premier tour). Vous pouvez avoir une vision du bilan du quinquennat de N. Sarkozy par le biais de la plume de Bruno Chaudret (chimiste et membre de l’Académie des sciences, élu par ses pairs, est Président du Conseil scientifique du CNRS).
– Front National – Le FN envisage de consacrer 3% du PIB d’ici 2017 à la recherche (pour information actuellement la France consacre 2,15 % de son PIB à la recherche -46% à la charge de l’Etat et 54% à la charge du secteur privé). Marine Le Pen propose de provoquer des états-généraux (un grenelle de plus?) de la recherche « pour fixer les priorités, pour définir les grands axes stratégiques, pour déterminer et mettre en œuvre les réformes structurelles permettant un rapprochement entre grandes écoles, universités et organismes de recherche. Cette réflexion de fond réunira tous les acteurs de la recherche, sachant que la situation économique de la France est telle que l’augmentation souhaitable des crédits ne pourra intervenir que très progressivement. En attendant, des choix devront être faits et des priorités fixées. » Une proposition encore un peu plus floue invoque l’augmentation du budget alloué à la recherche fondamentale dès lors que « la situation des finances publiques se rétablira ». Marine Le Pen conforte les organismes d’évaluation et d’attribution de finances de la recherche scientifique : « Les agences de financement, à l’instar de l’ANR (Agence Nationale de Recherche), verront leurs conseils scientifiques élargis et seront appelées à plus de transparence sur les conditions d’attribution des aides qui devront respecter des critères strictement scientifiques et s’inscrire en conformité avec les priorités fixées par la Nation. » Enfin, on ne peut passer à côté de l’humour sous-jacent dans la proposition suivante : « Les meilleurs chercheurs étrangers seront attirés dans le cadre d’une politique d’immigration ultra-sélective exclusivement centrée sur les talents (l’objectif global est de ramener l’immigration légale de 200 000 titres de séjour par an à 10 000 en fin de mandat)« . Pour conclure, M Le Pen souhaite favoriser l’usage de la langue française dans les colloques et publications scientifiques.
La recherche est une thématique mineure de la campagne des présidentielles de 2012. Malgré tout, ce thème fait l’objet de clivages partisans très archétypaux : la vision d’une coopération entre groupes de recherche d’un Mélenchon s’oppose à une compétition inter-groupes d’un Sarkozy. Il est étonnant d’observer que les partis les plus éloignés du centre développent largement plus ce thème. Thème de la recherche française que les PS, MoDem et UMP laissent de côté au profit de sujets plus « économiques »… pour les partis souhaitant gouverner il est certainement assez peu « rentable » en temps d’élection et de crise d’aborder un sujet aussi technique et glouton en dépenses publiques.
En pleine guerre froide des compagnies de biotechnologies, développeuses et vendeuses de matériel de séquençage… une fuite de documents est organisée. Le tout pour fournir aux personnes prescriptrices ou décideuses d’un futur achat de séquenceur, les arguments pour orienter ou justifier leur choix… parfois même leur mauvaise foi.

En cliquant sur la volute nucléique (depuis le 30/08/2012 – ce lien est censuré par Illumina- cf. commentaires de ce même article) ci-dessus vous aurez accès à un vrai-faux document confidentiel fourni par Illumina. Ce document assez bien réalisé a devancé celui de Life Technologies présenté dans notre article précédent. Il met l’accent sur le bénéfice, au niveau de la qualité des données générées, que procure un MiSeq (Illumina) par rapport à un PGM (Ion Torrent, Life Technologies)
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages