Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Tout d’abord, il semble utile de rappeler le concept d’Adam et Eve phylogénétique. Théologues rangez vos saintes écritures, il s’agit ici d’un concept scientifique et technique d’anthropogénétique. Les variations génétiques liées au chromosome Y, ainsi que celles présentes sur la mitochondrie sont utilisées pour étudier l’histoire de ces seuls fragments chromosomiques. L’Adam du chromosome Y et l’Ève mitochondriale ne sont donc que des vues de l’esprit.
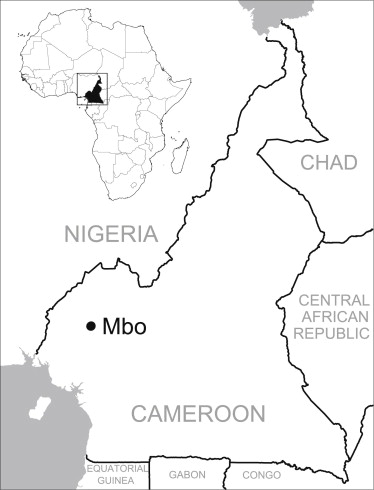
Parlons donc du cas d’Albert Perry dont le chromosome Y ne ressemblait à rien de connu. Actuellement, l’Adam du chromosome Y a vécu théoriquement entre 60.000 et 140.000 ans avant maintenant (la Eve mitochondriale quant à elle, aurait théoriquement vécu entre 140.000 et 240.000 ans avant maintenant). Surprise, après étude du chromosome Y d’Albert Perry, on s’étonne d’être passé à côté de cette lignée humaine. En exhumant des bases de données dont visiblement l’analyse n’avait pas été complète, Mendez et al. ont trouvé 11 séquences issues du chromosome Y de 11 membres de la tribu des Mbo (An African American Paternal Lineage Adds an Extremely Ancient Root to the Human Y Chromosome Phylogenetic Tree, AJHG, 2013). Cette tribu est plus ou moins localisée au Nord Ouest du Cameroun, une région d’où a été déporté un grand nombre d’esclaves. Le fait d’ajouter cette lignée humaine « oubliée », l’âge de notre Adam théorique doublerait (en accord avec la théorie de la coalescence) pour être comprise entre 237-581 000 ans.
- Carte localisant approximativement les personnes parlant le dialecte des Mbo – tirée de AJHG, Mendez et al., 2013
Le cas Perry est un exemple intéressant à plusieurs titres. En tout premier lieu, il permet de voir comment un cas particulier étudié peut avoir une incidence sur la compréhension de l’histoire humaine. En ce sens, ceci est à porter au crédit de la génomique personnelle : avec le développement des sociétés telles que 23andMe, on peut présumer que dans un avenir proche, les nouvelles données que le particulier payera afin de connaitre sa propre histoire, serviront peut être à l’ensemble de la communauté scientifique… tel que l’exemple d’Albert Perry (passé à la postérité) le suggère. Ensuite, cet exemple montre à quel point la connaissance est relative. Notre développement technologique, notre puissance de génération de données avec pour limite actuelle le temps accordé à leur analyse, laissent présager que de plus en plus vite notre connaissance sera volatile, les références changeront de plus en plus vite…
Ce petit article est aussi l’occasion de vous proposer une visite sur le très beau, très documenté blog francophone : The fossil and Mr Darwin et particulièrement de prendre le temps de lire l’article dont il est question ici : mais où est passé Adam ?
Bonne lecture !
Lancé en avril 2012, Coursera est une entreprise qui propose un accès gratuit à un ensemble de cours en ligne. Rien de nouveau par rapport aux nombreux MOOC (pour « massive open online course ») de plus en plus présents sur la toile? Dans cet article nous égrènerons les principales caractéristiques de Coursera : ses atouts, en quoi il se démarque du cours en ligne classique et pourquoi il pourrait constituer une petite révolution dans le monde éducatif .
– La plupart des MOOC sont très centrés autour de l’informatique (on peut le comprendre), des sciences mathématiques et de l’ingénierie. Coursera propose un panel de cours très variés, allant de la médecine à la poésie en passant par l’économie et l’histoire, sans oublier la biologie et la bioinformatique. On trouve même des cours de guitare en ligne!
– Coursera a conclu un partenariat avec des dizaines d’universités (dont de très prestigieuses) à travers le monde, offrant ainsi des cours de haute qualité dans différentes langues: fin 2012 Coursera annonce 680 000 inscrits provenant de 42 pays.
– Pour les instructeurs, Coursera est un catalyseur de propagation du savoir, certains cours dépassant les 100 000 étudiants, beaucoup plus que les 400 étudiants qu’un professeur peut espérer instruire chaque année dans son université. Un professeur de l’université de Stanford a ainsi calculé que son cours Coursera de 100 000 étudiants revenait à distiller des cours à une classe pendant 250 ans!
– Les cours sont fixés dans le temps, renouvelés, interactifs avec l’insertion de questionnaires au milieu des présentations, des exercices à rendre et à corriger, des forums pour partager. L’apprentissage est clairement maximisé si on se donne la peine de suivre assidument les cours. Coursera s’appuie sur des principes pédagogiques simples :
- Les questionnaires et exercices permettent d’ancrer le savoir
- Le partage et les corrections des exercices par ses pairs donnent une vision différente sur son travail et la façon dont l’étudiant a lui-même assimilé (se mettre à la place du correcteur en quelque sorte)
- L’organisation en classes: la présence d’un forum permet de partager son expérience et ses conseils avec ses pairs , ce qui est plus motivant.
– Pour les étudiants /autodidactes, c’ est une aubaine : participer à des cours réservés d’habitude à une élite et tout cela gratuitement, vive le e-learning!
Pour l’instant le plan d’affaire semble un peu flou. Pour plus de détails vous pouvez consulter la page Wikipedia dédiée à Coursera. Des certificats de participation authentifiés sont déjà délivrés (de l’ordre de 30 à 60$ par cours) pour permettre à l’e-étudiant de valoriser les compétences acquises. Coursera pourrait aussi vendre votre profil à des entreprises de recrutement (avec l’accord des étudiants). A terme des frais de scolarité pourraient être également appliqués.
Les nouveaux MOOC tels que Coursera, Udacity (MOOC plutôt centré sur les disciplines scientifiques) ou edX ( association à but non-lucratif ) sont-ils en train de révolutionner notre façon d’apprendre? Il nous faudra attendre quelques années avant de mesurer l’impact de cette propagation à grande échelle de l’apprentissage. Va-t-on assister à une véritable explosion du nombre d’autodidactes aussi compétents que de nouveaux diplômés? Ou cette manne de savoir va-t-elle rester marginale et utilisée par une élite déjà formée par le système universitaire « classique » et avide de nouvelles connaissances?
Pour finir, voici les principaux cours liés aux biotechnologies actuellement sur Coursera :
A plus long terme, vous retrouverez également un cours de l’université de Melbourne sur l’épigénétique qui commencera au 1er juillet et un cours d’introduction à la bioinformatique par l’université de San Diego d’ici la fin d’année 2013.
La liste complète des cours autour des sciences de la vie est disponible à cette adresse : https://www.coursera.org/courses?orderby=upcoming&cats=biology
Les médias se font l’écho des conséquences dues à la révolution technologique engendrée par l’évolution du séquençage haut-débit.
Qu’est ce que la biotechnologie peut faire pour vous ? Comment peut-elle repousser la date de péremption de vos artères ?
En effet, Arte (pour TF1, il va falloir attendre un épisode de Dr House traitant le sujet) diffuse un documentaire américain de moins d’une heure dont l’objectif est de présenter les évolutions technologiques dans le champ de la médecine personnalisée. Il s’agit tout d’abord de développer le concept pour le grand public (en s’appuyant sur des exemples où le mélodrame l’emporte un peu sur le débat bioéthique).
Ce document présente les acteurs majeurs de cette révolution biotechnologique qui sont tous américains (alors que, rappelons le, la plus grande plateforme de séquençage au monde est chinoise). Malgré quelques défauts, il s’agit d’un documentaire faisant tout d’abord oeuvre de pédagogie et développant des exemples plutôt précis comme autant d’arguments poussant à la large utilisation du séquençage haut-débit. Le documentaire vaut surtout pour l’accent mis sur la manière d’interpréter les résultats, sur la pertinence de ceux-ci mais tait les notions économiques liées à un futur marché qui s’annonce éléphantesque.
N’est ce pas un problème que votre génome soit, dans un avenir proche, séquencé, « interprété » et stocké par une firme privée ?
Le documentaire dont il est question est disponible ici-même (vous trouverez le synopsis dans les quelques lignes ci-dessous)
« LE DÉCRYPTAGE DU GÉNOME HUMAIN
D’ici peu, en échange de quelques centaines d’euros, chacun d’entre nous pourra obtenir un séquençage complet de son ADN. Des informations qu’il sera possible de lire, de stocker et de soumettre à l’analyse. Cette révolution est déjà en marche, ainsi que le montrent deux exemples spectaculaires : un patient cancéreux qui semble défier la mort et un malade de la mucoviscidose à présent capable de respirer normalement. Dans ces deux cas, les scientifiques sont parvenus à détecter et à neutraliser les anomalies génétiques à l’origine des maladies. Demain, grâce au décryptage du génome, il sera possible de faire un diagnostic personnalisé pour chaque individu et de mettre en place des traitements ultra personnalisés. Mais quelles sont les conséquences d’une telle révolution ? Connaître les maladies dont nous aurons à souffrir dans l’avenir, est-ce une bénédiction ou un fardeau ? Que se passerait-il si de telles informations tombaient entre les mains de compagnies d’assurance, d’employeurs ou de futurs conjoints ? Une chose est sûre : cette nouvelle ère, marquée par une médecine personnalisée s’appuyant sur nos gènes, est une réalité qui nous concerne tous. »
(Etats-Unis, 2012, 53mn)
ARTE F
Date de première diffusion : Jeu., 25 avr. 2013, 22h50
Date(s) de rediffusion : Dimanche, 5 mai 2013, 10h50
Pendant ce temps, de l’autre côté de l’Atlantique, Laurent Alexandre intervient dans l’émission grand public : ce soir (ou jamais !). Le débat avait pour thème l’obsolescence programmée. Ainsi au bout de 1h20 d’émission Frédéric Taddeï lance le débat sur l’obsolescence du corps humain. Après un extrait de l’assez mauvais film Repo Men de Miguel Sapochnik (réalisateur de certains épisodes de Dr House… c’est dire s’il doit s’y connaître en médecine personnalisée…), Laurent Alexandre, l’auteur du livre « la mort de la mort » et président de la société DNAVision, intervient ensuite. Son discours est clair et d’une grande pédagogie, il est peut être un peu dommage que l’exemple mis en avant fut le diagnostic prénatal non invasif de la trisomie 21 par séquençage haut-débit.
Evidemment très rapidement l’argument eugéniste se fait entendre. Finalement, l’intervention de Laurent Alexandre fait écho au documentaire américain.
Si les questionnements liés à la bioéthique sont au cœur du débat, rarement sont abordées les questions économiques et de confidentialité des données, associées à l’exploitation de nos données les plus intimes : la connaissance quasi-exhaustive de notre génome (et donc d’une partie de celui de nos descendants qui n’auraient rien souscrit).
Car depuis la publication de Gymrek et al., dans Science de janvier 2013 : Identifying personal genomes by surname inference, il semble difficile de soutenir l’anonymisation des données présentes dans les banques publiques. Il semble difficile d’imaginer que déposer son génome dans les banques d’une firme privée ne constitue pas un problème dont il faut d’ores et déjà prendre la mesure.
Car si tout le monde s’accorde sur les bienfaits potentiels de la médecine qui exploite la connaissance approfondie des bases nucléiques d’un patient ou d’un bien portant-futur mourant, actuellement le pouvoir est aux mains des firmes privées (Knome, 23andMe), qui se constituent des bases de données colossales. Ces bases de données hébergeant des quantités exponentielles de génomes humains (dans l’immédiat il s’agit plutôt de génotypes) permettront à leurs détenteurs de dominer un marché (voire plus !) qui s’annonce colossal…
 Société Française de Microbiologie, le Vendredi 17 Mai 2013, vous propose un colloque ayant pour thème central : les nouvelles méthodes de détection des micro -‐organismes : PCR/ESI-‐TOF MS, séquençage de nouvelle génération.
Société Française de Microbiologie, le Vendredi 17 Mai 2013, vous propose un colloque ayant pour thème central : les nouvelles méthodes de détection des micro -‐organismes : PCR/ESI-‐TOF MS, séquençage de nouvelle génération.
ASIEM, 6 Rue Albert de Lapparent, 75007 Paris (Métro Ségur, ligne 10)
L’intégralité du programme est disponible (ici, au format pdf) : les représentants des 3 fournisseurs de séquenceurs de paillasse de 2ème génération (Roche, Life et Illumina) y seront présents avec des interventions regroupées en deuxième partie de matinée. Outre le séquençage haut-débit, ce colloque est l’occasion d’entendre parler de PCRs couplées à de la spectrométrie de masse comme outil de détection moléculaire.
Des informations complémentaires sont disponibles sur le site de la SFM.
 L’accélération du débit bibliographique faisant référence aux « miRNA » atteste aisément de leur caractérisation récente (Lee RC et al., Cell (1993)) et de l’intérêt lié à leurs potentielles fonctions .
L’accélération du débit bibliographique faisant référence aux « miRNA » atteste aisément de leur caractérisation récente (Lee RC et al., Cell (1993)) et de l’intérêt lié à leurs potentielles fonctions .
Il aura fallu près de dix années supplémentaires pour mettre en évidence leur implication en tant que régulateurs biologiques (notamment au niveau de la régulation de l’expression des gènes) et leurs impacts dans de certains cancers… Aussi, le développement des nouvelles technologies de séquençage à haut débit contribue forcément à cette émergence.
Ce poste est l’occasion de présenter « miRNAtools » qui comme son nom l’indique, regroupe un grand nombre de liens renvoyant vers différents outils dédiés aux miRNA.
– Analyse de données NGS appliquées aux miRNAs (étude des profils d’expression). La liste des 7 softwares présentés n’est pas exhaustive et en voici quelques uns supplémentaires à tester: « mireap », « miRTRAP », « DSAP », « mirena », « miRNAkey », « SeqBuster », « E-mir », … . Une comparaison de l’efficacité de certains de ces outils fera l’objet d’un prochain poste.
– Prédiction de cibles selon les miRNA étudiés.
– Analyse de pathways impliquant les miR d’intérêt. Pour cette dernière application, le soft DIANA LAB – Mirpath proposé, bien que facile d’utilisation et gratuit, a le défaut de ne s’appliquer qu’aux organismes « humain » et « souris ». Dans ce registre et moyennant quelques milliers d ‘euros, « Ingenuity Pathway Analysis » (« IPA ») reste de loin l’outil idéal. En effet, en plus d’identifier les voies métaboliques au sein desquels sont impliqués les miR modulés comme proposé par Mirpath, « IPA » permet également d’intégrer les résultats de modulation de miR et d’expression de gènes pour une même condition d’étude…
 QIAGEN est un fournisseur très connu pour faire dans le haut de gamme du réactif de biologie moléculaire. Très connu pour ses colonnes de silice et sa polymérase plutôt robuste (HotStart Taq Plus). QIAGEN s’est lancé, à coups de rachats dans la qPCR et la robotique de pipettage, afin de mettre des instruments à son catalogue, instruments qui utiliseront ses consommables (on n’est jamais mieux servi que par soi-même). Ainsi la société allemande s’est payée Corbett Life Science, en 2008, 135 millions de dollars en cash. La société australienne a permis à QIAGEN d’enfin mettre à son catalogue une qPCR (à la technologie tout à fait singulière) et une robotique repeinte en bleu et gris.
QIAGEN est un fournisseur très connu pour faire dans le haut de gamme du réactif de biologie moléculaire. Très connu pour ses colonnes de silice et sa polymérase plutôt robuste (HotStart Taq Plus). QIAGEN s’est lancé, à coups de rachats dans la qPCR et la robotique de pipettage, afin de mettre des instruments à son catalogue, instruments qui utiliseront ses consommables (on n’est jamais mieux servi que par soi-même). Ainsi la société allemande s’est payée Corbett Life Science, en 2008, 135 millions de dollars en cash. La société australienne a permis à QIAGEN d’enfin mettre à son catalogue une qPCR (à la technologie tout à fait singulière) et une robotique repeinte en bleu et gris.
QIAGEN arrive sur le marché, mais encore une fois un peu tard, des séquenceurs haut débit de deuxième génération. La société de Francfort a donc racheté Intelligent Bio-Systems (IBS) dont la technologie est sous licence de Jingyue Ju (Université de Columbia). Cette licence propose une méthode permettant de diluer les molécules marquées (les terminateurs réversibles, ces mêmes molécules qui sont utilisées par la technologie d’Illumina dans Hiseq et Miseq). Ainsi les coûts des consommables s’en trouvent diminués.
Le Mini20 comme se nomme la plateforme développée par IBS, a tout d’une grande. Les coûts annoncés, il y a un an de cela, avoisinaient les 300 $ / Gbase (à la même date sur Miseq, les 600 $ / Gbase étaient dépassés). La technologie rachetée par QIAGEN permet d’utiliser « 20 flow-cells » , adressables individuellement pour un débit de 20 M reads / flow cell, débit total de 80 Gbases. Le séquenceur était annoncé à un prix de 120 k$ venant clairement marcher sur les plates bandes d’un Miseq. A n’en pas douter, l’arrivée d’un poids lourd comme QIAGEN sur le marché du séquençage haut-débit de 2ème génération devrait dynamiser et quelque peu perturber le ménage à deux de Life Technologies et Illumina. Certes, QIAGEN, avec ses solutions de préparations d’échantillons (chimie et instruments de pipettage), serait un nouvel acteur proposant des solutions complètes (et notamment en bio-informatique grâce à un partenariat avec SAP).
L’ambition de QIAGEN semble être de fournir une solution complète de l’échantillon à l’analyse des séquences que leur Mini-20 aura généré. Il faut juste espérer que les perspectives de croissance sur le marché des séquenceurs de 2ème génération ne soient pas anéanties par l’arrivée d’un séquenceur de 3ème génération à coût raisonnable. QIAGEN arrive tard dans la course mais avec une offre « tout inclus » qui serait susceptible de faire mouche pour qui n’a pas accès à un plateau technique de biologie moléculaire ou encore à des compétences en bio-informatique.
Dans la forme, cette vidéo (en anglais) se veut humoristique mais le fond est assez sérieux puisqu’il montre ce qui ne devrait pas arriver quand un scientifique effectue une demande (courtoise) de partage de données : Un enchainement d’échanges souvent à la limite de l’absurde reflet d’une situation malheureusement assez commune dans le domaine de la science.
Auteurs : Karen Hanson, Alisa Surkis and Karen Yacobucci
 A l’heure où Jonathan Rohtberg et Craig Venter cherchent de l’ADN sur Mars (lire : Searching for Alien Genomes), l’ADN sur Terre ne serait-il pas à la veille de changer de statut ? En effet, si l’ADN est « le support de l’information génétique »… est-il possible que cette macromolécule devienne le support de l’information tout court ?
A l’heure où Jonathan Rohtberg et Craig Venter cherchent de l’ADN sur Mars (lire : Searching for Alien Genomes), l’ADN sur Terre ne serait-il pas à la veille de changer de statut ? En effet, si l’ADN est « le support de l’information génétique »… est-il possible que cette macromolécule devienne le support de l’information tout court ?
– C’est humblement, dans le domaine de la traçabilité, que l’ADN a trouvé un rôle d’espion : Ainsi, au début des années 2000, la société norvégienne ChemTag, tente de développer des systèmes permettant de tracer dans nos océans, le pétrole retrouvé suite à d’éventuels dégazages. L’idée est simple : l’ADN est utilisé comme un code-barre, la succession du code de quatre lettres permet assez rapidement d’obtenir un code (4^n) ne permettant qu’avec une probabilité infime d’être retrouvé par hasard. Il faut savoir que par année, plus d’un million de tonnes de pétrole sont déversées dans les océans. La problématique en vaut la peine, Poséïdon en sera gré. Ici toute la difficulté qui se présentait à la société ChemTag : faire en sorte que l’ADN ait une grande affinité pour le pétrole (évidemment en cas de dégazage si le traceur ADN se dissout dans l’océan… adieu code-barre) et permettre que ce traceur soit purifié le plus simplement possible. L’ADN est une molécule plutôt stable et plutôt facile à « lire », ne modifiant pas les qualités organoleptiques d’une substance… et surtout cette molécule permet un nombre infini de combinaisons.
Moult industriels souhaiteraient être capables d’identifier en toute objectivité (légalement) tout organisme vivant leur appartenant (une souche de production par exemple). A cette fin, le code barre biologique peut être utilisé de deux manières :
– artificiellement, en « incorporant » par manipulation génétique une séquence connue. Cette option est bien souvent écartée parce qu’elle fait appel à la notion d’OGM, mention qui peut effrayer le consommateur.
– naturellement, en connaissant le patrimoine génétique du génome employé dans un procédé industriel. Cela implique de déterminer une séquence singulière qui n’appartiendrait qu’à cette organisme. Souvent ce type de procédures fait appel à une combinaison de séquences ou de loci polymorphes.
L’évolution des techniques de séquençages (augmentation des débits, diminutions des coûts, rapidités des runs) a permis de rendre accessible une séquence complète d’un micro-organisme ou une carte de SNPs haute-densité pour certains eucaryotes supérieurs dont on dispose de puces SNPs.
Aujourd’hui, certains envisagent d’exploiter l’ADN pour en faire un support robuste de l’information numérique.

Depuis quelques semaines, une petite bataille a lieu entre l’équipe de Georges Church de la Medical School de Boston et Christophe Dessimoz de l’EBI.
Le premier a encodé un livre dont il est le co-auteur : Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves in DNA comportant 53000 mots et 11 images en jpeg accompagné d’un programme Javascript pour un total de 5,37 Mo dans un picogramme d’ADN (un peu moins de 1 Gbase). Le second a encodé du Shakespeare, un discours de Martin Luther King, une photo et la copie d’un article de 1953 décrivant la structure de l’ADN… on s’amuse plutôt pas mal aussi de ce côté de l’Atlantique !
Ces querelles d’encodeurs ont malgré tout un intérêt. Ils démontrent la faisabilité de ce type de système et montrent qu’il est désormais envisageable de sauvegarder pour des échelles de temps très longues, de grandes quantités d’informations qui échapperaient à bien des autodafés.
Les systèmes d’encodage au sein même de l’ADN relégueront nos séquenceurs haut-débit au niveau de lecteurs DVD ou Blu Ray du futur… La limite du système, actuellement, tient plus des synthétiseurs d’ADN et des technologies de séquençage qui n’ont pas réellement été conçus à cette fin. Mais à l’heure où l’on promet d’ici quelques dizaines de mois un séquençage de génome humain pour une centaine de dollars contre quelques milliards il y a 10 ans, tous les espoirs sont permis pour que l’information séculaire contenue dans une bibliothèque patrimoine de l’Humanité devienne une bibliothèque d’Alexandrie du futur, franchissant les âges, échappant aux catastrophes naturelles, aux censures et à l’oubli.
 Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Quelques médias d’outre-Manche évoquent cette fameuse technologie de séquençage en l’affublant du qualificatif de « future grande invention britannique » : lire à ce sujet la page web de « The Raconter » : Britain greatest inventions ». Cette invention est associée à la seule médecine personnalisée comme pour envisager le futur marché du séquençage haut-débit.
Les voyants semblent donc au vert pour Oxford Nanopore. Pour préparer le terrain, sort en ce début d’année, un article de Nature Methods : « disruptive nanopores« . Un titre qui fait écho à celui de Forbes (février 2012) repris dans notre image ci-dessus.
Nicole Rusk, rédactrice en chef à Nature Methods, vient avancer les principales caractéristiques du futur produit :
– des reads entre 10 et 100 kb
– des taux d’erreurs entre 1 et 4 %
– la possibilité de connaître les bases méthylées
– la possibilité de séquencer directement l’ARN
– la méthode est non destructive

La technologie de séquençage de 3ème génération par nanopores promet de révolutionner plusieurs applications à commencer par le séquençage de novo en laissant peu d’espoir à la technologie de Pacific Biosciences. Elle devrait remplacer PacBio dans les stratégies de séquençage hybride (qui consiste « à coupler » un séquenceur de 2ème génération permettant d’être très profond et d’une technologie de 3ème génération permettant de générer des reads très longs ce qui permet au final un assemblage de meilleure qualité).
Après avoir engendré de la curiosité, de l’impatience, puis déçu avec une arrivée sans cesse repoussée, il semble qu’Oxford Nanopore doive prouver de l’efficacité de sa technologie. Ainsi, la société britannique a annoncé le 8 janvier 2013, une série d’accords avec plusieurs institutions telles que l’ Université de l’Illinois, l’Université Brown, l’Université de Stanford , l’Université de Boston, de Cambridge et de Southampton. Oxford Nanopore prend son temps ou rencontre des difficultés avec son exonucléase. Malgré ses dizaines de brevets, pour conserver sa crédibilité la société a dû communiquer pour convaincre de l’efficience de sa technologie en minimisant les difficultés de développements, en alimentant les tuyaux de communication avec des séquenceurs sorti de palettes graphiques loin d’être finalisés.
Ce retard de lancement s’apparente t’il à un gage de sérieux ou est-il la preuve que le séquençage par nanopores rencontre de grosses, très grosses difficultés de développement ? Réponse en 2013.
 En guise d’épilogue à nos quelques évocations de l’Agence d’Evaluation de la Recherche dans l’Enseignement Supérieur, voici une copie d‘un article du Monde du 03 janvier 2013 sobrement titré : Fin de vie pour l’Aéres.
En guise d’épilogue à nos quelques évocations de l’Agence d’Evaluation de la Recherche dans l’Enseignement Supérieur, voici une copie d‘un article du Monde du 03 janvier 2013 sobrement titré : Fin de vie pour l’Aéres.
L’article évoque l’élément du discours, où au dîner du 20 décembre 2012, à la Conférence des Présidents d’Université, Geneviève Fioraso a scellé définitivement le cercueil de l’Aéres qui agonisait depuis sa naissance. La ministre de l’Enseignement Supérieur et de la Recherche a déclaré à cette occasion : « la simplification des procédures de l’évaluation implique également des mesures législatives. Ajoutant : Je souhaite à cet égard que l’AERES actuelle soit remplacée par une agence nationale entièrement redéfinie à partir des principes d’indépendance, de simplicité de fonctionnement et de procédures ainsi que de légitimité scientifique et de transparence «
Ces propos font suite à la publication, fin septembre 2012, par l’Académie des Sciences, d’un rapport où l’AERES est présentée comme un « machin » bureaucratique et peu efficace, préconisant sa suppression.
Dans cette période de transition, il est difficile d’avoir une visibilité sur les outils qui seront mis en place pour évaluer la recherche scientifique. Il faut dire que l’idée de publier les notes des évaluations a eu l’effet prévisible d’engendrer un statu quo contre-productif pour ce qui est de l’objectif fixé : l’innovation… plus une unité est bien évaluée, mieux elle sera financée, donc mieux elle sera évaluée… Ce type d’évaluation engendre des circonvolutions plus que des innovations.
Sur le portail de l’AERES aucune ligne sur la mort annoncée de l’agence.
L’Aéres en chiffres :
Bilan :
Entre 2007 et 2011, l’Aéres a évalué 3 960 formations, 3 196 unités de recherche, 5 483 équipes de chercheurs, 325 établissements d’enseignementsupérieur et 18 organismes de recherche.
Notations :
Sur les 2 613 unités de recherche auditées entre 2008 et 2011 :
21,5 % ont obtenu la note A + ; 43,6 % la note A ; 28 % B ; et 5,8 % C.
Pour les 3 740 cursus (licences, masters et doctorats), 6 % de A + ; 44 % de A ; 41 % de B et 9 % de C.
Moyens :
5 900 experts (18 % étrangers), un budget de 17 millions d’euros, 170 salariés.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages