Malgré, le tirage d’oreilles de la FDA (la Food and Drug Administration), les consommateurs de génomique récréative ne se sont pas réellement détournés de 23andMe, société dans laquelle Google a injecté 4,0 millions d’USD. La société de Anne Wojcicki l’ex-épouse du cofondateur de Google, Sergey Brin, n’a pas vu une perte significative de clientèle et, au contraire, a continué de recruter de nouveaux clients… pour franchir le cap du 1.000.000 durant l’année 2016.

L’implication de Google dans la société s’essoufflant (un divorce prononcé entre les fondateurs respectifs des deux sociétés, en 2015), 23andMe a cherché à transformer sa base de données de profils génomiques en profits financiers directs. Ainsi, plus d’une dizaine de sociétés ont négocié un accès à ces informations (Genentech, Pfizer, etc.). Ces informations semblent aujourd’hui constituer le vrai « business model » de 23andMe.
Si cette monétisation était complètement prévisible, il faut avouer que l’impact ou plutôt le non impact de la prise de position de la FDA atténue grandement ce que l’on avait pu annoncer au sein de l’article : 23andMe au point mort.

 La toute puissante FDA (Food and Drug Administration) a calmé les ardeurs la société 23andMe en remettant en question la diffusion par la société californienne de tests génomiques personnels. (Étonnamment ?), la FDA ne semble pas réellement heurtée par le principe même d’une société de droit privé glanant des informations génomiques pour réaliser un commerce des plus lucratifs, motus concernant le devenir de ce type de données. En réalité, l’administration américaine, soucieuse du service rendu au consommateur, émet des doutes quant à la pertinence, quant à l’exploitation des résultats, après génotypages, des individus consentants, ainsi que sur la façon dont le tout est présenté au consommateur. Ainsi, 23andMe est mise en cause au niveau de la qualité de son service. Voici son seul « crime » aux yeux de la FDA!
La toute puissante FDA (Food and Drug Administration) a calmé les ardeurs la société 23andMe en remettant en question la diffusion par la société californienne de tests génomiques personnels. (Étonnamment ?), la FDA ne semble pas réellement heurtée par le principe même d’une société de droit privé glanant des informations génomiques pour réaliser un commerce des plus lucratifs, motus concernant le devenir de ce type de données. En réalité, l’administration américaine, soucieuse du service rendu au consommateur, émet des doutes quant à la pertinence, quant à l’exploitation des résultats, après génotypages, des individus consentants, ainsi que sur la façon dont le tout est présenté au consommateur. Ainsi, 23andMe est mise en cause au niveau de la qualité de son service. Voici son seul « crime » aux yeux de la FDA!
L’administration américaine lui reproche :
– malgré les sollicitations récurrentes de la part de la FDA, 23andMe n’a pas fait valider par l’administration ses dispositifs diagnostiques in vitro. Elle n’a cessé d’allonger la liste (stratégie commerciale du restaurant chinois) des maladies et caractères que leur système était susceptible de diagnostiquer -avant « sa mise en demeure », 23andMe proposait un menu comportant le « diagnostic » de 254 prédispositions à des « maladies » ou aptitudes diverses.
– au sujet des maladies multi-factorielles, telles que le cancer de l’ovaire ou du sein, les résultats rendus aux clients sont très probablement constitués d’un grand nombre de faux positifs et de faux négatifs.
la FDA souhaite que l’intégralité des assertions de 23andMe soit validée pour que la société continue de vendre son kit phare : health-related genetic tests.
La société qui comptait régner en maître sur le marché de la génomique récréative, a peut-être fini de jouer les apprentis sorciers (franchement peu probable !). En effet, si l’intérêt de certains de ces tests génomiques, en soi, n’est pas à remettre en question, leur encadrement législatif est à souhaiter. En outre, avant de valider des kits « diagnostiques », il serait appréciable de lever le quiproquo : les kits proposés par 23andMe sont ils des dispositifs de génomique récréative ou des dispositifs médicaux ? La libéralisation incontrôlée de ce type de marchés pourrait avoir des conséquences dramatiques… Si des prédispositions peuvent être inscrites dans notre code génétique, il n’en demeure pas moins qu’une large majorité de maladies peut être qualifiée de multifactorielle.
Se sentir à l’abri d’une maladie parce que 23andMe vous rend un résultat dans ce sens ou dans le cas contraire, sentir l’ombre de l’épée de Damoclès, peut avoir, pour le consommateur de ces tests, des conséquences graves.
La lettre d’avertissement du 22 novembre 2013, de la FDA adressée à Anne Wojciki, montre l’étendue de l’ambiguïté : LETTRE d’AVERTISSEMENT ainsi que la réponse de 23andMe.
Anne Wojcicki a déclaré que « 23andMe travaille activement avec la FDA pour s’assurer que la firme fournit des informations de haute qualité afin que les consommateurs puissent avoir confiance ».
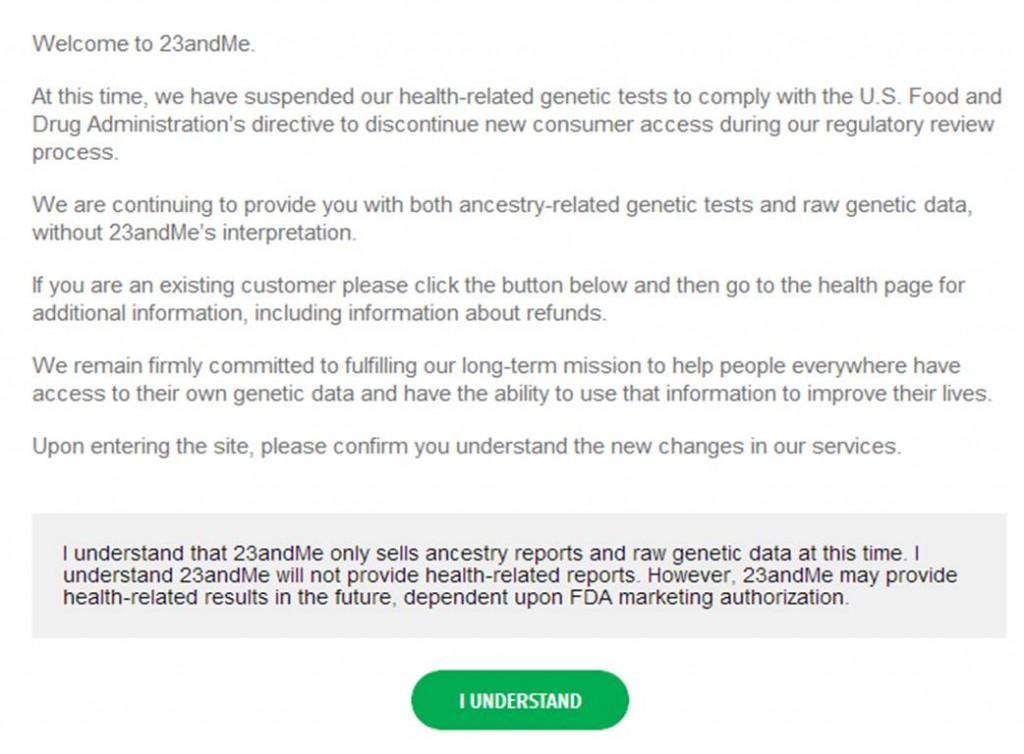
Ci-dessous, voici la nouvelle page qui vous accueille sur le site de 23andMe qui peut continuer à vendre ses tests phylogénétiques.
Comme le disait Stephen Hawking :
« J’ai remarqué que même les gens qui affirment que tout est prédestiné et que nous ne pouvons rien y changer regardent avant de traverser la rue. » (Trous noirs et bébés univers, Éditions Odile Jacob, 1994)
Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !
 Un slogan, une baseline : « nous avons cartographié le monde, maintenant cartographions la santé humaine » annonce la volonté de Google de persévérer dans le domaine de la santé humaine. C’est ainsi qu’Alphabet le conglomérat appartenant à et détenant Google tout à la fois, propose de constituer une cohorte humaine phénotypiquement caractérisée le plus finement possible : Verily (anciennement Google Life Sciences) une filiale d’Alphabet spécialisée dans la santé, a annoncé mercredi 19 avril qu’elle souhaitait recruter 10 000 volontaires pour son projet Baseline, annoncé en 2014 et déjà testé sur une centaine de volontaires. S’adjoignant des chercheurs du monde académique avec la participation de l’université de Duke (Caroline du Nord) et de l’université Stanford, Google vise à collecter des données de santé très précises sur ces personnes pendant plusieurs années. Assurément le nombre de personnes visées est pour l’instant moindre que la célèbre cohorte Nurses’ Health Study débutée en 1976 et dénombrant 280.000 participants, l’innovation consiste en la qualité des données collectées par l’intermédiaire de capteurs connectés. Les études épidémiologiques faisant intervenir des cohortes ne sont certes pas nouvelles, elles sont un outil formidable, grandes pourvoyeuses de résultats scientifiques valorisables (la célèbre cohorte de Framingham enregistre plusieurs centaines de publications). Dans le cas précis du projet Baseline, ce qui est nouveau est la promotion de ce type d’approche par et pour aussi un peu, une entité privée. Google, enfin Alphabet, peu importe finalement, est un conglomérat qui possède notamment la société de biotechnologie Calico, et qui a des liens capitalistiques avec 23andMe et possède donc encore Verily dont certains projets consistent en :
Un slogan, une baseline : « nous avons cartographié le monde, maintenant cartographions la santé humaine » annonce la volonté de Google de persévérer dans le domaine de la santé humaine. C’est ainsi qu’Alphabet le conglomérat appartenant à et détenant Google tout à la fois, propose de constituer une cohorte humaine phénotypiquement caractérisée le plus finement possible : Verily (anciennement Google Life Sciences) une filiale d’Alphabet spécialisée dans la santé, a annoncé mercredi 19 avril qu’elle souhaitait recruter 10 000 volontaires pour son projet Baseline, annoncé en 2014 et déjà testé sur une centaine de volontaires. S’adjoignant des chercheurs du monde académique avec la participation de l’université de Duke (Caroline du Nord) et de l’université Stanford, Google vise à collecter des données de santé très précises sur ces personnes pendant plusieurs années. Assurément le nombre de personnes visées est pour l’instant moindre que la célèbre cohorte Nurses’ Health Study débutée en 1976 et dénombrant 280.000 participants, l’innovation consiste en la qualité des données collectées par l’intermédiaire de capteurs connectés. Les études épidémiologiques faisant intervenir des cohortes ne sont certes pas nouvelles, elles sont un outil formidable, grandes pourvoyeuses de résultats scientifiques valorisables (la célèbre cohorte de Framingham enregistre plusieurs centaines de publications). Dans le cas précis du projet Baseline, ce qui est nouveau est la promotion de ce type d’approche par et pour aussi un peu, une entité privée. Google, enfin Alphabet, peu importe finalement, est un conglomérat qui possède notamment la société de biotechnologie Calico, et qui a des liens capitalistiques avec 23andMe et possède donc encore Verily dont certains projets consistent en :
- des lentilles de contact permettant de contrôler le niveau de glucose chez les personnes diabétiques
- des cuillères pour les personnes ayant des tremblements, par exemple atteintes de la maladie de Parkinson (projet Liftware)
- une plateforme permettant la détection de maladie par l’intermédiaire de nanoparticules
- un bracelet connecté permettant de suivre des paramètres liés à la santé
Certains de ces objets connectés auront une exposition majeure du fait de leur positionnement central dans la cohorte de Baseline. Ainsi une montre, au design contestable, complétera la panoplie de capteurs associée au projet. Le fameux capteur d’activités nocturnes qui se faufile jusque sous la couette peut laisser perplexe… Ces objets connectés, pourvoyeurs de données, de beaucoup de données, nécessiteront les méthodologies que Google s’attache à développer pour traiter de façon automatisée et optimale la manne visée par la cohorte Baseline ! En outre, ces objets trouveront un écho, à n’en pas douter, auprès de futurs consommateurs bien au-delà des personnes constituant la cohorte initiale. En effet, les modèles établis sur les personnes, population référente constitutive de la cohorte (pour lesquelles sont à disposition l’intégralité des données phénotypiques) pourront être appliqués à de futures personnes dotées de la batterie de capteurs mais n’ayant pour autant pas été caractérisées finement. La cohorte servirait à établir un modèle, modèle qui serait appliqué à de futurs utilisateurs des solutions connectées proposées par Google. Ainsi des prédictions de l’état de santé et pourquoi pas la mise en place d’un système d’alerte… pouvant aller jusqu’à signifier la nécessité de consulter pourraient trouver une application commerciale.
L’objectif qu’Alphabet revendique est de cartographier la santé humaine, cet objectif passe par la création d’une immense base de données avec la santé pour finalité, permettant, grâce aux nouvelles technologies, d’«explorer la santé en profondeur». Après anonymisation comme garantie de sécurité des données, cette base de données est destinée à être transmise à des chercheurs. Pour recruter ses volontaires et les inciter à rejoindre le projet (la stratégie de recrutement peut laisser entrevoir d’ailleurs quelques biais de recrutement), Verily assure qu’il s’agit d’une manière « de participer à la création de cette carte de la santé humaine, et de laisser durablement une trace», en contribuant à son échelle à la recherche médicale. Ces futurs participants pourront partager leurs données avec leur médecin. Verily cherche des personnes nord-américaines, en bonne santé qui seront suivies et invitées à subir, pendant deux jours, chaque année, une batterie de tests médicaux. Ces bilans médicaux accompagnés des données des bio-capteurs dont ils seront dotés seront agrégés sous forme de « Big Data », matière première dont Google et ses méthodologies sont devenus experts…
Plusieurs questions peuvent être soulevées :
- est ce que Google, entreprise commerciale, finira par monétiser ces données comme matière première à une recherche scientifique ?
- est ce que la stratégie de Google ne serait pas multiple : (i) faire la promotion de ces outils d’analyses sur une matière première (dont Google serait propriétaire) tout en (ii) faisant la promotion de ces objets connectés, surfant sur un marché de la e-santé, marché en pleine expansion, tout en (iii) rentabilisant son investissement pour « partager » ses données recrutées moyennant des contreparties soumises au secret ?
Par le passé, Google Health est – était- un service Internet d’archivage de dossiers médicaux pour les internautes américains, mis en place par Google en mars 2008. L’avantage reste celui de laisser le pouvoir aux pharmaciens américains de mettre à jour automatiquement les traitements ou encore de trouver un spécialiste pour traiter des maladies adaptées. Google fermera ce service le 1er janvier 2012, faute d’un nombre insuffisant d’utilisateurs. La volonté de Google de développer une offre trouvant des applications en santé, médecine de précision, auxiliaire personnel de santé n’est donc pas nouvelle. Aujourd’hui, dans un contexte américain où les fonds publics alloués à la recherche médicale se tarissent (lire l’article de Nature sur la coupe budgétaire sans précédents de Donald Trump), le temps des partenariats public/privé est peut être venu. Dans ce contexte, même si la démarche de Google est nappée de marketing, des projets tels que Baseline pourraient être favorablement accueillis par la communauté scientifique.
 Le développement des réseaux sociaux sur internet nous a permis d’appréhender la notion du « quand c’est gratuit, c’est toi le produit » en faisant référence, à des Facebook parmi d’autres, qui monnaient les informations personnelles des adhérents à leur service à des fins de profilage marketing. L’avènement des technologies à haut-débit en génomique a vu, il y a une dizaine d’années, émerger le concept de médecine personnalisée. Depuis, ce concept a évolué vers celui de médecine de précision (certainement plus consensuel et moins égotique). A partir de 2005, des sociétés privées se sont appropriées ce concept pour le faire déborder de la sphère de la recherche ou de la clinique pour proposer l’exploitation de profils génomiques à des fins de génomique récréative (23andMe, 2006). Un retour à l’égotisme en quelque sorte.
Le développement des réseaux sociaux sur internet nous a permis d’appréhender la notion du « quand c’est gratuit, c’est toi le produit » en faisant référence, à des Facebook parmi d’autres, qui monnaient les informations personnelles des adhérents à leur service à des fins de profilage marketing. L’avènement des technologies à haut-débit en génomique a vu, il y a une dizaine d’années, émerger le concept de médecine personnalisée. Depuis, ce concept a évolué vers celui de médecine de précision (certainement plus consensuel et moins égotique). A partir de 2005, des sociétés privées se sont appropriées ce concept pour le faire déborder de la sphère de la recherche ou de la clinique pour proposer l’exploitation de profils génomiques à des fins de génomique récréative (23andMe, 2006). Un retour à l’égotisme en quelque sorte.
23andMe a d’ailleurs un business model assez osé, consistant à vendre à des clients, leurs profils génomiques (données de puce à ADN) en échange de quelques indications concernant leur « pedigree génomique » (lire: 23 chromosomes, la NSA et moi) et quelques informations hautement critiquables. Ces dernières consistaient en des probabilités de susceptibilité à certaines afflictions (ce point là a suscité l’émoi de la FDA et ce service est aujourd’hui interrompu). En clair, 23andMe subventionne sa population de référence, ses bases de données génomiques par ses propres clients. En substance ceci constitue une évolution par rapport à Facebook, si l’on était taquin on pourrait abréger ceci en un : « vous payez pour devenir un produit ». Aujourd’hui, 23andMe habille le tout d’un « On average, a customer who consents to research contributes to over 230 studies« , se faire génotyper pour le bien de la recherche… pas loin de la servitude volontaire nappée de bon sentiment. Il est temps ici de rappeler ce que disait il y a peu un des plus grands promoteurs de la génomique personnelle : « Si je devais conseiller un jeune Craig Venter, je dirais, réfléchissez bien avant que vous veniez déverser votre génome sur Internet« . De l’aveu même de Craig Venter donc, il ne faudrait pas s’emballer avant d’envisager hypothéquer 50 % du patrimoine de ses propres enfants nés ou à naître sans qu’eux-mêmes n’aient eu mots à dire.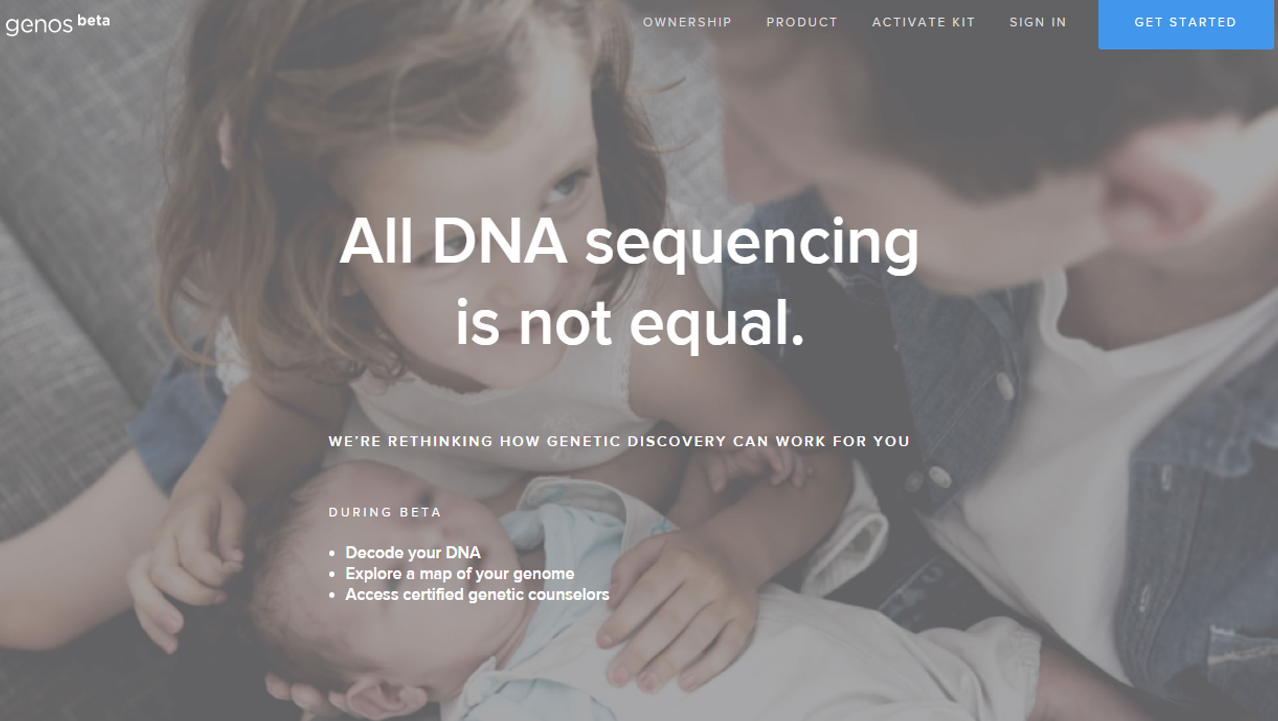
Aujourd’hui, arrive sur ce marché en pleine croissance, un nouvel acteur, Genos Research (page d’accueil ci-dessus). Ce dernier propose un nouveau business model : vous financez à raison de 399 USD votre propre séquençage d’exome (délivrant en moyenne 50 x fois plus de mutations que celles génotypées par l’intermédiaire des puces Illumina, telles que l’utilise 23andMe). Vous devenez propriétaire de vos données, à l’inverse des solutions concurrentes pour lesquelles, en général, vous en déléguez la propriété et l’utilisation future. Vous devenez donc le promoteur de votre propre information génomique que vous pouvez monnayer sur le marché de la recherche -priez pour avoir une maladie rare, ceci devrait accroître votre la cote de cette information. En outre, Genos permet aux utilisateurs de partager leurs données à travers leur plate-forme, ou d’exporter leurs données séquencées afin qu’ils puissent les transmettre à d’autres systèmes analytiques ou des conseillers génétiques –un business entier est d’ailleurs à bâtir : analyste de données personnelles génomiques. Genos est une société qui a bourgeonné à partir de Complete Genomics, une société de service de séquençage haut-débit, et a complété un financement privé soutenu par des entreprises en mai 2016. La société a  été co-fondée par Mark Blumling (l’ancien fondateur d’Hyperion Therapeutics, rachetée en mai 2015 par Horizon Pharma plc) et Clifford Reid (l’ancien fondateur et PDG de Complete Genomics, devenu une société de l’incroyable BGI’s company).
été co-fondée par Mark Blumling (l’ancien fondateur d’Hyperion Therapeutics, rachetée en mai 2015 par Horizon Pharma plc) et Clifford Reid (l’ancien fondateur et PDG de Complete Genomics, devenu une société de l’incroyable BGI’s company).
Pour le moins sensibles, que deviennent vos données génomiques au gré des acquisitions, fusions de sociétés de biotechnologie ? Ces changements de propriétaire sont monnaie courante et peuvent vous faire perdre de vue vos données qui pourraient finir par être le nouveau pétrole de l’ère post Donald Trump.
 23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
Début 2013, le produit coûte : 99 USD auxquels il faut ajouter 79,95 USD pour les frais de port (ces frais limitent l’accès au vieux continent à 23andMe), la note se monte donc pour un Français à 137,70 € (mi 2011, le prix était de 200 €). Il est facile dorénavant de comprendre que, les volumes des puces Illumina grimpant, la part du coût allouée aux consommables a baissé, mais que la distribution web a ses limites… la poste.
Tests génomiques ?
Le test génétique que propose 23andMe permet :
– d’estimer les facteurs de risques d’être, un jour, atteint d’une maladie à composante majoritairement génétique
– d’estimer vos aptitudes physiques et intellectuelles (en présupposant que la variabilité génétique de ces traits contribue fortement auxdits traits estimés)

– de déterminer son origine géographique, ainsi que, pour le folklore, de quel personnage célèbre dont 23andMe possède le génotype, vous vous rapprochez le plus (sur le ce point lire notre article : A la recherche du chromosome perdu : aux sources des hominidés)
– enfin, 23andMe accumule des génotypages venant alimenter une base de données associée à une population de référence et ainsi accroître l’information génomique disponible pour d’éventuelles recherches (à mener par d’autres…), tout cela financé par les clients de 23andMe… il fallait y penser et oser, c’est chose faite !
Principe ?
Contrairement à un amalgame souvent rencontré, la société californienne n’utilise que très peu le séquençage pour son produit phare. Le principe réside dans l’extraction de l’ADN contenu dans les cellules de la salive, ADN qui fera l’objet d’un génotypage haut-débit permettant de cartographier le génome du client. 23andMe ne fait que compiler les données de GWAS (Genome Wide Association Studies), les études d’association publiées et agglomérées plus ou moins subtilement. Ce type d’études consiste en l’association de profils génétiques à des phénotypes caractérisés. En outre, 23andMe exploite la connaissance des marqueurs en Déséquilibre de Liaison avec une maladie ou une aptitude, le tout en concaténant l’information scientifique publique.
Des témoignages de clients pour en convaincre de nouveaux
Depuis le début 2013, plusieurs témoignages ont été utilisés par la presse pour décrire ce que proposait 23andMe, une publicité à moindre frais. Celui de Slate relate l’histoire de Jill Uchiyama, une personne qui a été adoptée à la naissance et qui à l’âge de 41 ans par l’intermédiaire des services proposés par 23ansMe a retrouvé quelques cousins plus ou moins proches (300 cousins) et en a rencontré un… un manière de recréer de simili-liens de sang. Un autre présent sur le site Quora.com témoigne du cas d’un père découvrant qu’il avait des risques de développer une intolérance au gluten alors que des praticiens ont perdu un temps infini à déterminer l’étiologie de la maladie cœliaque dont souffrait sa fille. Autant de témoignages de nature à convaincre de futurs clients !
Un accueil scientifique mitigé
Un premier article de Jane Kaye « The regulation of direct to consumer genetic tests » (Human Molecular Genetics, 2008), s’inquiète de la perte de contrôle des professionnels de santé, de l’évolution de la législation et de la confidentialité des données transitant par ces nouvelles sociétés de services. L’auteure appelle de ses vœux une régulation de cette « génomique récréative » où l’on peut partager sur les réseaux sociaux son potentiel génétique, ses risques de développer une maladie ou encore rechercher des « cousins » potentiels.
Deux ans après ce premier article, celui de Barbara Prainsack et Howard Wolinsky (Future Medicine, 2010) « Direct-to-consumer genome testing: opportunities for pharmacogenomics research ? » pondère le propos. Dans cet article, les auteurs ne s’inquiètent plus vraiment de l’existence d’un système divulguant des informations génomiques directement au consommateur sans passer par la case du prescripteur médical, mais s’interrogent sur les bénéfices que la recherche scientifique pourrait tirer de ces génotypages payés par les consommateurs eux-mêmes. Entrerait-on dans l’ère du libéralisme génomique ? L’article de Future Medicine développe l’aspect crowdsourcing (en français collaborat ou externalisation ouverte) en s’inquiétant légèrement tout de même d’un problème majeur : le respect de la vie privée…
Une inquiétude majeure
A une époque où des services étatiques développent des projets tels que PRISM, l’outil de surveillance du web utilisé par la NSA ou d’autres que la DGSE utiliserait, le consommateur de la génomique personnalisée pourrait et devrait s’inquiéter de la confidentialité des données génomiques le concernant. En effet, le web est le tuyau par lequel transite toutes les informations génomiques émanant des analyses de ces sociétés de service. La confidentialité des données semble être le nouveau frein au développement de ce type de marché.
 Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Tout d’abord, il semble utile de rappeler le concept d’Adam et Eve phylogénétique. Théologues rangez vos saintes écritures, il s’agit ici d’un concept scientifique et technique d’anthropogénétique. Les variations génétiques liées au chromosome Y, ainsi que celles présentes sur la mitochondrie sont utilisées pour étudier l’histoire de ces seuls fragments chromosomiques. L’Adam du chromosome Y et l’Ève mitochondriale ne sont donc que des vues de l’esprit.
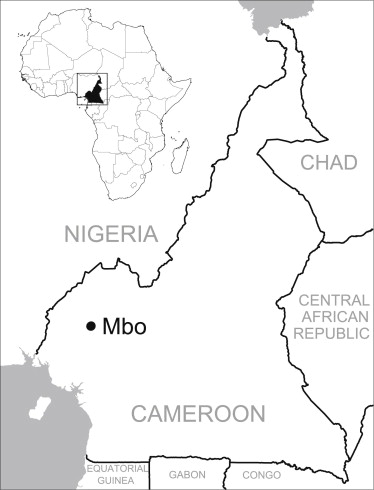
Parlons donc du cas d’Albert Perry dont le chromosome Y ne ressemblait à rien de connu. Actuellement, l’Adam du chromosome Y a vécu théoriquement entre 60.000 et 140.000 ans avant maintenant (la Eve mitochondriale quant à elle, aurait théoriquement vécu entre 140.000 et 240.000 ans avant maintenant). Surprise, après étude du chromosome Y d’Albert Perry, on s’étonne d’être passé à côté de cette lignée humaine. En exhumant des bases de données dont visiblement l’analyse n’avait pas été complète, Mendez et al. ont trouvé 11 séquences issues du chromosome Y de 11 membres de la tribu des Mbo (An African American Paternal Lineage Adds an Extremely Ancient Root to the Human Y Chromosome Phylogenetic Tree, AJHG, 2013). Cette tribu est plus ou moins localisée au Nord Ouest du Cameroun, une région d’où a été déporté un grand nombre d’esclaves. Le fait d’ajouter cette lignée humaine « oubliée », l’âge de notre Adam théorique doublerait (en accord avec la théorie de la coalescence) pour être comprise entre 237-581 000 ans.
- Carte localisant approximativement les personnes parlant le dialecte des Mbo – tirée de AJHG, Mendez et al., 2013
Le cas Perry est un exemple intéressant à plusieurs titres. En tout premier lieu, il permet de voir comment un cas particulier étudié peut avoir une incidence sur la compréhension de l’histoire humaine. En ce sens, ceci est à porter au crédit de la génomique personnelle : avec le développement des sociétés telles que 23andMe, on peut présumer que dans un avenir proche, les nouvelles données que le particulier payera afin de connaitre sa propre histoire, serviront peut être à l’ensemble de la communauté scientifique… tel que l’exemple d’Albert Perry (passé à la postérité) le suggère. Ensuite, cet exemple montre à quel point la connaissance est relative. Notre développement technologique, notre puissance de génération de données avec pour limite actuelle le temps accordé à leur analyse, laissent présager que de plus en plus vite notre connaissance sera volatile, les références changeront de plus en plus vite…
Ce petit article est aussi l’occasion de vous proposer une visite sur le très beau, très documenté blog francophone : The fossil and Mr Darwin et particulièrement de prendre le temps de lire l’article dont il est question ici : mais où est passé Adam ?
Bonne lecture !
Les médias se font l’écho des conséquences dues à la révolution technologique engendrée par l’évolution du séquençage haut-débit.
Qu’est ce que la biotechnologie peut faire pour vous ? Comment peut-elle repousser la date de péremption de vos artères ?
En effet, Arte (pour TF1, il va falloir attendre un épisode de Dr House traitant le sujet) diffuse un documentaire américain de moins d’une heure dont l’objectif est de présenter les évolutions technologiques dans le champ de la médecine personnalisée. Il s’agit tout d’abord de développer le concept pour le grand public (en s’appuyant sur des exemples où le mélodrame l’emporte un peu sur le débat bioéthique).
Ce document présente les acteurs majeurs de cette révolution biotechnologique qui sont tous américains (alors que, rappelons le, la plus grande plateforme de séquençage au monde est chinoise). Malgré quelques défauts, il s’agit d’un documentaire faisant tout d’abord oeuvre de pédagogie et développant des exemples plutôt précis comme autant d’arguments poussant à la large utilisation du séquençage haut-débit. Le documentaire vaut surtout pour l’accent mis sur la manière d’interpréter les résultats, sur la pertinence de ceux-ci mais tait les notions économiques liées à un futur marché qui s’annonce éléphantesque.
N’est ce pas un problème que votre génome soit, dans un avenir proche, séquencé, « interprété » et stocké par une firme privée ?
Le documentaire dont il est question est disponible ici-même (vous trouverez le synopsis dans les quelques lignes ci-dessous)
« LE DÉCRYPTAGE DU GÉNOME HUMAIN
D’ici peu, en échange de quelques centaines d’euros, chacun d’entre nous pourra obtenir un séquençage complet de son ADN. Des informations qu’il sera possible de lire, de stocker et de soumettre à l’analyse. Cette révolution est déjà en marche, ainsi que le montrent deux exemples spectaculaires : un patient cancéreux qui semble défier la mort et un malade de la mucoviscidose à présent capable de respirer normalement. Dans ces deux cas, les scientifiques sont parvenus à détecter et à neutraliser les anomalies génétiques à l’origine des maladies. Demain, grâce au décryptage du génome, il sera possible de faire un diagnostic personnalisé pour chaque individu et de mettre en place des traitements ultra personnalisés. Mais quelles sont les conséquences d’une telle révolution ? Connaître les maladies dont nous aurons à souffrir dans l’avenir, est-ce une bénédiction ou un fardeau ? Que se passerait-il si de telles informations tombaient entre les mains de compagnies d’assurance, d’employeurs ou de futurs conjoints ? Une chose est sûre : cette nouvelle ère, marquée par une médecine personnalisée s’appuyant sur nos gènes, est une réalité qui nous concerne tous. »
(Etats-Unis, 2012, 53mn)
ARTE F
Date de première diffusion : Jeu., 25 avr. 2013, 22h50
Date(s) de rediffusion : Dimanche, 5 mai 2013, 10h50
Pendant ce temps, de l’autre côté de l’Atlantique, Laurent Alexandre intervient dans l’émission grand public : ce soir (ou jamais !). Le débat avait pour thème l’obsolescence programmée. Ainsi au bout de 1h20 d’émission Frédéric Taddeï lance le débat sur l’obsolescence du corps humain. Après un extrait de l’assez mauvais film Repo Men de Miguel Sapochnik (réalisateur de certains épisodes de Dr House… c’est dire s’il doit s’y connaître en médecine personnalisée…), Laurent Alexandre, l’auteur du livre « la mort de la mort » et président de la société DNAVision, intervient ensuite. Son discours est clair et d’une grande pédagogie, il est peut être un peu dommage que l’exemple mis en avant fut le diagnostic prénatal non invasif de la trisomie 21 par séquençage haut-débit.
Evidemment très rapidement l’argument eugéniste se fait entendre. Finalement, l’intervention de Laurent Alexandre fait écho au documentaire américain.
Si les questionnements liés à la bioéthique sont au cœur du débat, rarement sont abordées les questions économiques et de confidentialité des données, associées à l’exploitation de nos données les plus intimes : la connaissance quasi-exhaustive de notre génome (et donc d’une partie de celui de nos descendants qui n’auraient rien souscrit).
Car depuis la publication de Gymrek et al., dans Science de janvier 2013 : Identifying personal genomes by surname inference, il semble difficile de soutenir l’anonymisation des données présentes dans les banques publiques. Il semble difficile d’imaginer que déposer son génome dans les banques d’une firme privée ne constitue pas un problème dont il faut d’ores et déjà prendre la mesure.
Car si tout le monde s’accorde sur les bienfaits potentiels de la médecine qui exploite la connaissance approfondie des bases nucléiques d’un patient ou d’un bien portant-futur mourant, actuellement le pouvoir est aux mains des firmes privées (Knome, 23andMe), qui se constituent des bases de données colossales. Ces bases de données hébergeant des quantités exponentielles de génomes humains (dans l’immédiat il s’agit plutôt de génotypes) permettront à leurs détenteurs de dominer un marché (voire plus !) qui s’annonce colossal…
 DNA-Vision est devenue en 2011, le prestataire européen en génomique avec la plus grande puissance de séquençage (quatre systèmes Solid, deux HiSeq2000, un GAIIx, tous deux d’Illumina, et deux 454-de chez Roche). La petite société issue de l’Université Libre de Bruxelles (ULB) et fondée en 2004 a bien grandi et est actuellement dirigée par Laurent Alexandre. Ce dernier est le créateur du site e-toubib, Doctissimo, aujourd’hui détenteur de 80 % de DNA-Vision, suite à la vente de son site internet à Lagardère qui a permis à l’ex-chirurgien de devenir riche… engrangeant plus de 139 millions d’euros suite à la transaction. Il faut dire que le docteur en médecine, chirurgien urologue, énarque et diplômé en sciences politiques a été à l’origine de plusieurs sociétés dans le domaine médical : Benefit, Medcost, Clinics et enfin le site préféré des hypocondriaques, Doctissimo. C’est avec pour idée principale d’appliquer le séquençage, en profitant de sa vertigineuse baisse de coûts, au champ de la médecine personnalisée que le médecin essayiste, auteur de « la mort de la mort » est devenu président de la société DNAvision. Même s’il est vrai que, l’encore assez nouveau président de DNAVision, préparant l’entrée en bourse de son entreprise, parle de médecine personnalisée, la société belge est connue pour sa maîtrise des technologies haut-débit et à titre d’exemple, pour son expertise du séquençage appliqué aux études métagénomiques (vous pouvez lire : Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa, PNAS, avril 2010).
DNA-Vision est devenue en 2011, le prestataire européen en génomique avec la plus grande puissance de séquençage (quatre systèmes Solid, deux HiSeq2000, un GAIIx, tous deux d’Illumina, et deux 454-de chez Roche). La petite société issue de l’Université Libre de Bruxelles (ULB) et fondée en 2004 a bien grandi et est actuellement dirigée par Laurent Alexandre. Ce dernier est le créateur du site e-toubib, Doctissimo, aujourd’hui détenteur de 80 % de DNA-Vision, suite à la vente de son site internet à Lagardère qui a permis à l’ex-chirurgien de devenir riche… engrangeant plus de 139 millions d’euros suite à la transaction. Il faut dire que le docteur en médecine, chirurgien urologue, énarque et diplômé en sciences politiques a été à l’origine de plusieurs sociétés dans le domaine médical : Benefit, Medcost, Clinics et enfin le site préféré des hypocondriaques, Doctissimo. C’est avec pour idée principale d’appliquer le séquençage, en profitant de sa vertigineuse baisse de coûts, au champ de la médecine personnalisée que le médecin essayiste, auteur de « la mort de la mort » est devenu président de la société DNAvision. Même s’il est vrai que, l’encore assez nouveau président de DNAVision, préparant l’entrée en bourse de son entreprise, parle de médecine personnalisée, la société belge est connue pour sa maîtrise des technologies haut-débit et à titre d’exemple, pour son expertise du séquençage appliqué aux études métagénomiques (vous pouvez lire : Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa, PNAS, avril 2010).
Revenons au moins un instant sur le séquençage haut-débit appliqué à la médecine personnalisée, vu comme le fait de rendre accessible au soignant l’information génomique de son patient.
A une époque où le Dossier Médical Unique (DMP) a grand mal à devenir une évidence, à une période où l’accès universel aux soins est de plus en plus remis en question pour des raisons de disette financière, l’accès à une archive de plusieurs Gigaoctets de données par patient semble peu crédible à court terme. Ajoutés à cela les problèmes d’éthique, de confidentialité de données et de formation du personnel soignant à l’interprétation de ces données et vous comprendrez que le rêve d’un Laurent Alexandre aura du mal à toucher la médecine de masse mais pourra séduire les cliniques privées qui échappent à la problématique d’un accès universalisé aux soins.
On voit qu’à l’instar du tourisme spatial, la cible ne semble pas vraiment être le chômeur en fin de droit. La médecine pour pas grand monde… puisque si un séquençage coûte de moins en moins cher, l’interprétation des données générées et l’intégration de ces données dans un réel parcours médical est encore inaccessible aux 99 % des personnes les plus pauvres des 10 % des pays les plus riches.
Malgré tout, la médecine personnalisée dépasse cette vision de l’ADN. En effet, la médecine personnalisée comporte plusieurs approches :
– la détermination du risque (un génotypage haut-débit chez 23andme ou un séquençage chez un autre quidam pour vous dire que vous avez 2,03 % de chance de plus que la moyenne d’être atteinte de la maladie d’Alzheimer)
– la médecine stratifiée où seuls les patients répondant positivement à l’administration d’une solution curative, minimisant les effets secondaires de celle-ci, seront traités.
– la thérapie génique (un peu passée de mode) pour laquelle il existe trois types de thérapies : la transfection permanente d’ADN, la transfection transitoire d’ADN, les thérapies portant sur l’ARN.
 Si le séquençage multiparallélisé de l’ADN a tendance à se démocratiser au sein des laboratoires de recherche et de certains laboratoires en usant comme diagnostic, il semble qu’un pas de géant reste à franchir pour que celui-ci transforme complètement notre vision de la médecine. La médecine personnalisée n’est cependant pas à remettre en question… il s’agit juste de lui donner un horizon réaliste et réalisable, sans quoi cette notion sera à ranger, comme beaucoup d’autres avant, aux rayons des mots clés usés par la mode.
Si le séquençage multiparallélisé de l’ADN a tendance à se démocratiser au sein des laboratoires de recherche et de certains laboratoires en usant comme diagnostic, il semble qu’un pas de géant reste à franchir pour que celui-ci transforme complètement notre vision de la médecine. La médecine personnalisée n’est cependant pas à remettre en question… il s’agit juste de lui donner un horizon réaliste et réalisable, sans quoi cette notion sera à ranger, comme beaucoup d’autres avant, aux rayons des mots clés usés par la mode.

Il est douloureusement fatigant de se rendre compte de l’absence de mémoire Humaine! Cette amnésie collective provoque les mêmes écueils, les mêmes cercueils. « L’Histoire est un éternel recommencement » cette sentence attribuée à Thucydide a beau être un cliché des sujets de philosophie, une partie de la Hongrie a voté pour l’extrême droite (troisième force politique du pays, troisième force politique de notre pays aussi, à en croire les résultats du premier tour de l’élection présidentielle) -cette même extrême droite hongroise se sert de test génétique humain en guise de néo-certificat d’aryanité. C’est ce que relate l’article de Charlotte Chabas du Monde (15 juin 2012) – à lire le test de « pureté génétique » qui choque la Hongrie. Des sociétés privées surfent sur la mode du test génétique, devenu désormais largement accessible pour qui veut (peut) débourser quelques centaines d’euros ou de dollars pour connaître ses origines ethniques (test de 23andme) pour le moins pire ou pour s’assurer, comme c’est le cas ici, de sa non appartenance à un groupe ethnique rejeté par une politique clairement xénophobe et antisémite. Ce certificat hongrois qui émeut une majorité de la classe politique de Budapest est exploité à des fins nauséabondes par sa franche la plus extrême. Un test dont le résultat annonce « Aucune trace génétique d’ancêtres juifs ou roms » peut faire frémir n’importe quel démocrate.
L’Histoire se répète donc, et au gré de l’avancée des connaissances et des technologies, des hommes éditent des certificats d’aryanités phylogénétiques ou encore (à l’inverse) marquent d’une étoile jaune génomique des personnes pour lesquels l’Humain n’est que le fruit de son hérédité biologique. La science raciste qui a justifié par le passé la colonisation, les pogroms, refait surface (avait elle seulement disparu) et on ne peut s’empêcher, en tant que scientifique biologiste, d’exhumer quelques tristes mots de James Watson (co-élaborateur du modèle en double hélice de l’ADN). Le scientifique alors âgé de 79 ans a déclaré dans les colonnes du Sunday Times qu’il était «fondamentalement triste au sujet du devenir de l’Afrique» parce «toute nos politiques sociales sont fondées sur le fait que leur intelligence (celles des Africains, ndlr) est identique aux nôtres (occidentaux, ndlr) alors que tous les tests ne le disent pas vraiment». La science et l’accès à la connaissance n’éloigne pas nécessairement les idées les plus abjectes, à chaque humain sa morale, à chaque scientifique son éthique… mais les partisans de la science avec conscience, celle censée élevée l’âme ne peuvent que s’indigner.
D’un 23andMe, présentant de manière positive l’accès démocratisé à la connaissance de ses origines par le biais d’études GWS (Genome Wide Scan), à un prestataire hongrois cynique s’abritant derrière des notions d’éthique pour diffuser des résultats de tests génétiques de « pureté raciale », l’accès à ce type d’informations peut être sujet à interrogations quant à leurs utilisations (personnelle ou politique). La diffusion de tels tests ouvre une boîte de Pandore; ces tests génétiques (interdits sur notre territoire national) offrent une arme aux pires propagandistes et une information sujette à interprétation et pouvant être manipulée pour le pire rarement pour le meilleur… l’Histoire est un éternel recommencement.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages