A la croisée de la biologie, de la biochimie, de l’électronique, de la mécanique des fluidiques et de la thermodynamique, de nouveaux outils se préparent. Ceux-ci viendront peut être trouver des applications dans les laboratoires de génomique mais au-delà de cette perspective, ils pourraient trouver des applications au niveau des professionnels de santé eux-mêmes avant d’être accessibles aux clients-patients-citoyens directement.
Les Lab-on-Chips (ou Laboratoire sur puce) sont un concept qui a émergé dans les années 90 et qui au sein des laboratoires de biologie moléculaire a trouvé une application largement répandue sous la forme des puces Agilent consommables pour BioAnalyzer. La technologie d’Agilent, basée sur une électrophorèse ou une cytométrie de flux microfluidique, offre un outil de contrôle de la quantité, qualité (distribution de taille, intégrité des ARNs extraits etc.) des molécules considérées. Même si les puces Agilent sont affublées du qualificatif de « Lab-on-Chip« , elles nécessitent un appareillage dédié et bien entendu un PC relié. L’outil est encore loin du modèle rêvé, c’est-à-dire complètement autonome (en une version « stand alone »).
L’émergence de ce type de laboratoires a été, en partie, permise grâce à des budgets alloués à la lutte contre le bioterrorisme. En effet, des laboratoires qui tiennent dans une poche de treillis, qui peuvent être employés sur n’importe quel théâtre d’opération, peuvent venir parfaire le paquetage des experts en sécurité intérieure. Les laboratoires Sandia (Sandia National Laboratories) font partie des principaux laboratoires nationaux du département de l’Énergie des États-Unis et sont des pionniers de ces applications avec les développements réalisés par MicroChemLab qui est actif depuis 1988 sur ce type de développements.
Aujourd’hui les laboratoires unipersonnels de diagnostic embarqués sont devenus très populaires, pour des applications liées à des analyses biochimiques. L’un des exemples les plus fameux concerne le BG-Star (Sanofi Diabète) permettant de réaliser en quelques secondes, très aisément, des lectures de glycémie. L’outil est basé sur le principe de l’électrochimie dynamique où une bandelette jetable accueille une goutte de sang qui vient activer une glucose oxydase.
Les avantages de ce type de technologies :
– Une faible consommation de volume de fluide, en raison des volumes internes faibles des puces, ce qui minimise la quantité de déchets, baisse des coûts des réactifs onéreux et surtout minimise la quantité nécessaire d’échantillon de départ
– Une rapidité d’exécution où l’efficacité des réactions biochimiques est permise par la miniaturisation de l’outil, en effet la taille du dispositif ne nécessite qu’une faible quantité d’énergie pour fonctionner
– Un meilleur contrôle du processus en raison d’une réponse rapide du système (par exemple, le contrôle thermique pour des réactions chimiques exothermiques)
– Une parallélisation massive due à la compacité, qui permet l’analyse à haut-débit
– Une réduction des coûts de fabrication, permettant une utilisation de puces jetables, un processus qui deviendra rentable s’il est fabriqué en masse
– Une plateforme plus sûre pour les produits chimiques, les études radioactives ou biologiques en raison de la grande intégration du système (souvent en système clos), des faibles quantités de réactifs et d’échantillons nécessaires
Les vertus de la miniaturisation ne nécessitent pas plus amples apologies ! Depuis quelques années l’idée d’un laboratoire qui tient au creux de la main a réellement commencé à voir le jour.
Des exemples de ces laboratoires portatifs commencent à poindre dans des versions de plus en plus intégrées.
Ainsi la technologie Mondrian développée par NuGEN, technologie basée sur de la microfluidique digitale, permet de simplifier les étapes de constitution de librairie de séquences haut-débit.
Destinagenomics propose un système analytique ne faisant pas intervenir d’enzymes. Ce système fait intervenir des sondes interrogatives de mutations que la société souhaite embarquer dans des puces : la preuve du concept a été publiée par Pernagallo et al. (Sensors (Basel). 2012).
http://youtu.be/k-NnBS2xDLo
Panasonic, le groupe japonais, numéro 4 mondial de l’électronique, en partenariat avec la société belge, Imec, propose d’embarquer dans une puce d’ 1 cm2 : une pompe permettant aux fluides de circuler, une unité d’extraction d’ADN à partir de la goutte de sang prélevée, un thermocycleur, une unité de purification de produits de PCR, et un système de détection de SNPs.

Le concept est séduisant, mais pour être viable et concurrencer les coûts de génotypages basse densité qui sont relativement bas (quelques euros / marqueur), les concepteurs des Laboratoires sur Puces misent donc sur une large diffusion de leur produit. Ainsi la production de masse impacte drastiquement le coût de production qui lui-même est impacté sur le prix vente… un prix de vente attractif et une simplicité d’utilisation qui dope les ventes… et donc fait baisser les coûts de production…
Le pari réside dans le fait d’amorcer ce cercle vertueux, en quelque sorte chaque opérateur du marché – au niveau d’une application donnée- souhaite assommer la concurrence ! Une course au développement est donc lancée.
Revenons à la science ! Vous trouverez ici le document pdf. présentant la technologie de Panasonic/Imec,
Concernant l’application de séquençage, Oxford Nanopore proposera une « clé USB » capable de séquencer, le Minion.
Dans le futur il est envisageable, d’imaginer un outil combinant la technologie de séquençage de 3ème génération, d’un Oxford Nanopore, par exemple, avec en amont un dispositif intégrant (à l’instar du projet de Panasonic), une micro-pompe, un réseau microfluidique permettant d’alimenter en échantillon et réactifs les différents éléments permettant une extraction d’ADN/ARN, la constitution d’une librairie, une purification…
Parle-t’on ici de science fiction ? Pour un début de réponse, il faut comprendre que séparément toutes ces solutions technologiques existent déjà.
Pour les plus férus de laboratoires miniaturisés, un journal scientifique est là pour eux, explicitement nommé : Lab on a Chip.
 23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
Début 2013, le produit coûte : 99 USD auxquels il faut ajouter 79,95 USD pour les frais de port (ces frais limitent l’accès au vieux continent à 23andMe), la note se monte donc pour un Français à 137,70 € (mi 2011, le prix était de 200 €). Il est facile dorénavant de comprendre que, les volumes des puces Illumina grimpant, la part du coût allouée aux consommables a baissé, mais que la distribution web a ses limites… la poste.
Tests génomiques ?
Le test génétique que propose 23andMe permet :
– d’estimer les facteurs de risques d’être, un jour, atteint d’une maladie à composante majoritairement génétique
– d’estimer vos aptitudes physiques et intellectuelles (en présupposant que la variabilité génétique de ces traits contribue fortement auxdits traits estimés)

– de déterminer son origine géographique, ainsi que, pour le folklore, de quel personnage célèbre dont 23andMe possède le génotype, vous vous rapprochez le plus (sur le ce point lire notre article : A la recherche du chromosome perdu : aux sources des hominidés)
– enfin, 23andMe accumule des génotypages venant alimenter une base de données associée à une population de référence et ainsi accroître l’information génomique disponible pour d’éventuelles recherches (à mener par d’autres…), tout cela financé par les clients de 23andMe… il fallait y penser et oser, c’est chose faite !
Principe ?
Contrairement à un amalgame souvent rencontré, la société californienne n’utilise que très peu le séquençage pour son produit phare. Le principe réside dans l’extraction de l’ADN contenu dans les cellules de la salive, ADN qui fera l’objet d’un génotypage haut-débit permettant de cartographier le génome du client. 23andMe ne fait que compiler les données de GWAS (Genome Wide Association Studies), les études d’association publiées et agglomérées plus ou moins subtilement. Ce type d’études consiste en l’association de profils génétiques à des phénotypes caractérisés. En outre, 23andMe exploite la connaissance des marqueurs en Déséquilibre de Liaison avec une maladie ou une aptitude, le tout en concaténant l’information scientifique publique.
Des témoignages de clients pour en convaincre de nouveaux
Depuis le début 2013, plusieurs témoignages ont été utilisés par la presse pour décrire ce que proposait 23andMe, une publicité à moindre frais. Celui de Slate relate l’histoire de Jill Uchiyama, une personne qui a été adoptée à la naissance et qui à l’âge de 41 ans par l’intermédiaire des services proposés par 23ansMe a retrouvé quelques cousins plus ou moins proches (300 cousins) et en a rencontré un… un manière de recréer de simili-liens de sang. Un autre présent sur le site Quora.com témoigne du cas d’un père découvrant qu’il avait des risques de développer une intolérance au gluten alors que des praticiens ont perdu un temps infini à déterminer l’étiologie de la maladie cœliaque dont souffrait sa fille. Autant de témoignages de nature à convaincre de futurs clients !
Un accueil scientifique mitigé
Un premier article de Jane Kaye « The regulation of direct to consumer genetic tests » (Human Molecular Genetics, 2008), s’inquiète de la perte de contrôle des professionnels de santé, de l’évolution de la législation et de la confidentialité des données transitant par ces nouvelles sociétés de services. L’auteure appelle de ses vœux une régulation de cette « génomique récréative » où l’on peut partager sur les réseaux sociaux son potentiel génétique, ses risques de développer une maladie ou encore rechercher des « cousins » potentiels.
Deux ans après ce premier article, celui de Barbara Prainsack et Howard Wolinsky (Future Medicine, 2010) « Direct-to-consumer genome testing: opportunities for pharmacogenomics research ? » pondère le propos. Dans cet article, les auteurs ne s’inquiètent plus vraiment de l’existence d’un système divulguant des informations génomiques directement au consommateur sans passer par la case du prescripteur médical, mais s’interrogent sur les bénéfices que la recherche scientifique pourrait tirer de ces génotypages payés par les consommateurs eux-mêmes. Entrerait-on dans l’ère du libéralisme génomique ? L’article de Future Medicine développe l’aspect crowdsourcing (en français collaborat ou externalisation ouverte) en s’inquiétant légèrement tout de même d’un problème majeur : le respect de la vie privée…
Une inquiétude majeure
A une époque où des services étatiques développent des projets tels que PRISM, l’outil de surveillance du web utilisé par la NSA ou d’autres que la DGSE utiliserait, le consommateur de la génomique personnalisée pourrait et devrait s’inquiéter de la confidentialité des données génomiques le concernant. En effet, le web est le tuyau par lequel transite toutes les informations génomiques émanant des analyses de ces sociétés de service. La confidentialité des données semble être le nouveau frein au développement de ce type de marché.
Ce poste a vocation de passer en revue les différentes étapes de la technologie de séquençage à haut-débit du PGM Ion Torrent, depuis la préparation de la librairie jusqu’à l’obtention des données brutes. Ce survol technologique permet de rassembler un maximum d’explications techniques et de termes clefs associés à cette technologie. L’intérêt est de répondre essentiellement à une attente de néophytes ou futurs utilisateurs du PGM friands de retours d’expériences.
L’arbre de décision Ion Torrent s’étant considérablement développé, seuls les axes « Whole genome » et « Amplicon » serviront de support à l’ensemble de la présentation.
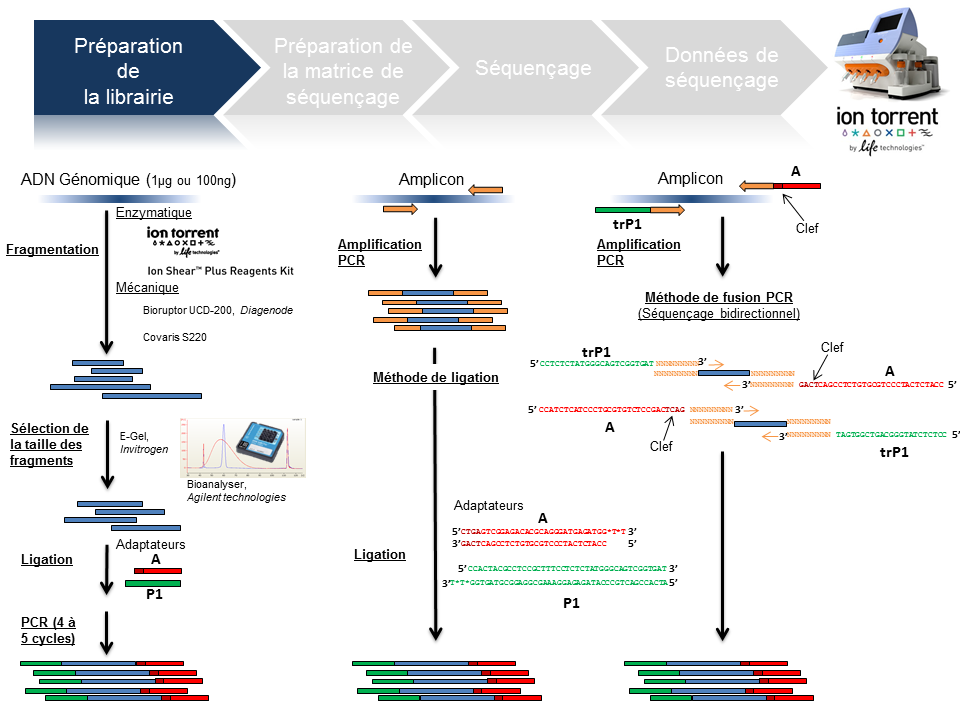
Préparation de la librairie
La finalité d’une préparation de librairie pour le PGM Ion Torrent est de lier aux fragments d’ADN à séquencer le couple d’adaptateurs A et P1. La taille médiane des fragments est variable et définie selon la chimie employée: 100, 200 , 300 ou 400 bases (Cf tableau ci-dessous).
Le traitement d’un échantillon d’ADN génomique débute par une étape de fragmentation mécanique ou enzymatique. Cette dernière présente l’avantage d’être considérablement plus rapide.
En amplicon-seq, la méthode pour flanquer les adaptateurs est double, par ligation ou par fusion PCR.
Par ailleurs, il est envisageable de traiter plusieurs échantillons en parallèle en utilisant des adaptateurs avec code barre ( En standard chez Life technologies: Au nombre de 96 pour les échantillons ADN et 16 pour les échantillons ARN).
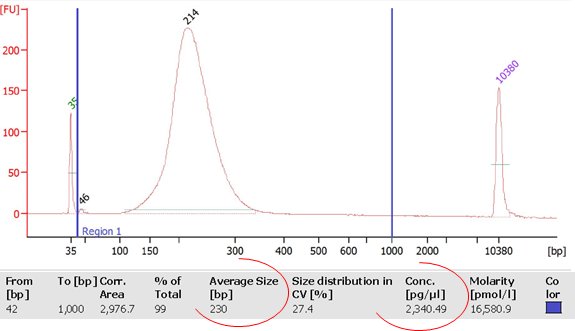
La librairie est monitorée sur puce DNA High sensitivity (Bioanalyzer, Agilent) avec comme objectifs de calculer sa concentration et d’identifier la taille moyenne des fragments qui la composent. Ces valeurs permettront ainsi de déterminer la concentration molaire de la libairie et d’y appliquer le facteur de dilution nécessaire pour favoriser le ratio idéal 1/1 (Fragment ADN de la librairie/Ion Sphere Particle) pour l’étape suivante de PCR en émulsion.
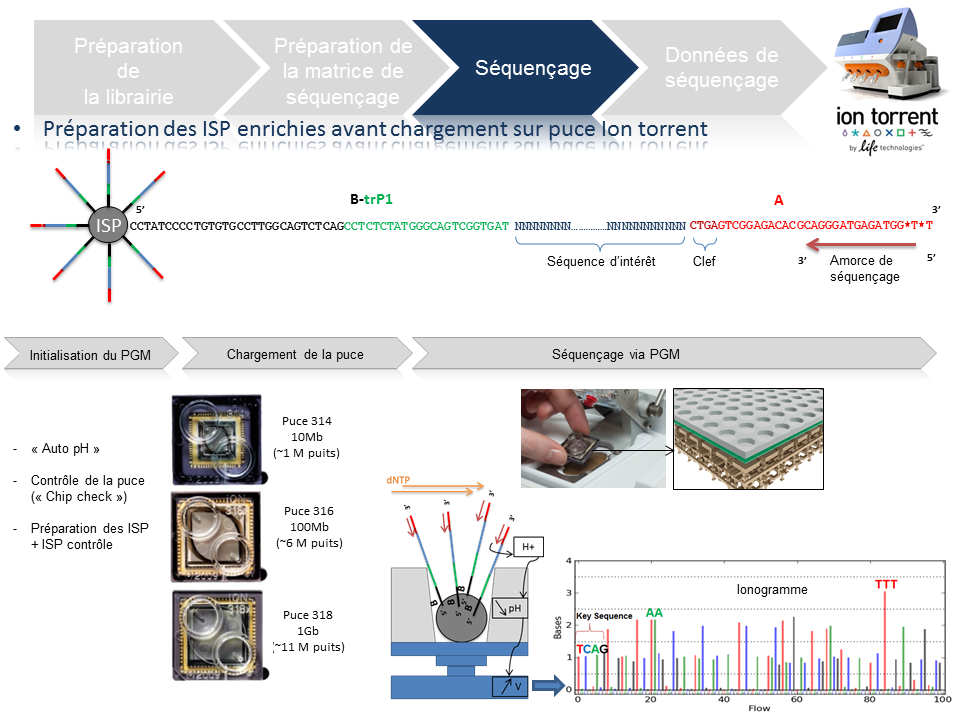
Préparation de la matrice de séquençage
Cette étape automatisée permet l’amplification clonale (OneTouch2) suivi d’un enrichissement en « Ion Sphere Particles » (ISPs) à la surface desquelles un fragment de librairie est amplifié (OneTouch ES).
L’amplification clonale est réalisée au cours de la PCR en émulsion (emPCR) et contribuera à atteindre un seuil de détection du signal nécessaire et suffisant au moment du séquençage. Malgré une optimisation du ratio 1/1 (ISP / Fragment ADN), plusieurs configurations de microréacteurs sont envisageables. Seule la configuration de monoclonalité est souhaitée car elle seule, est source de données de séquençage. Les autres configurations généreront des données qui seront filtrées lors de la primo-analyse par la « Torrent suite ».
L’amorce ePCR-A couplée à la biotine permettra l’enrichissement ultérieure par un système de capture sur billes liées à la streptavidine.
Séquençage
En amont de l’étape de séquençage, une initialisation du PGM est requise et permet notamment une homogénéisation des valeurs de pH ~ 7,8 au sein des différents réactifs de l’appareil (« Auto pH« ).
La matrice de séquençage couplée aux amorces de séquençage et à la polymérase est chargée sur la puce Ion Torrent selon un protocole bien spécifique. Les puces se déclinent selon 3 capacités de séquençage ( Chip 314 >10Mb, Chip 316 >100Mb, Chip 318 >1Gb). A noter qu’une version « v2 » pour chacune des puces précitées existe et est indispensable pour toute application de séquençage nécessitant la chimie 400. Le séquençage multiparallélisé revient donc au décryptage simultané des fragments ADN couplés aux ISP. A chaque polymérisation de nucléotides non modifiés, la libération d’ ions H+ entraine une variation de pH, elle même détectée au niveau de la couche mince (technologie des semi-conducteurs) située au fond de chaque puits. L’ensemble des données brutes générées est transcrits sous forme de ionogrammes.
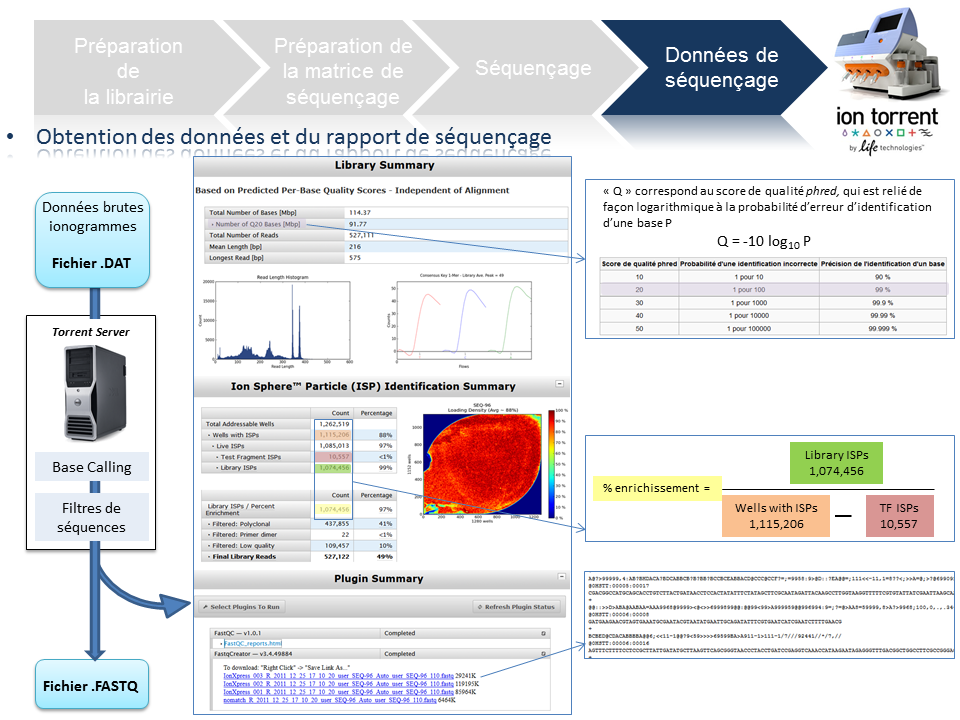
Données de séquençage
A l’issue du « run » de séquençage, le fichier .DAT regroupe l’ensemble des données brutes (ionogrammes). Ces fichiers sont transférés du PGM vers le Torrent Server. L’algorithme de « base calling » permet la conversion des données sous forme de lettres en séquences (A,T,C,G) formant le read (séquence au format fasta) associé à un score de qualité (Phred Score codé en ASCII), les deux types de données étant associés dans un fichier .FASTQ (qui tend à devenir le format de référence).
Un prétraitement est également appliqué sur la base des reads générés et qui équivaut au nombre d’ISPs vivantes ou « Live ISPs » (On parle d’ISPs vivantes pour les ISPs associées à la clef):
– trimming : élimination des adaptateurs et/ou portions de reads de mauvaise qualité
– filtres : élimination des « reads » de petites tailles, de mauvaise qualité, des polyclonaux
L’ensemble de ces informations est repris au travers du « report » généré à l’issue du séquençage et du prétraitement. Y sont également renseignés, le nombre de reads générés ainsi que leur taille moyenne.
 Ce qui suit pourrait être tiré d’un roman d’anticipation technofuturiste où le héros s’empêtre dans un polar aux implications géopolitiques. Après la mise en application de la politique de l’enfant unique, politique lancée en 1979 par Deng Xiaoping qui a eu la fâcheuse conséquence d’induire un déséquilibre entre les sexes (116 garçons pour 100 filles, 2005), la Chine, forte de sa population de presque 1 milliard 400 millions de ressortissants, cherche à appliquer les lois de la sélection génomique sur sa propre population. Le Q.I., phénotype relativement héritable (héritabilité supérieure à 0,5) est le deuxième caractère, après le sexe, « choisi » par les autorités. Cette article développe les différents éléments qui ont permis à la Chine de devenir le premier pays transhumaniste.
Ce qui suit pourrait être tiré d’un roman d’anticipation technofuturiste où le héros s’empêtre dans un polar aux implications géopolitiques. Après la mise en application de la politique de l’enfant unique, politique lancée en 1979 par Deng Xiaoping qui a eu la fâcheuse conséquence d’induire un déséquilibre entre les sexes (116 garçons pour 100 filles, 2005), la Chine, forte de sa population de presque 1 milliard 400 millions de ressortissants, cherche à appliquer les lois de la sélection génomique sur sa propre population. Le Q.I., phénotype relativement héritable (héritabilité supérieure à 0,5) est le deuxième caractère, après le sexe, « choisi » par les autorités. Cette article développe les différents éléments qui ont permis à la Chine de devenir le premier pays transhumaniste.
Acte 1 : une politique de reconquête
La Chine possède des ports en Europe, une partie significative de la dette américaine, des milliers d’hectares agricoles en Afrique et un parc de bombes atomiques qui ne laisserait pas insensible le docteur Folamour. Malgré un budget de millions de dollars alloué à une armée aux ordres du parti unique, l’usine du monde ne semble pas avoir de volonté expansionniste impérialiste en vue de repousser ses frontières (si on omet le sujet du Tibet). Il semble que les ambitions de la Chine actuelle résident moins dans le fait de mettre à la disposition de l’occident sa main-d’œuvre bon marché que de devenir la première puissance mondiale matérielle et intellectuelle. c’est pourquoi le pays mise toujours sur sa vraie matière première : l’humain. Devenue une puissance spatiale avec ses Taikonautes, la Chine envoie dans les meilleures universités du monde, ses étudiants qui, de retour (pour ceux qui reviennent), accomplissent une recherche de tout premier plan. Dans le champ de la génomique et des biotechnologies, la Chine s’impose aujourd’hui comme un acteur de tout premier plan.
Acte 2 : une recherche efficace
En effet, la Chine compte plus d’un cinquième de la population mondiale et compte aussi sur la volonté de fer pour retrouver son lustre d’antan. Il est alors, relativement aisé d’imaginer que même si une portion faible de sa population atteint les études supérieures, le pays sélectionne des étudiants qui deviendront des chercheurs motivés et ayant émergé des tous meilleurs étudiants d’une classe d’âge donné. Il est passé le temps où les meilleurs chercheurs chinois s’exilaient inconditionnellement aux Etats-Unis pour finir par être naturalisés – le classement académique des universités mondiales (aussi appelé classement de Shanghaï, ou encore « Academic Ranking of World Universities » en anglais) est un outil parfait pour identifier les meilleures universités où envoyer ses étudiants. La Chine devient même attractive sur la scène mondiale de la recherche scientifique et technologique. Selon un rapport de l’OCDE, le pays est deuxième pour la dépense en recherche et développement, injectant 136 milliards US$ en 2006 (USA : 330 milliards US$ pour la même période).
L’Empire du milieu oriente sa politique du développement technologique dans des domaines jugés comme hautement stratégiques: l’énergie, les ressources naturelles, l’environnement, les techniques de production industrielle et les technologies de l’information, les biotechnologies, les nanotechnologies, les technologies spatiales et maritimes. La Chine est en tête des pays en nombre de brevets déposés par année, ceci est l’aboutissement de trente ans durant lesquels le pays a misé sur la recherche scientifique pour rattraper son retard technologique sur l’Occident. Depuis le début des années 2000, le gouvernement promeut une innovation purement chinoise. Concernant le niveau des étudiants chinois, ceux-ci commencent à être l’objet de discriminations dans certaines universités américaines où ils occupent les meilleurs places. La recherche scientifique, basée sur une « exportation » massive d’étudiants a permis à la Chine de rattraper son retard technologique, cette technologie aujourd’hui sert une recherche scientifique de tout premier plan et sert, en ce qui concerne la biotechnologie, un dessein qui peut faire peur, vu d’ici.
Acte 3 : des moyens puissants
L’exemple qui vient à l’esprit immédiatement est le BGI (Beijing Genomics Institute). A ce sujet, vous pouvez lire notre précédent article : Taylor + Deng Xiao Ping + Watson = la Chine marche sur les chromosomes. Rappelons que le BGI est la plus grande plateforme génomique mondiale et un contributeur scientifique de tout premier ordre. Grâce à des plateformes de haute-technologie permettant de générer des volumes de séquences et de typages à de très bas coût, la Chine aurait la possibilité d’appliquer les résultats de sa recherche scientifique à l’échelle de sa vaste population.

Le BGI développe un laboratoire de génétique cognitive avec pour ambition de répondre à ces questions : Comment fonctionne le cerveau humain ? Comment les gènes affectent la capacité cognitive ? Comment les gènes et l’environnement interagissent pour produire l’intelligence et la personnalité humaine ? Le BGI a lancé un recrutement d’une cohorte de personnes volontaires aux Q.I. élevés. L’objectif est donc de rechercher les variations génétiques associées à des variations significatives au niveau du Q.I. (notons qu’une étude cas-témoin eut nécessité une cohorte de Q.I. bas à très bas… difficile de procéder à un recrutement volontaire !). Le Q.I. étant plutôt héritable, il sera donc techniquement possible de sélectionner les porteurs des variants « Q.I. élevé ». Ainsi il est possible de procéder à deux niveaux :
– orientation / optimisation « d’accouplements » : les porteurs de ces variations « Q.I. élevé », après s’être génotypés, auront tendance à rechercher un conjoint lui aussi porteur de ces mêmes variations afin de maximiser leur chance de procréer un enfant lui même porteur de ces variations (notons ici qu’il n’y a pas de réel progrès génétique, en tant que tel)
– passons par l’étape FIV : évitant de miser sur le hasard, les gamètes des deux futurs parents produiront plusieurs dizaines d’embryons, chaque œuf sera biopsié (une petite étape de phi29, un génotypage ciblé ou une sélection génomique sur la base de l’exploitation d’un génotypage pan-génomique à l’aide d’une cartographie basée sur plusieurs millions de marqueurs SNPs humains). Fort de la connaissance du « Q.I. estimé » de ces dizaines d’œufs… il ne restera qu’à « contrecarrer » l’aléa de méiose et de ne ré-implanter que ceux présentant le meilleur score de Q.I. estimé…
 La sélection humaine pose bien évidemment des problème de bioéthique. Si l’occident sélectionne d’ores et déjà des êtres humains dans le but de limiter les enfants nés porteurs de tares génétiques majeures, l’Empire du milieu semble parti sur une autre voie (même si cette voie n’empêche pas l’autre) : améliorer significativement le Q.I. de sa population et ainsi basculer dans l’ère de la transhumanisation (selon ce mouvement intellectuel, l’Homme doit prendre en main sa propre évolution).
La sélection humaine pose bien évidemment des problème de bioéthique. Si l’occident sélectionne d’ores et déjà des êtres humains dans le but de limiter les enfants nés porteurs de tares génétiques majeures, l’Empire du milieu semble parti sur une autre voie (même si cette voie n’empêche pas l’autre) : améliorer significativement le Q.I. de sa population et ainsi basculer dans l’ère de la transhumanisation (selon ce mouvement intellectuel, l’Homme doit prendre en main sa propre évolution).
Après le Q.I., le grand jeu du moment consiste à deviner quel sera le phénotype de choix (la docilité ?, le courage ?), il faut espérer que l’on attendra quelques décennies avant d’envisager le premier humain OGM… Envisageant cela, on caricature la démarche actuelle chinoise et on s’imagine, un peu, à la place de Charlton Heston à la fin de Soleil Vert (film de Richard Fleischer, sorti en 1973) quand il découvre la matière première constitutive de l’aliment qui nourrit une planète devenue stérile.
 Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Tout d’abord, il semble utile de rappeler le concept d’Adam et Eve phylogénétique. Théologues rangez vos saintes écritures, il s’agit ici d’un concept scientifique et technique d’anthropogénétique. Les variations génétiques liées au chromosome Y, ainsi que celles présentes sur la mitochondrie sont utilisées pour étudier l’histoire de ces seuls fragments chromosomiques. L’Adam du chromosome Y et l’Ève mitochondriale ne sont donc que des vues de l’esprit.
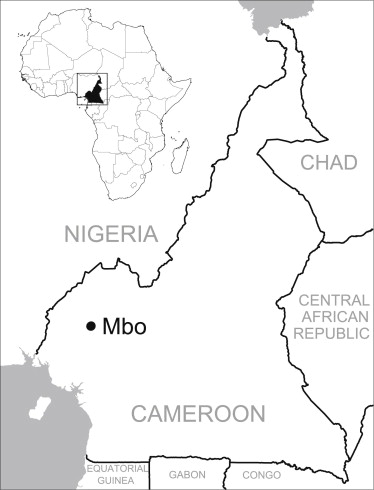
Parlons donc du cas d’Albert Perry dont le chromosome Y ne ressemblait à rien de connu. Actuellement, l’Adam du chromosome Y a vécu théoriquement entre 60.000 et 140.000 ans avant maintenant (la Eve mitochondriale quant à elle, aurait théoriquement vécu entre 140.000 et 240.000 ans avant maintenant). Surprise, après étude du chromosome Y d’Albert Perry, on s’étonne d’être passé à côté de cette lignée humaine. En exhumant des bases de données dont visiblement l’analyse n’avait pas été complète, Mendez et al. ont trouvé 11 séquences issues du chromosome Y de 11 membres de la tribu des Mbo (An African American Paternal Lineage Adds an Extremely Ancient Root to the Human Y Chromosome Phylogenetic Tree, AJHG, 2013). Cette tribu est plus ou moins localisée au Nord Ouest du Cameroun, une région d’où a été déporté un grand nombre d’esclaves. Le fait d’ajouter cette lignée humaine « oubliée », l’âge de notre Adam théorique doublerait (en accord avec la théorie de la coalescence) pour être comprise entre 237-581 000 ans.
- Carte localisant approximativement les personnes parlant le dialecte des Mbo – tirée de AJHG, Mendez et al., 2013
Le cas Perry est un exemple intéressant à plusieurs titres. En tout premier lieu, il permet de voir comment un cas particulier étudié peut avoir une incidence sur la compréhension de l’histoire humaine. En ce sens, ceci est à porter au crédit de la génomique personnelle : avec le développement des sociétés telles que 23andMe, on peut présumer que dans un avenir proche, les nouvelles données que le particulier payera afin de connaitre sa propre histoire, serviront peut être à l’ensemble de la communauté scientifique… tel que l’exemple d’Albert Perry (passé à la postérité) le suggère. Ensuite, cet exemple montre à quel point la connaissance est relative. Notre développement technologique, notre puissance de génération de données avec pour limite actuelle le temps accordé à leur analyse, laissent présager que de plus en plus vite notre connaissance sera volatile, les références changeront de plus en plus vite…
Ce petit article est aussi l’occasion de vous proposer une visite sur le très beau, très documenté blog francophone : The fossil and Mr Darwin et particulièrement de prendre le temps de lire l’article dont il est question ici : mais où est passé Adam ?
Bonne lecture !
Lancé en avril 2012, Coursera est une entreprise qui propose un accès gratuit à un ensemble de cours en ligne. Rien de nouveau par rapport aux nombreux MOOC (pour « massive open online course ») de plus en plus présents sur la toile? Dans cet article nous égrènerons les principales caractéristiques de Coursera : ses atouts, en quoi il se démarque du cours en ligne classique et pourquoi il pourrait constituer une petite révolution dans le monde éducatif .
– La plupart des MOOC sont très centrés autour de l’informatique (on peut le comprendre), des sciences mathématiques et de l’ingénierie. Coursera propose un panel de cours très variés, allant de la médecine à la poésie en passant par l’économie et l’histoire, sans oublier la biologie et la bioinformatique. On trouve même des cours de guitare en ligne!
– Coursera a conclu un partenariat avec des dizaines d’universités (dont de très prestigieuses) à travers le monde, offrant ainsi des cours de haute qualité dans différentes langues: fin 2012 Coursera annonce 680 000 inscrits provenant de 42 pays.
– Pour les instructeurs, Coursera est un catalyseur de propagation du savoir, certains cours dépassant les 100 000 étudiants, beaucoup plus que les 400 étudiants qu’un professeur peut espérer instruire chaque année dans son université. Un professeur de l’université de Stanford a ainsi calculé que son cours Coursera de 100 000 étudiants revenait à distiller des cours à une classe pendant 250 ans!
– Les cours sont fixés dans le temps, renouvelés, interactifs avec l’insertion de questionnaires au milieu des présentations, des exercices à rendre et à corriger, des forums pour partager. L’apprentissage est clairement maximisé si on se donne la peine de suivre assidument les cours. Coursera s’appuie sur des principes pédagogiques simples :
- Les questionnaires et exercices permettent d’ancrer le savoir
- Le partage et les corrections des exercices par ses pairs donnent une vision différente sur son travail et la façon dont l’étudiant a lui-même assimilé (se mettre à la place du correcteur en quelque sorte)
- L’organisation en classes: la présence d’un forum permet de partager son expérience et ses conseils avec ses pairs , ce qui est plus motivant.
– Pour les étudiants /autodidactes, c’ est une aubaine : participer à des cours réservés d’habitude à une élite et tout cela gratuitement, vive le e-learning!
Pour l’instant le plan d’affaire semble un peu flou. Pour plus de détails vous pouvez consulter la page Wikipedia dédiée à Coursera. Des certificats de participation authentifiés sont déjà délivrés (de l’ordre de 30 à 60$ par cours) pour permettre à l’e-étudiant de valoriser les compétences acquises. Coursera pourrait aussi vendre votre profil à des entreprises de recrutement (avec l’accord des étudiants). A terme des frais de scolarité pourraient être également appliqués.
Les nouveaux MOOC tels que Coursera, Udacity (MOOC plutôt centré sur les disciplines scientifiques) ou edX ( association à but non-lucratif ) sont-ils en train de révolutionner notre façon d’apprendre? Il nous faudra attendre quelques années avant de mesurer l’impact de cette propagation à grande échelle de l’apprentissage. Va-t-on assister à une véritable explosion du nombre d’autodidactes aussi compétents que de nouveaux diplômés? Ou cette manne de savoir va-t-elle rester marginale et utilisée par une élite déjà formée par le système universitaire « classique » et avide de nouvelles connaissances?
Pour finir, voici les principaux cours liés aux biotechnologies actuellement sur Coursera :
A plus long terme, vous retrouverez également un cours de l’université de Melbourne sur l’épigénétique qui commencera au 1er juillet et un cours d’introduction à la bioinformatique par l’université de San Diego d’ici la fin d’année 2013.
La liste complète des cours autour des sciences de la vie est disponible à cette adresse : https://www.coursera.org/courses?orderby=upcoming&cats=biology
Les médias se font l’écho des conséquences dues à la révolution technologique engendrée par l’évolution du séquençage haut-débit.
Qu’est ce que la biotechnologie peut faire pour vous ? Comment peut-elle repousser la date de péremption de vos artères ?
En effet, Arte (pour TF1, il va falloir attendre un épisode de Dr House traitant le sujet) diffuse un documentaire américain de moins d’une heure dont l’objectif est de présenter les évolutions technologiques dans le champ de la médecine personnalisée. Il s’agit tout d’abord de développer le concept pour le grand public (en s’appuyant sur des exemples où le mélodrame l’emporte un peu sur le débat bioéthique).
Ce document présente les acteurs majeurs de cette révolution biotechnologique qui sont tous américains (alors que, rappelons le, la plus grande plateforme de séquençage au monde est chinoise). Malgré quelques défauts, il s’agit d’un documentaire faisant tout d’abord oeuvre de pédagogie et développant des exemples plutôt précis comme autant d’arguments poussant à la large utilisation du séquençage haut-débit. Le documentaire vaut surtout pour l’accent mis sur la manière d’interpréter les résultats, sur la pertinence de ceux-ci mais tait les notions économiques liées à un futur marché qui s’annonce éléphantesque.
N’est ce pas un problème que votre génome soit, dans un avenir proche, séquencé, « interprété » et stocké par une firme privée ?
Le documentaire dont il est question est disponible ici-même (vous trouverez le synopsis dans les quelques lignes ci-dessous)
« LE DÉCRYPTAGE DU GÉNOME HUMAIN
D’ici peu, en échange de quelques centaines d’euros, chacun d’entre nous pourra obtenir un séquençage complet de son ADN. Des informations qu’il sera possible de lire, de stocker et de soumettre à l’analyse. Cette révolution est déjà en marche, ainsi que le montrent deux exemples spectaculaires : un patient cancéreux qui semble défier la mort et un malade de la mucoviscidose à présent capable de respirer normalement. Dans ces deux cas, les scientifiques sont parvenus à détecter et à neutraliser les anomalies génétiques à l’origine des maladies. Demain, grâce au décryptage du génome, il sera possible de faire un diagnostic personnalisé pour chaque individu et de mettre en place des traitements ultra personnalisés. Mais quelles sont les conséquences d’une telle révolution ? Connaître les maladies dont nous aurons à souffrir dans l’avenir, est-ce une bénédiction ou un fardeau ? Que se passerait-il si de telles informations tombaient entre les mains de compagnies d’assurance, d’employeurs ou de futurs conjoints ? Une chose est sûre : cette nouvelle ère, marquée par une médecine personnalisée s’appuyant sur nos gènes, est une réalité qui nous concerne tous. »
(Etats-Unis, 2012, 53mn)
ARTE F
Date de première diffusion : Jeu., 25 avr. 2013, 22h50
Date(s) de rediffusion : Dimanche, 5 mai 2013, 10h50
Pendant ce temps, de l’autre côté de l’Atlantique, Laurent Alexandre intervient dans l’émission grand public : ce soir (ou jamais !). Le débat avait pour thème l’obsolescence programmée. Ainsi au bout de 1h20 d’émission Frédéric Taddeï lance le débat sur l’obsolescence du corps humain. Après un extrait de l’assez mauvais film Repo Men de Miguel Sapochnik (réalisateur de certains épisodes de Dr House… c’est dire s’il doit s’y connaître en médecine personnalisée…), Laurent Alexandre, l’auteur du livre « la mort de la mort » et président de la société DNAVision, intervient ensuite. Son discours est clair et d’une grande pédagogie, il est peut être un peu dommage que l’exemple mis en avant fut le diagnostic prénatal non invasif de la trisomie 21 par séquençage haut-débit.
Evidemment très rapidement l’argument eugéniste se fait entendre. Finalement, l’intervention de Laurent Alexandre fait écho au documentaire américain.
Si les questionnements liés à la bioéthique sont au cœur du débat, rarement sont abordées les questions économiques et de confidentialité des données, associées à l’exploitation de nos données les plus intimes : la connaissance quasi-exhaustive de notre génome (et donc d’une partie de celui de nos descendants qui n’auraient rien souscrit).
Car depuis la publication de Gymrek et al., dans Science de janvier 2013 : Identifying personal genomes by surname inference, il semble difficile de soutenir l’anonymisation des données présentes dans les banques publiques. Il semble difficile d’imaginer que déposer son génome dans les banques d’une firme privée ne constitue pas un problème dont il faut d’ores et déjà prendre la mesure.
Car si tout le monde s’accorde sur les bienfaits potentiels de la médecine qui exploite la connaissance approfondie des bases nucléiques d’un patient ou d’un bien portant-futur mourant, actuellement le pouvoir est aux mains des firmes privées (Knome, 23andMe), qui se constituent des bases de données colossales. Ces bases de données hébergeant des quantités exponentielles de génomes humains (dans l’immédiat il s’agit plutôt de génotypes) permettront à leurs détenteurs de dominer un marché (voire plus !) qui s’annonce colossal…
 QIAGEN est un fournisseur très connu pour faire dans le haut de gamme du réactif de biologie moléculaire. Très connu pour ses colonnes de silice et sa polymérase plutôt robuste (HotStart Taq Plus). QIAGEN s’est lancé, à coups de rachats dans la qPCR et la robotique de pipettage, afin de mettre des instruments à son catalogue, instruments qui utiliseront ses consommables (on n’est jamais mieux servi que par soi-même). Ainsi la société allemande s’est payée Corbett Life Science, en 2008, 135 millions de dollars en cash. La société australienne a permis à QIAGEN d’enfin mettre à son catalogue une qPCR (à la technologie tout à fait singulière) et une robotique repeinte en bleu et gris.
QIAGEN est un fournisseur très connu pour faire dans le haut de gamme du réactif de biologie moléculaire. Très connu pour ses colonnes de silice et sa polymérase plutôt robuste (HotStart Taq Plus). QIAGEN s’est lancé, à coups de rachats dans la qPCR et la robotique de pipettage, afin de mettre des instruments à son catalogue, instruments qui utiliseront ses consommables (on n’est jamais mieux servi que par soi-même). Ainsi la société allemande s’est payée Corbett Life Science, en 2008, 135 millions de dollars en cash. La société australienne a permis à QIAGEN d’enfin mettre à son catalogue une qPCR (à la technologie tout à fait singulière) et une robotique repeinte en bleu et gris.
QIAGEN arrive sur le marché, mais encore une fois un peu tard, des séquenceurs haut débit de deuxième génération. La société de Francfort a donc racheté Intelligent Bio-Systems (IBS) dont la technologie est sous licence de Jingyue Ju (Université de Columbia). Cette licence propose une méthode permettant de diluer les molécules marquées (les terminateurs réversibles, ces mêmes molécules qui sont utilisées par la technologie d’Illumina dans Hiseq et Miseq). Ainsi les coûts des consommables s’en trouvent diminués.
Le Mini20 comme se nomme la plateforme développée par IBS, a tout d’une grande. Les coûts annoncés, il y a un an de cela, avoisinaient les 300 $ / Gbase (à la même date sur Miseq, les 600 $ / Gbase étaient dépassés). La technologie rachetée par QIAGEN permet d’utiliser « 20 flow-cells » , adressables individuellement pour un débit de 20 M reads / flow cell, débit total de 80 Gbases. Le séquenceur était annoncé à un prix de 120 k$ venant clairement marcher sur les plates bandes d’un Miseq. A n’en pas douter, l’arrivée d’un poids lourd comme QIAGEN sur le marché du séquençage haut-débit de 2ème génération devrait dynamiser et quelque peu perturber le ménage à deux de Life Technologies et Illumina. Certes, QIAGEN, avec ses solutions de préparations d’échantillons (chimie et instruments de pipettage), serait un nouvel acteur proposant des solutions complètes (et notamment en bio-informatique grâce à un partenariat avec SAP).
L’ambition de QIAGEN semble être de fournir une solution complète de l’échantillon à l’analyse des séquences que leur Mini-20 aura généré. Il faut juste espérer que les perspectives de croissance sur le marché des séquenceurs de 2ème génération ne soient pas anéanties par l’arrivée d’un séquenceur de 3ème génération à coût raisonnable. QIAGEN arrive tard dans la course mais avec une offre « tout inclus » qui serait susceptible de faire mouche pour qui n’a pas accès à un plateau technique de biologie moléculaire ou encore à des compétences en bio-informatique.
 Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Quelques médias d’outre-Manche évoquent cette fameuse technologie de séquençage en l’affublant du qualificatif de « future grande invention britannique » : lire à ce sujet la page web de « The Raconter » : Britain greatest inventions ». Cette invention est associée à la seule médecine personnalisée comme pour envisager le futur marché du séquençage haut-débit.
Les voyants semblent donc au vert pour Oxford Nanopore. Pour préparer le terrain, sort en ce début d’année, un article de Nature Methods : « disruptive nanopores« . Un titre qui fait écho à celui de Forbes (février 2012) repris dans notre image ci-dessus.
Nicole Rusk, rédactrice en chef à Nature Methods, vient avancer les principales caractéristiques du futur produit :
– des reads entre 10 et 100 kb
– des taux d’erreurs entre 1 et 4 %
– la possibilité de connaître les bases méthylées
– la possibilité de séquencer directement l’ARN
– la méthode est non destructive

La technologie de séquençage de 3ème génération par nanopores promet de révolutionner plusieurs applications à commencer par le séquençage de novo en laissant peu d’espoir à la technologie de Pacific Biosciences. Elle devrait remplacer PacBio dans les stratégies de séquençage hybride (qui consiste « à coupler » un séquenceur de 2ème génération permettant d’être très profond et d’une technologie de 3ème génération permettant de générer des reads très longs ce qui permet au final un assemblage de meilleure qualité).
Après avoir engendré de la curiosité, de l’impatience, puis déçu avec une arrivée sans cesse repoussée, il semble qu’Oxford Nanopore doive prouver de l’efficacité de sa technologie. Ainsi, la société britannique a annoncé le 8 janvier 2013, une série d’accords avec plusieurs institutions telles que l’ Université de l’Illinois, l’Université Brown, l’Université de Stanford , l’Université de Boston, de Cambridge et de Southampton. Oxford Nanopore prend son temps ou rencontre des difficultés avec son exonucléase. Malgré ses dizaines de brevets, pour conserver sa crédibilité la société a dû communiquer pour convaincre de l’efficience de sa technologie en minimisant les difficultés de développements, en alimentant les tuyaux de communication avec des séquenceurs sorti de palettes graphiques loin d’être finalisés.
Ce retard de lancement s’apparente t’il à un gage de sérieux ou est-il la preuve que le séquençage par nanopores rencontre de grosses, très grosses difficultés de développement ? Réponse en 2013.
La nouvelle provient de l’Institut Italien de Technologie de Gènes : l’équipe du professeur Enzo di Fabricio (responsable du département de Nanostructures) a réussi à capturer l’image d’une molécule d’ADN à l’aide d’un microscope électronique en transmission et d’un support en silicone hydrophobe.
L’équipe a développé un support ( formé de colonnes dites « nanopiliers » ) extrêmement hydrophobe, provoquant ainsi l’évaporation, pour ne laisser visibles que des brins d’ADN tendus. L’équipe a également percé de petits puits dans le fond du support « nanopilier » (voir la photographie du support ci-dessous), à travers lequel ils font pénétrer des faisceaux d’électrons pour obtenir leur capture en haute résolution.
Cette image est générée en utilisant la technologie de microscopie TEM pour « transmission electron microscopy » qui repose sur le principe de transmission d’un faisceau d’électrons à travers un support très mince. Plus précisément une image est formée à partir des d’interactions entres les électrons et le support; le résultat peut ensuite être imprimé sur un film photographique ou capturé via un détecteur numérique. Cette technologie offre une grande résolution indispensable pour visualiser un brin d’adn.
Sur l’image en en-tête on repère bien la forme hélicoïdale de la double-hélice d’ADN.
Outre l’aspect « inédit » de cette technique, elle ouvre également des perspectives intéressantes: on pourrait être ainsi en mesure d’observer (réellement) la façon dont une molécule d’ADN interagit avec diverses biomolécules.
Pour en savoir plus : l’article « Direct Imaging of DNA Fibers: The Visage of Double Helix » publié dans Nanoletters (American Chemical Society) en Novembre 2012
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages