 En guise d’épilogue à nos quelques évocations de l’Agence d’Evaluation de la Recherche dans l’Enseignement Supérieur, voici une copie d‘un article du Monde du 03 janvier 2013 sobrement titré : Fin de vie pour l’Aéres.
En guise d’épilogue à nos quelques évocations de l’Agence d’Evaluation de la Recherche dans l’Enseignement Supérieur, voici une copie d‘un article du Monde du 03 janvier 2013 sobrement titré : Fin de vie pour l’Aéres.
L’article évoque l’élément du discours, où au dîner du 20 décembre 2012, à la Conférence des Présidents d’Université, Geneviève Fioraso a scellé définitivement le cercueil de l’Aéres qui agonisait depuis sa naissance. La ministre de l’Enseignement Supérieur et de la Recherche a déclaré à cette occasion : « la simplification des procédures de l’évaluation implique également des mesures législatives. Ajoutant : Je souhaite à cet égard que l’AERES actuelle soit remplacée par une agence nationale entièrement redéfinie à partir des principes d’indépendance, de simplicité de fonctionnement et de procédures ainsi que de légitimité scientifique et de transparence «
Ces propos font suite à la publication, fin septembre 2012, par l’Académie des Sciences, d’un rapport où l’AERES est présentée comme un « machin » bureaucratique et peu efficace, préconisant sa suppression.
Dans cette période de transition, il est difficile d’avoir une visibilité sur les outils qui seront mis en place pour évaluer la recherche scientifique. Il faut dire que l’idée de publier les notes des évaluations a eu l’effet prévisible d’engendrer un statu quo contre-productif pour ce qui est de l’objectif fixé : l’innovation… plus une unité est bien évaluée, mieux elle sera financée, donc mieux elle sera évaluée… Ce type d’évaluation engendre des circonvolutions plus que des innovations.
Sur le portail de l’AERES aucune ligne sur la mort annoncée de l’agence.
L’Aéres en chiffres :
Bilan :
Entre 2007 et 2011, l’Aéres a évalué 3 960 formations, 3 196 unités de recherche, 5 483 équipes de chercheurs, 325 établissements d’enseignementsupérieur et 18 organismes de recherche.
Notations :
Sur les 2 613 unités de recherche auditées entre 2008 et 2011 :
21,5 % ont obtenu la note A + ; 43,6 % la note A ; 28 % B ; et 5,8 % C.
Pour les 3 740 cursus (licences, masters et doctorats), 6 % de A + ; 44 % de A ; 41 % de B et 9 % de C.
Moyens :
5 900 experts (18 % étrangers), un budget de 17 millions d’euros, 170 salariés.
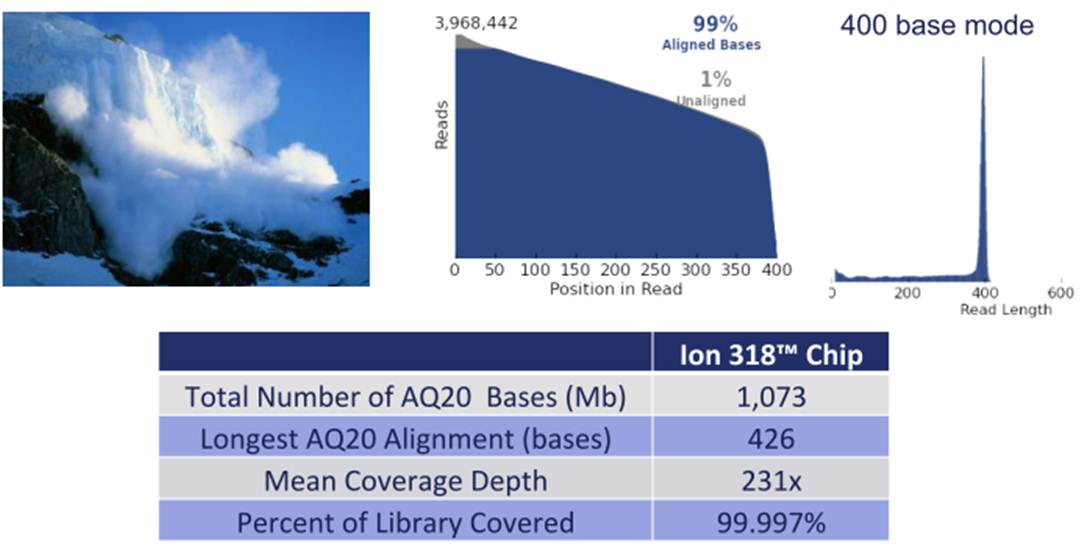
 Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Ion PGM™ Template OT2 400 Kit) qui est principalement attendu pour les applications de séquençage de novo et de séquençage d’amplicons ciblant le 16S, 18S ou encore ITS2 (respectivement pour l’étude de la diversité bactérienne, eucaryote ou fongique) pour les études métagénomiques. Comme vous pouvez le constater cette application est uniquement supportée par le One Touch 2 devenu nécessaire pour accéder aux reads de 400 bases (c’est en partie pour cette raison que notre plateforme PEGASE-Biosciences a décidé de se doter de cette automate).
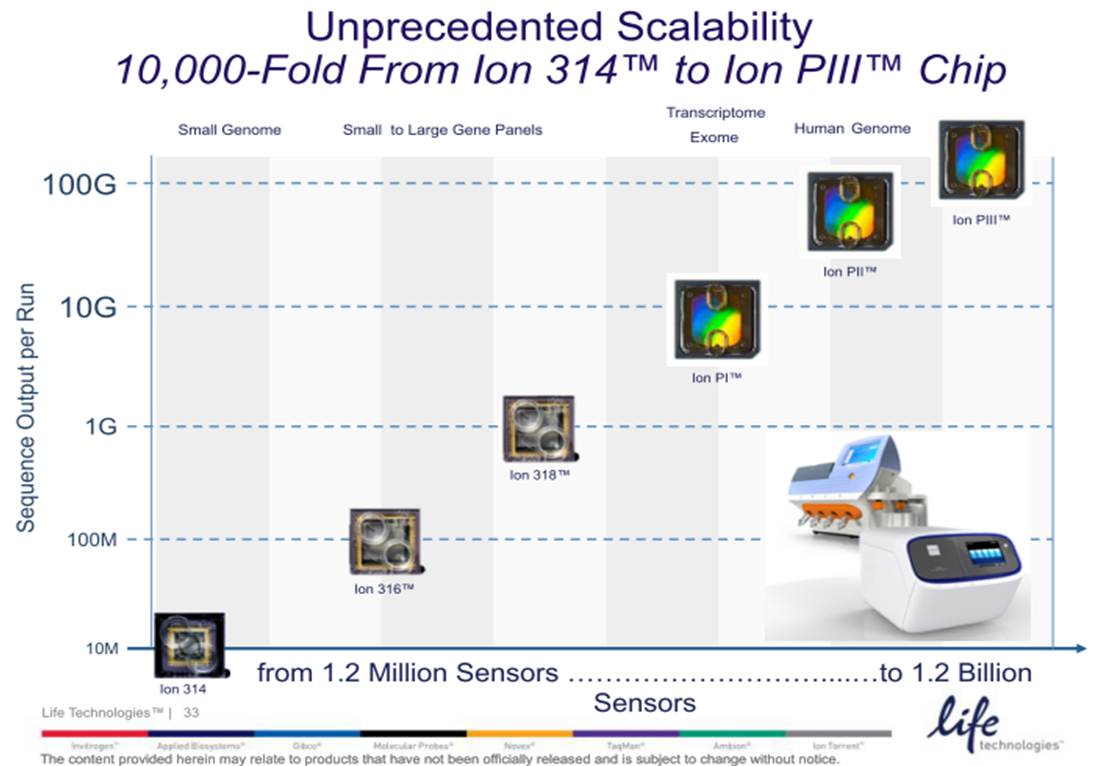
L’autre nouveauté est portée par les consommables de séquençage associés au Ion Proton (le grand frère du PGM). En effet, si la Puce Ion PIII est encore virtuelle, la PI et PII, permettent de délivrer : 10 Gb et 70 millions de reads filtrés environ pour la première, contre plus de 280 de millions de reads et plus de 70 Gb pour la seconde (de quoi séquencer un génome humain à 20 X).
Du côté de Life Technologies et de sa technologie Ion Torrent, sans aucun doute, la principale nouveauté de l’année 2013 touche l’amplification clonale (phase où, à partir de la librairie préparée, est réalisée la matrice de séquençage fixée de manière covalente sur des billes qui se logeront dans les puits présents sur la puce). Ainsi, la fameuse (et pour certains la fâcheuse) phase de PCR en émulsion trouvera une alternative avec la chimie isothermique dite « avalanche » (dont vous pouvez voir l’aperçu d’un run ci-dessous). A en croire Life Technologies, cette chimie ouvre la voie à des tailles de séquences générées encore plus longues.
Bien entendu, Life Tech annonce l’arrivée d’un nouvel automate permettant d’intégrer la nouvelle chimie mais aussi de charger les puces (et pour quelques dizaines de k€, d’épargner au manipulateur les deux phases délicates du séquençage).
Pour conclure brièvement, l’une des nouveautés et non des moindres : l’acceptation du PGM comme diagnostic moléculaire CE-IVD (un parcours pas vraiment de tout repos) a n’en pas douter une conformité qui ne devrait pas laisser insensible les chercheurs, praticiens hospitaliers.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages

