La toute puissante FDA (Food and Drug Administration) a calmé les ardeurs la société 23andMe en remettant en question la diffusion par la société californienne de tests génomiques personnels. (Étonnamment ?), la FDA ne semble pas réellement heurtée par le principe même d’une société de droit privé glanant des informations génomiques pour réaliser un commerce des plus lucratifs, motus concernant le devenir de ce type de données. En réalité, l’administration américaine, soucieuse du service rendu au consommateur, émet des doutes quant à la pertinence, quant à l’exploitation des résultats, après génotypages, des individus consentants, ainsi que sur la façon dont le tout est présenté au consommateur. Ainsi, 23andMe est mise en cause au niveau de la qualité de son service. Voici son seul « crime » aux yeux de la FDA!
La toute puissante FDA (Food and Drug Administration) a calmé les ardeurs la société 23andMe en remettant en question la diffusion par la société californienne de tests génomiques personnels. (Étonnamment ?), la FDA ne semble pas réellement heurtée par le principe même d’une société de droit privé glanant des informations génomiques pour réaliser un commerce des plus lucratifs, motus concernant le devenir de ce type de données. En réalité, l’administration américaine, soucieuse du service rendu au consommateur, émet des doutes quant à la pertinence, quant à l’exploitation des résultats, après génotypages, des individus consentants, ainsi que sur la façon dont le tout est présenté au consommateur. Ainsi, 23andMe est mise en cause au niveau de la qualité de son service. Voici son seul « crime » aux yeux de la FDA!
L’administration américaine lui reproche :
– malgré les sollicitations récurrentes de la part de la FDA, 23andMe n’a pas fait valider par l’administration ses dispositifs diagnostiques in vitro. Elle n’a cessé d’allonger la liste (stratégie commerciale du restaurant chinois) des maladies et caractères que leur système était susceptible de diagnostiquer -avant « sa mise en demeure », 23andMe proposait un menu comportant le « diagnostic » de 254 prédispositions à des « maladies » ou aptitudes diverses.
– au sujet des maladies multi-factorielles, telles que le cancer de l’ovaire ou du sein, les résultats rendus aux clients sont très probablement constitués d’un grand nombre de faux positifs et de faux négatifs.
la FDA souhaite que l’intégralité des assertions de 23andMe soit validée pour que la société continue de vendre son kit phare : health-related genetic tests.
La société qui comptait régner en maître sur le marché de la génomique récréative, a peut-être fini de jouer les apprentis sorciers (franchement peu probable !). En effet, si l’intérêt de certains de ces tests génomiques, en soi, n’est pas à remettre en question, leur encadrement législatif est à souhaiter. En outre, avant de valider des kits « diagnostiques », il serait appréciable de lever le quiproquo : les kits proposés par 23andMe sont ils des dispositifs de génomique récréative ou des dispositifs médicaux ? La libéralisation incontrôlée de ce type de marchés pourrait avoir des conséquences dramatiques… Si des prédispositions peuvent être inscrites dans notre code génétique, il n’en demeure pas moins qu’une large majorité de maladies peut être qualifiée de multifactorielle.
Se sentir à l’abri d’une maladie parce que 23andMe vous rend un résultat dans ce sens ou dans le cas contraire, sentir l’ombre de l’épée de Damoclès, peut avoir, pour le consommateur de ces tests, des conséquences graves.
La lettre d’avertissement du 22 novembre 2013, de la FDA adressée à Anne Wojciki, montre l’étendue de l’ambiguïté : LETTRE d’AVERTISSEMENT ainsi que la réponse de 23andMe.
Anne Wojcicki a déclaré que « 23andMe travaille activement avec la FDA pour s’assurer que la firme fournit des informations de haute qualité afin que les consommateurs puissent avoir confiance ».
Ci-dessous, voici la nouvelle page qui vous accueille sur le site de 23andMe qui peut continuer à vendre ses tests phylogénétiques.
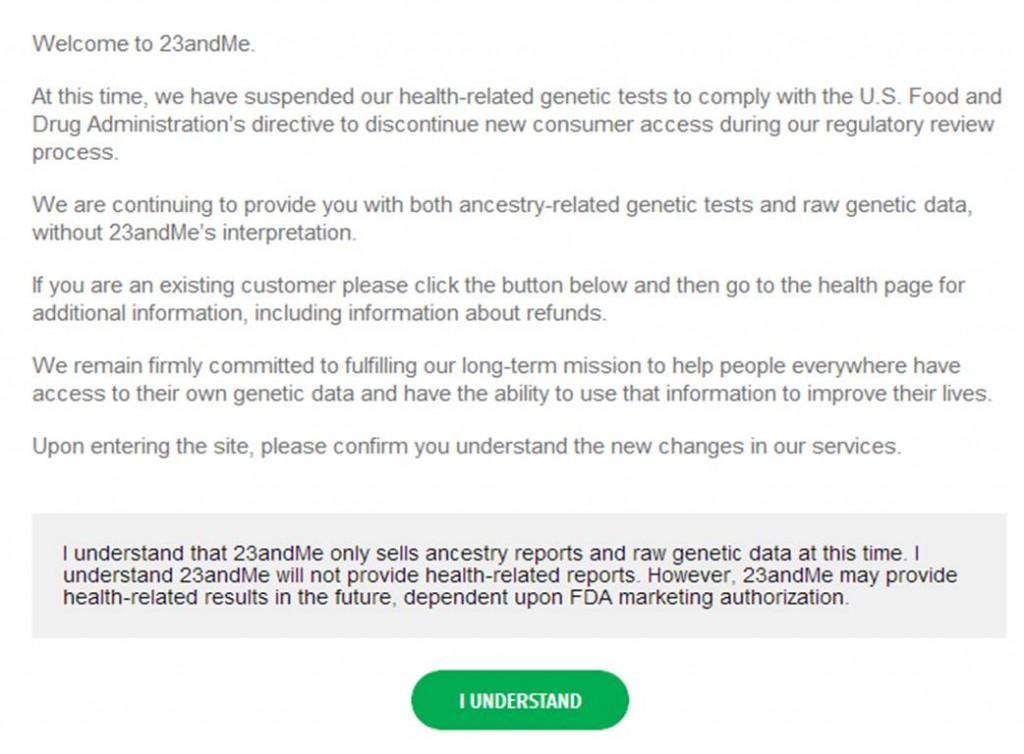
Comme le disait Stephen Hawking :
« J’ai remarqué que même les gens qui affirment que tout est prédestiné et que nous ne pouvons rien y changer regardent avant de traverser la rue. » (Trous noirs et bébés univers, Éditions Odile Jacob, 1994)
 Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
En 2010, David Weitz et son équipe de l’Université d’Harvard ambitionnent de développer une nouvelle technologie de séquençage à haut-débit, alliant les technologies de biologie moléculaire aux procédés de microfluidique développés quelques années plus tôt (2004) au sein de la société RainDance technologies.
Cette nouvelle approche repose sur la capacité à générer des gouttes de l’ordre du picolitre et pouvant être déplacée sur une puce microfluidique. Ces gouttes peuvent renfermer soit un couple d’amorces, des adaptateurs, ou tout autre type de réactifs nécessaires aux étapes de préparation de librairie et de séquençage (séquençage par hybridation-ligation, type SOLiD avec une fidélité de 99.99%). Dès lors, leurs quantités utilisées au sein de ces picogouttes sont considérablement revues à la baisse, ce qui constitue le point clé à une réduction des coûts de séquençage et donc la perspective d’un séquençage de génome humain à 30$, selon David Weitz.
Les projets de GnuBio sont désormais d’élargir le champs d’applications de leur séquenceur à l’analyse transcriptomique (RNA-seq), l’étude de la méthylation (ChiP-seq) ou encore le séquençage de génome entier. La société ambitionne une commercialisation de leur équipement au cours de l’année 2014.
A suivre…

Le séquençage haut-débit voit cohabiter depuis quelques années deux générations de séquenceurs.
Au passage, une question Trivial Pursuit pour laquelle il faudra avoir un œil de caracal : quelqu’un sait quelle société a développé la première génération de séquenceurs haut-débit ? et quand ?
Les séquenceurs de deuxième génération se voient conditionnés sous forme de séquenceurs de paillasse (PGM de Ion Torrent, Miseq d’Illumina, GS-junior de Roche) permettant une démocratisation du séquençage, pendant que leurs grands frères pulvérisent la loi de Moore pour envisager un rendement (coût / Mb) toujours plus compétitif.
La large diffusion du séquençage de 3ème génération se laisse désirer laissant le champ libre à la génération précédente. Cet article vise à réaliser un court état des lieux du séquençage haut-débit de troisième génération : un futur plus ou moins lointain, de nouvelles applications potentielles.
La question : séquenceurs de 3ème génération, l’âge de raison, c’est pour quand ? est l’interrogation qui a hanté l’AGBT 2013 marqué par le silence d’Oxford Nanopore. Cette année 2013 fut marquée par le retrait d’Illumina du capital de la société britannique : « Oxford Nanopore Technologies Ltd a annoncé la vente d’une participation détenue par son concurrent américain Illumina Inc., une étape vers la fin d’une relation pleine de conflits dans la course au développement des séquenceurs haut-débit permettant de séquencer plus rapidement et pour moins cher. »
Avant de caractériser ce que sont, seront, pourront être les 3ème générations de séquenceurs, commençons par un rapide tour des caractéristiques générales de leurs prédécesseurs et principalement de ce qui constitue leurs points faibles :
– la phase d’amplification clonale (réalisée par PCR) est source de biais (doublons, erreurs de PCR)
– les problèmes liés au déphasage engendrant une chute de la qualité le long du read produit (ce qui bride la production de reads vraiment longs)
-des reads courts (de moins d’une centaine à environ 800 bases – vous l’aurez noté ce point est en partie une conséquence du précédent)
-des machines et des consommables onéreux
– des temps de run longs
Ainsi l’objectif principal des séquenceurs de 3ème génération est de palier les défauts de leurs aînés en produisant des reads plus longs, plus vite pour moins cher. Les séquenceurs de 2ème génération, quels que soient leurs modes de détection (mesure de fluorescence, mesure de pH) sont trop peu sensibles pour envisager la détection d’une simple molécule, d’un simple nucléotide : nécessairement la librairie doit être amplifiée, ce qui provoque des biais, des temps de préparation relativement longs et l’usage de consommables qui impacte le coût final de séquençage… assez rapidement la qualité chute plus vos reads s’allongent ce qui oblige à brider les tailles de reads que ces technologies sont capables de délivrer. En outre, travaillant sur une matrice qui est une copie de votre librairie initiale, l’information portée par les bases méthylées est perdue (ceci oblige à ajouter une phase de traitement au bisulfite qui peut être hasardeuse)
Actuellement l’une des seules technologies de 3ème génération réellement utilisée est celle de Pacific Biosciences (les hipsters disent « PacBio »). La firme, fondée en 2004, a lancé en 2010, son premier séquenceur de troisième génération le Pacbio RS basé sur une technique de séquençage SMRT (Single Molecule Real Time sequencing.) Aujourd’hui la société Roche qui n’a pu absorber Illumina lors de son OPA, a investi 75 millions de USD, le 25 septembre 2013, pour co-développer des kits diagnostiques in vitro exploitant la technologie de PacBio.
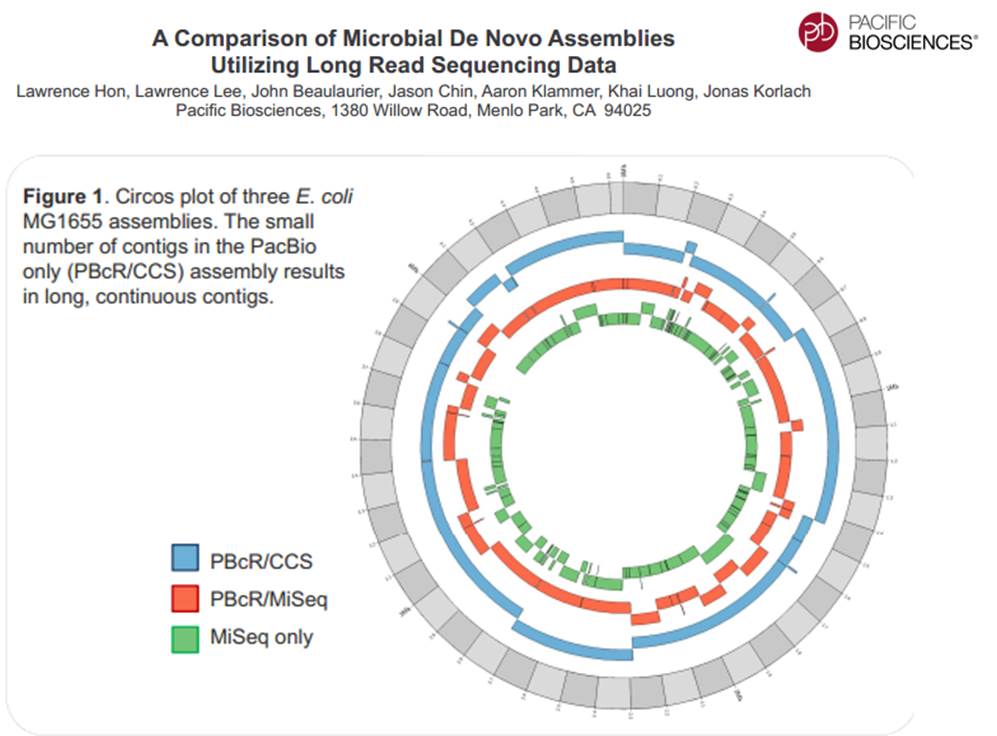
La technologie de PacBio est aujourd’hui exploitée pour réaliser du séquençage de novo de petits génomes :
Avec ces 200 à 300 Mb délivrés par SMRT-cell, séquencer des organismes eucaryotes supérieurs demande un investissement important, malgré tout, cette technologie délivrant des reads de plusieurs milliers de bases, permet d’envisager une diminution du nombre de contigs obtenus par les seules stratégies reads-courts / gros débit.
Face à la technologie proposée par PacBio, d’autres technologies essaient d’émerger pour arriver à occuper le marché du séquençage de 3ème génération :
– La combinaison détection optique et multipore est une voie envisagée pour le séquençage de 3ème génération avec le travail mené par NobleGen biosciences.
– L’imagerie directe de l’ADN
Le microscope électronique offre une résolution possible jusqu’à 100 pm, de sorte que les biomolécules et les structures microscopiques tels que des virus, des ribosomes, des protéines, des lipides, des petites molécules et des atomes même simples peuvent être observés. Bien que l’ADN est visible lorsqu’on l’observe avec un microscope électronique, la résolution de l’image obtenue n’est pas suffisamment élevée pour permettre le déchiffrement de la séquence, c’est à dire, le séquençage de l’ADN. Cependant, lors du marquage différentiel des bases de l’ADN avec des atomes lourds ou des métaux, un tel séquençage devient possible.

– Le séquençage à l’aide de transistor (Transistor-mediated DNA sequencing– une technologie développée par IBM)

Dans le système conceptualisé par IBM, l’ADN est contraint de passer par le pore à cause de la tension électrique subie, la vitesse de passage de la molécule à séquencer est maîtrisée à l’aide de contacts métalliques à l’intérieur du nanopore. La lecture des bases serait réalisée lors du passage de l’ADN simple brin au travers du pore (ça rappelle quelque chose…)
– Et Oxford Nanopore dans tout cela ? Si la société anglaise a annoncé la vente de la participation d’Illumina, elle a marqué l’année 2013 par son silence assourdissant. Passé l’oxymore, en cette fin d’année, coup de poker ou réel lancement, Oxford Nanopore propose un programme d’accès à sa technologie Minion où pour 1000 USD, il est possible de postuler à l’achat des clés USB de séquençage.
La stratégie d’Oxford Nanopore est basée, en partie, sur la possible démocratisation du séquençage de 3ème génération, elle s’oppose à celle de PacBio qui mise sur son arrivée précoce sur le secteur du séquençage haut-débit : décentralisation contre l’inverse. En clair, l’investissement d’un PacBio est tel que l’outil est réservé à des centres, des prestataires de services pouvant assumer cet investissement, ce qui oblige à centraliser les échantillons pour les séquencer, contre les produits (encore en développement) d’Oxford Nanopore dont la promesse est : le séquençage pour tous (ou presque).
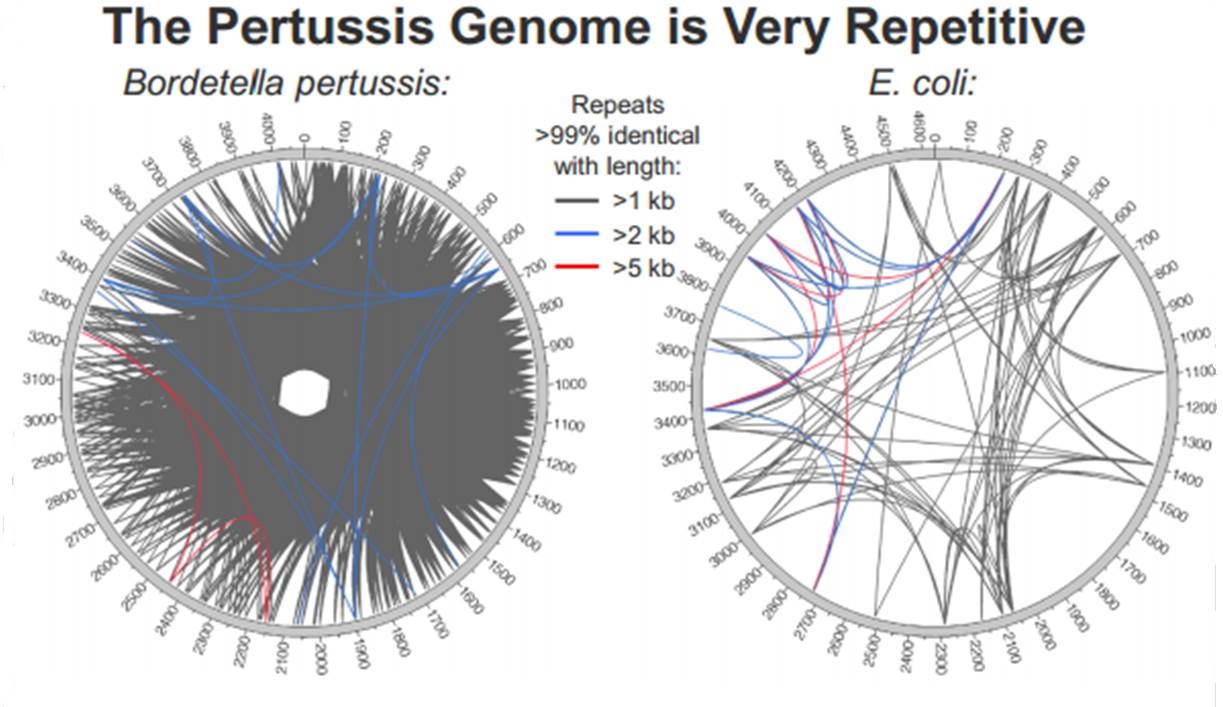
PacBio revendique sa participation à un projet qui consiste à doubler la quantité de génomes bactériens « terminés » (actuellement de 2384) en quelques mois.
En cliquant ci-dessus sur la représentation graphique qui illustre la différence de plasticité de génome entre le génome d’une Bordetella pertussis (l’agent pathogène responsable de la coqueluche) et celui d’Escherichia coli, un poster vous apparaîtra. Ce dernier reprend les caractéristiques de l’utilisation de la technologie de PacBio à des fins d’assemblage de novo de génomes bactériens par une stratégie non-hybride (seuls des reads de PacBio sont utilisés). Les résultats sont assez bluffants, la longueur des reads de PacBio permet un assemblage complet (au prix de plusieurs SMRT cells tout de même !), de génomes bactériens « difficiles » tel que celui de Bordetella pertussis connu pour posséder un GC % relativement élevé (environ 65 %) ainsi que de nombreux éléments transposables. Les génomes possédant de nombreux éléments répétés posent de grandes difficultés d’assemblage, c’est un des arguments qui permet à PacBio de positionner sa technologie actuellement… en quelques mois les stratégies hybrides (reads courts générés par des séquenceurs de 2ème génération) ont laissé place aux stratégies non-hybrides où le séquençage PacBio se suffit à lui-même.
L’évolution des technologies de biologie moléculaire combinée à des systèmes de détection toujours plus sensibles, des techniques d’amplification accessibles dans des systèmes intégrés ont rendu possible l’analyse du transcriptome d’une cellule isolée. Dans de nombreux domaines tels que la cancérologie, la biologie des cellules souches, l’ingénierie des tissus, le signal moyen peut cacher la pertinence du signal noyé dans la masse des cellules d’intérêt.
Défier la loi de la moyenne, en tenant compte de l’hétérogénéité cellulaire pour tenter de capter ce signal qui échappe aux systèmes n’analysant que le mélange de cellules, est le credo du système proposé par Fluidigm : le C1™ Single-Cell Auto Prep System.
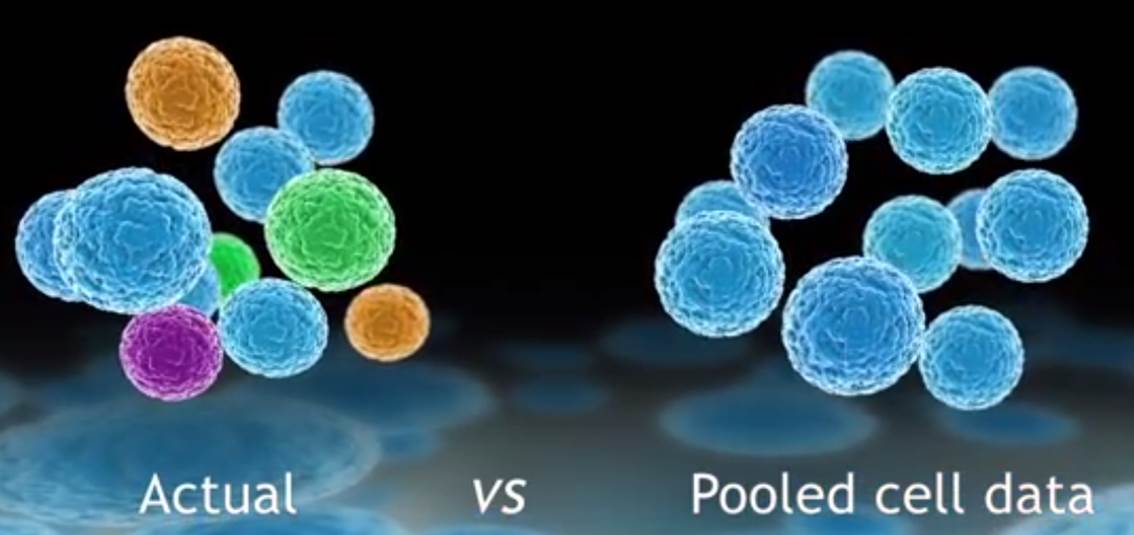
Isoler des cellules pour extraire leur ARN peut être une tâche technique ardue consommatrice de temps et très sensible aux contaminations.
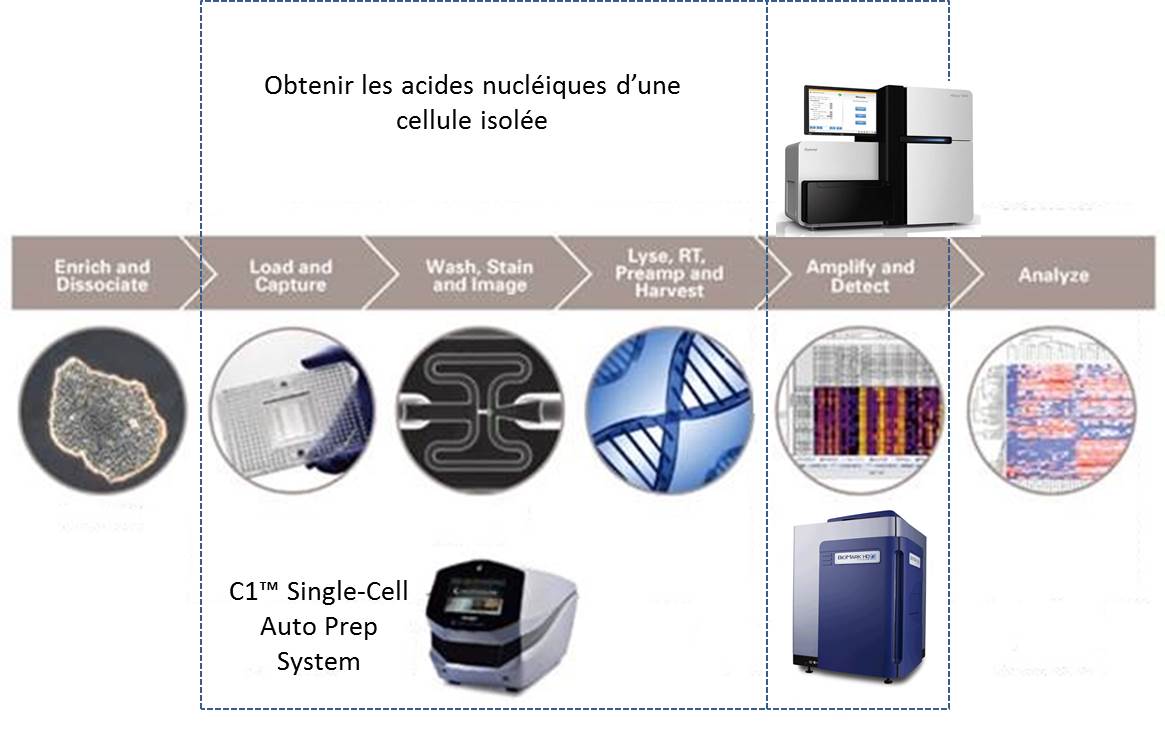
La société américaine, Fluidigm propose donc un système plutôt simple permettant de capturer quelques cellules pour en extraire leurs acides nucléiques. Le consommable est composé d’une plaque microfluidique comportant 96 pièges à cellules enchaînés. La multiplicité de ces pièges permet d’accroître les chances de capturer une cellule dans l’état escompté. Une fois la phase de capture accomplie, les divers pièges sont individualisés. Un système de by-pass permet de « pousser » chacune des cellules piégées dans une succession de « microréacteurs » isolés enchaînant phases de lyse, de transcription inverse, d’amplification des ARNs (WTA) et de collecte des acides nucléiques amplifiés.
Le schéma ci-dessous reprend les 2 phases principales permettant d’aboutir à l’analyse du transcriptome de cellules isolées :
(i) l’amplification d’ARN de cellules isolées
(ii) l’analyse de ces ARNs
Ainsi que le montre la vidéo promotionnelle ci-dessous le système est compact et se veut simple d’utilisation. Il va de soi, malgré tout, que pour optimiser vos chances de capturer un signal d’intérêt un marquage préalable cellules vivantes/mortes peut s’imposer. Ceci impose aussi d’observer la plaque microfluidique au microscope à fluorescence (en se hâtant quelque peu). Bien évidemment l’adjonction de marqueurs discriminants (anticorps couplés à un agent fluorescent) est la bienvenue.
Cette dernière vidéo aborde le « workflow » aboutissant à l’analyse du transcriptome monocellulaire par Biomark, il va de soi qu’il s’agit là d’une « suggestion de présentation » puisqu’il est possible d’analyser le transcriptome amplifié par le système C1™ Single-Cell Auto Prep System avec un grand nombre de systèmes de qPCR ou de séquençage haut-débit.
http://youtu.be/TF4NJRE4Xg4
L’analyse des biomarqueurs des cellules tumorales circulantes devient un élément majeur de la médecine personnalisée (lire L’enjeu des cellules tumorales circulantes). L’article d’Ashley A. Powell et al. (Single Cell Profiling of Circulating Tumor Cells: Transcriptional Heterogeneity and Diversity from Breast Cancer Cell Lines (Plos One, 2102)) montre l’application de ce système au niveau de l’analyse de « biopsie liquide« .
L’outil proposé par Fluidigm trouve d’ores et déjà des applications cliniques très concrètes : l’analyse de biomarqueurs des cellules tumorales circulantes à visée pronostique.
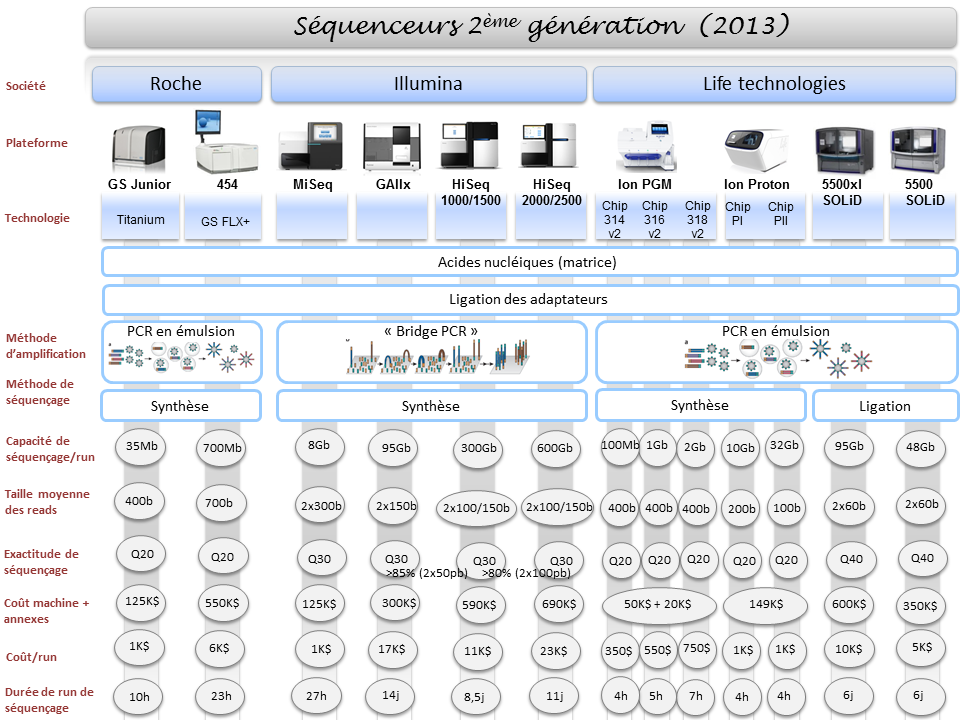
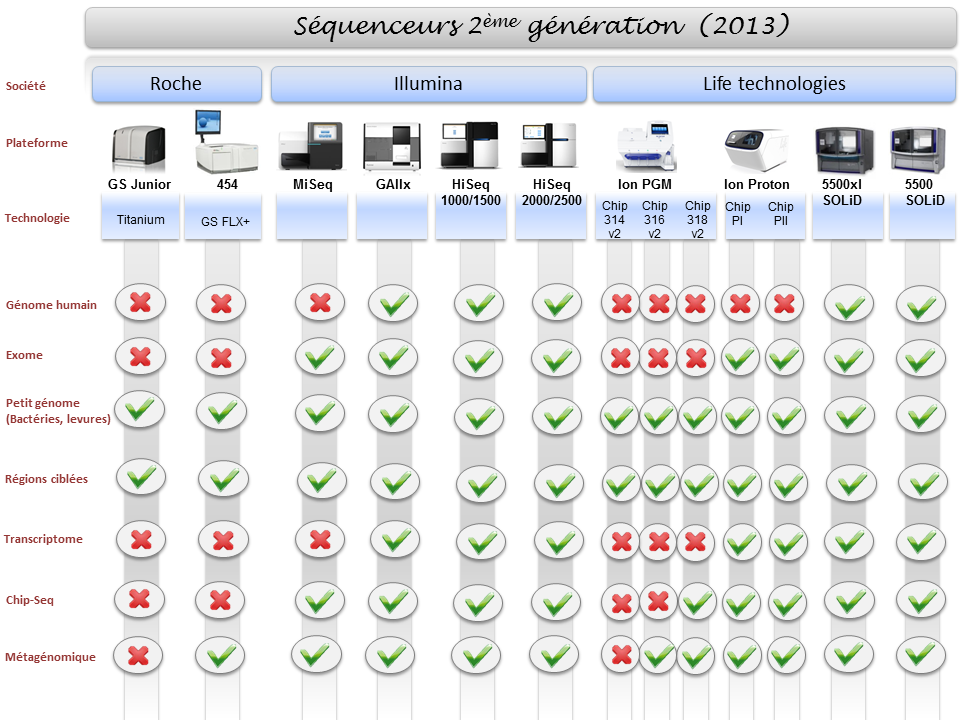
La diversité du parc technologique des séquenceurs de deuxième génération n’est plus une surprise pour personne. Ceci étant, il devenait indispensable de mettre à jour l’ensemble des informations postées sur ce site, il y a exactement deux ans (2011), faisant un état de l’art des différentes caractéristiques technologiques des séquenceurs, ainsi que les possibles applications biologiques associées.
Pour ne mentionner que les trois plus gros fournisseurs du marché, les sociétés Roche, Illumina et Life Technologies n’ont cessé de faire évoluer leur gamme, tant sur le plan des équipements que sur le plan des capacités de séquençage.
Par voie de conséquence, ce survol est l’occasion de refaire le point sur les technologies appropriées selon l’application biologique recherchée. A noter que le Ion proton, dernier en date sur le marché des séquenceurs de deuxième génération disposera au cours de l’année 2014 d’une puce « PIII » permettant de générer environ 64Gb. Cette capacité de séquençage permettra à Ion torrent de se positionner sur le séquençage de génome humain à partir d’un séquenceur de paillasse et accèdera ainsi à la gamme complète des applications citées ci-dessous.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages