Vous qui cherchez une méthode permettant de mettre en évidence des variants rares au sein d’une population hétérogène de produits PCR, l’ice-COLD PCR est peut être faite pour vous.
En effet, identifier des variants rares noyés dans de l’ADN « sauvage » a été techniquement une demande forte de divers champs d’expertises médicales tels que la cancérologie, l’infectiologie et le diagnostic prénatal. Nous avons déjà parlé sur Biorigami, d’une approche assez largement répandue qui consiste à séquencer très profondément (à haut-débit donc) tout ou partie de génome en vue d’identifier ces variants rares potentiellement associés à des phénomènes de résistance à des antibiotiques (à titre d’exemple vous pouvez consulter le travail d’Eurekagenomics présenté sous forme de poster)
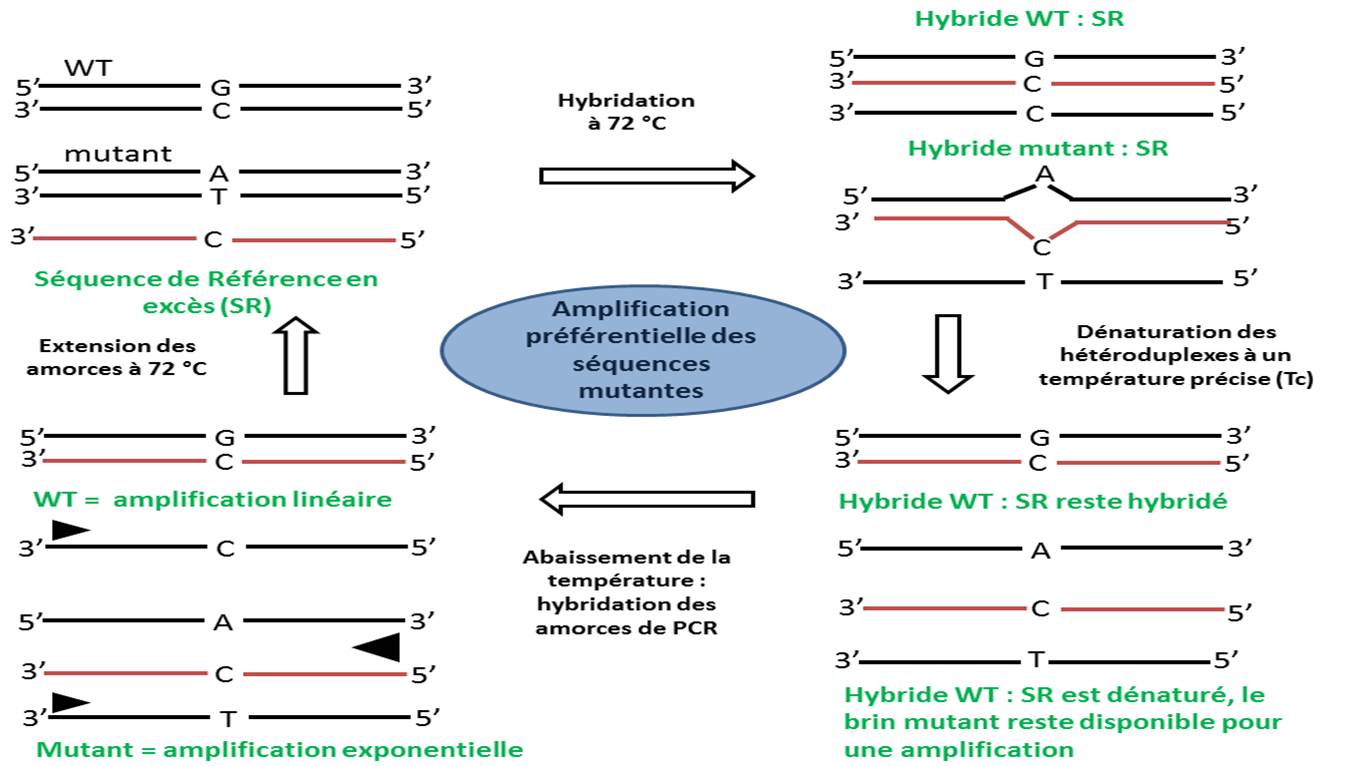
Derrière l’acronyme tautologique, ice COLD, se cache la terminologie : Improved & Complete Enrichment CO-amplification at Lower Denaturation temperature PCR, ce qui pourrait se traduire en français approximatif par : une amélioration de la COLD PCR qui est elle même : une co-amplification à sub-température de dénaturation-PCR. Qu’est ce qui se cache derrière cet énième acronyme ? Pour y voir plus plus clair, voici pour commencer, ce petit schéma:
En observant le schéma ci-dessus, commençons en haut à gauche : nous avons donc des séquences double brin sauvage en large quantité et potentiellement de l’ADN mutant. En premier lieu, il convient de designer des amorces permettant une amplification du locus d’intérêt (pour un amplicon de taille généralement autour de 100 pb). Il vous faudra ensuite une séquence (notée SR sur le schéma) 3′ phosphate pour prévenir des amplifications lors des phases de PCR. Cet oligonucléotide (employé autour d’une concentration de 25 nM finale) viendra s’hybrider parfaitement sur la cible ADN wt et constituera un hétéroduplexe avec toute cible contenant un polymorphisme. A l’aide de la connaissance du Tm (melting temperature ou température de fusion) de l’homoduplexe ADNwt//SR -(note : pour ce faire, il sera nécessaire de designer un autre couple d’amorces afin que la taille du brin SR amplifiée soit égale au brin SR qui lui même est plus court que l’amplicon généré par les premières amorces designées) acquise par la réalisation de courbe de fusion par PCR en temps réel, les hétéroduplexes seront dénaturés en chauffant à une température critique (Tc), généralement de 1 °C inférieure au Tm constaté de l’homoduplexe. Nous sommes, à l’issue de cette étape, en bas à droite de notre schéma, ensuite il restera à réaliser la PCR qui ciblera largement préférentiellement les ADNs disponibles, dénaturés… donc ceux portant des mutations.
Les avantages de cette technique sont multiples :
Cette technique peut être appliquée sur diverses types d’échantillons : ADN fœtal circulant (sérum, plasma), cellules tumorales circulantes, divers fluides corporels, FNA, FFPE, TMA. Elle nécessite de petites quantités de tissu ou d’ADN et peut permettre détecter toutes les mutations présentes au sein du locus amplifié.
En aval, cette méthode accepte une foultitude de techniques de détection qui permettent de réaliser le diagnostic à proprement parlé des mutations :
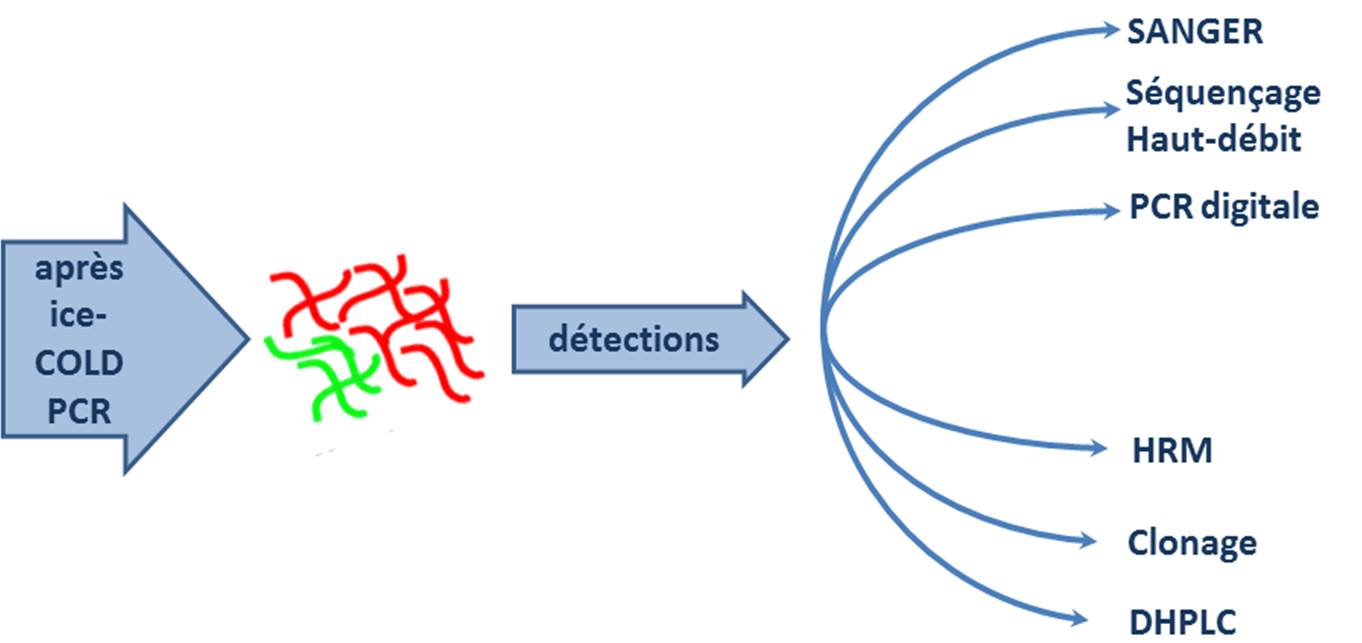
L’ice-COLD PCR est une technique qui est peu exigeante, elle nécessite de disposer d’un thermocycleur assez correct et d’un fournisseur d’oligonucléotides performant (rapide et pas cher) et évidemment de s’y connaitre quelque peu en PCR…
Si, par hasard, vous souhaiteriez plus de précisions, voici une vidéo des plus instructives, l’occasion pour nous de présenter l’excellent site LabTube.com dont une capture d’écran vous est proposée ci-dessus. Ce site internet héberge moult vidéos de conférences, diapositives illustrant une technique (dont l’ice COLD-PCR), bref une source d’informations techniques dont il ne faut pas se priver. Pour preuve au sujet de la recherche de mutations rares voici une présentation de Jorg Tost, directeur du laboratoire épigénétique et environnement (LEE), du Centre national de génotypage, Institut de Génomique/CEA.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages