 Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Ion PGM™ Template OT2 400 Kit) qui est principalement attendu pour les applications de séquençage de novo et de séquençage d’amplicons ciblant le 16S, 18S ou encore ITS2 (respectivement pour l’étude de la diversité bactérienne, eucaryote ou fongique) pour les études métagénomiques. Comme vous pouvez le constater cette application est uniquement supportée par le One Touch 2 devenu nécessaire pour accéder aux reads de 400 bases (c’est en partie pour cette raison que notre plateforme PEGASE-Biosciences a décidé de se doter de cette automate).
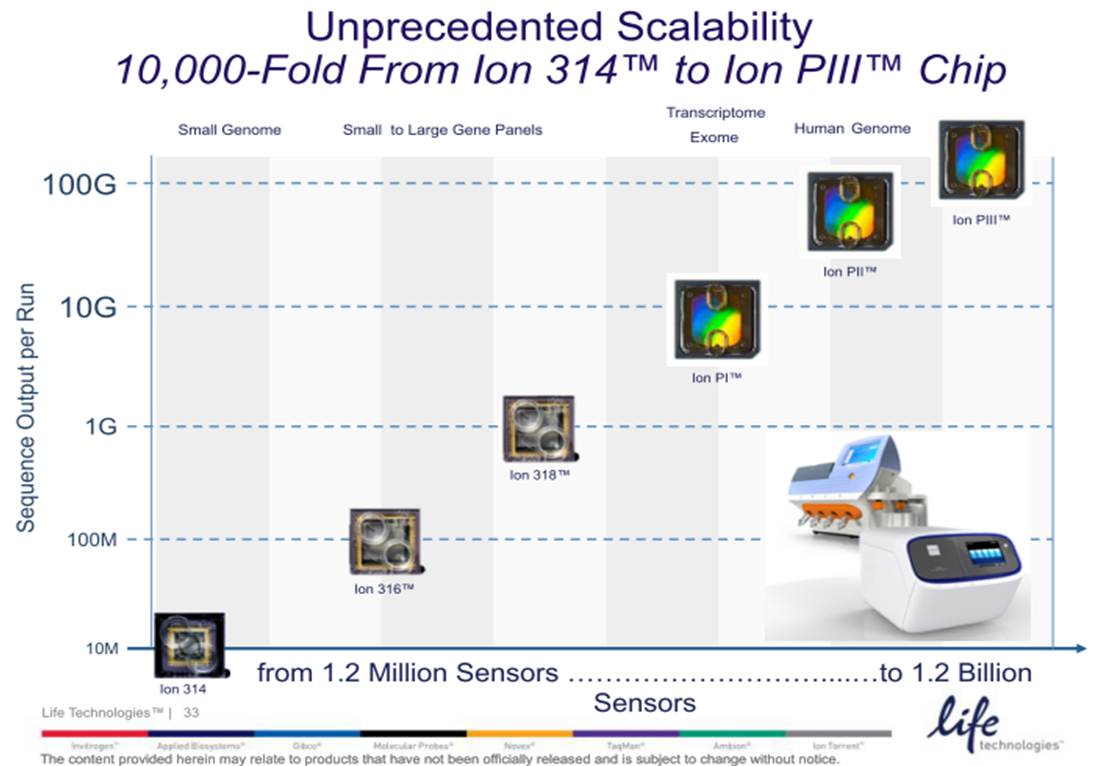
L’autre nouveauté est portée par les consommables de séquençage associés au Ion Proton (le grand frère du PGM). En effet, si la Puce Ion PIII est encore virtuelle, la PI et PII, permettent de délivrer : 10 Gb et 70 millions de reads filtrés environ pour la première, contre plus de 280 de millions de reads et plus de 70 Gb pour la seconde (de quoi séquencer un génome humain à 20 X).
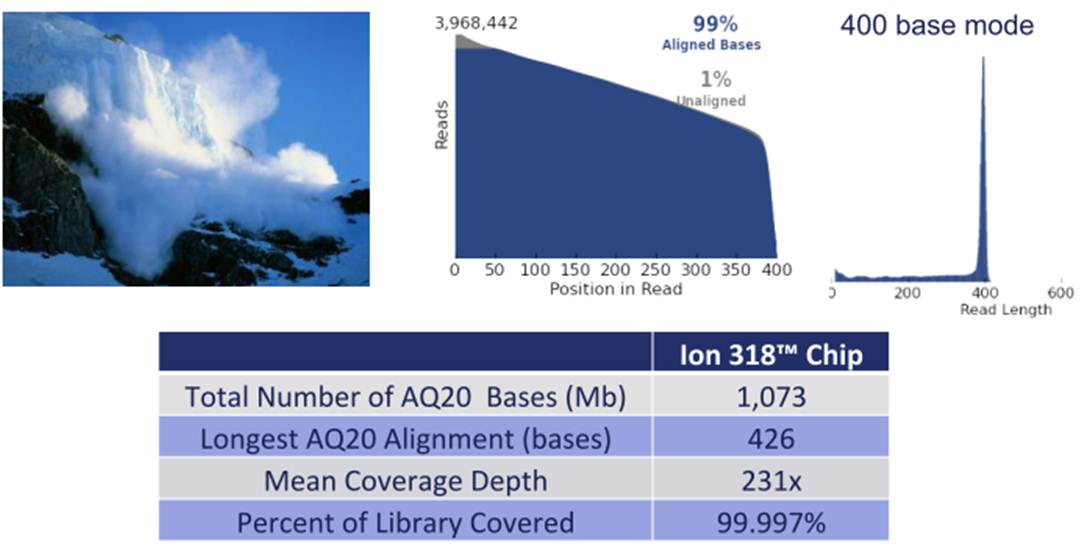
Du côté de Life Technologies et de sa technologie Ion Torrent, sans aucun doute, la principale nouveauté de l’année 2013 touche l’amplification clonale (phase où, à partir de la librairie préparée, est réalisée la matrice de séquençage fixée de manière covalente sur des billes qui se logeront dans les puits présents sur la puce). Ainsi, la fameuse (et pour certains la fâcheuse) phase de PCR en émulsion trouvera une alternative avec la chimie isothermique dite « avalanche » (dont vous pouvez voir l’aperçu d’un run ci-dessous). A en croire Life Technologies, cette chimie ouvre la voie à des tailles de séquences générées encore plus longues.
Bien entendu, Life Tech annonce l’arrivée d’un nouvel automate permettant d’intégrer la nouvelle chimie mais aussi de charger les puces (et pour quelques dizaines de k€, d’épargner au manipulateur les deux phases délicates du séquençage).
Pour conclure brièvement, l’une des nouveautés et non des moindres : l’acceptation du PGM comme diagnostic moléculaire CE-IVD (un parcours pas vraiment de tout repos) a n’en pas douter une conformité qui ne devrait pas laisser insensible les chercheurs, praticiens hospitaliers.
 Derrière le pseudonyme de Jack London (1876-1916) se cache un écrivain autodidacte, à la jeunesse vagabonde, qui a fait fortune en écrivant des livres avec une nature sauvage pour décor et personnage principal. L’écrivain, après avoir été balayeur de jardins publics, menuisier, agriculteur, éleveur de poulets, chasseur de phoques, pilleur d’huîtres, patrouilleur maritime, blanchisseur, chercheur d’or – seul Bukowski, depuis, a pratiqué plus de « jobs » différents – est passé à la postérité pour ses romans d’aventures, pour ses romans faisant la description d’une nature en voie de domestication et pour ses romans aux héros canidés : Croc Blanc, Le Fils du loup, Le loup des mers, Mikaël-chien de cirque et Jerry-chiens des îles.
Derrière le pseudonyme de Jack London (1876-1916) se cache un écrivain autodidacte, à la jeunesse vagabonde, qui a fait fortune en écrivant des livres avec une nature sauvage pour décor et personnage principal. L’écrivain, après avoir été balayeur de jardins publics, menuisier, agriculteur, éleveur de poulets, chasseur de phoques, pilleur d’huîtres, patrouilleur maritime, blanchisseur, chercheur d’or – seul Bukowski, depuis, a pratiqué plus de « jobs » différents – est passé à la postérité pour ses romans d’aventures, pour ses romans faisant la description d’une nature en voie de domestication et pour ses romans aux héros canidés : Croc Blanc, Le Fils du loup, Le loup des mers, Mikaël-chien de cirque et Jerry-chiens des îles.
La peste écarlate est une nouvelle d’anticipation (parue en 1912) assez méconnue. Elle ouvre sur une Terre dévastée et revenue à l’état sauvage suite à une pandémie qui a ravagé l’écrasante majorité de l’espèce humaine en 2013.
L’action se déroule 60 ans après cette apocalypse, le narrateur est un homme âgé en haillons qui a connu l’avant : la civilisation, la culture… et qui tâche de conter cet avant à des jeunes hommes redevenus préhistoriques (l’écriture, la lecture ont été oubliées, en une génération, seules diffusent les techniques et le savoir nécessaires à la survie).
Jack London réussit en une soixantaine de pages à poser les bases de ce qu’un nombre incroyables de récits post-apocalyptiques a pu produire comme usines à fantasmes et peurs millénaristes : de Ravage de Barjavel (1943) à la Route de Cormac Mc Carthy (2006), de MadMax de George Miller (1979) à 28 jours plus tard de Danny Boyle (2002). Jack London exploite bien évidemment la peur de la fin des temps humains. Cette fin du livre est provoquée par un micro-organisme qui engendre une mort aussi rapide que théâtralisée. Face au mal microbien les scientifiques sont inefficients et ne peuvent éviter l’apocalypse. Fort de ses thèmes de prédilection l’auteur qui aime les chiens, Marx et Nietzsche, propose une description de ce à quoi ressemblerait l’Homme sans civilisation, condamné à chercher sa nourriture… et à reconstruire ce qu’il a perdu. Entre Arche de Noé et Radeau de la Méduse, la vision de London parait moderne, certains diraient, un peu rapidement, prophétique parce que notre humanité a connu depuis ces écrits deux Guerres Mondiales, deux catastrophes nucléaires majeures, un tsunami et le VIH… Jack London ne traite pas réellement d’apocalypse mais du comportement d’une Humanité qui doit se rebâtir après un passage par la barbarie et d’une nouvelle lutte contre la nature. Finalement ce roman nous rappelle au cycle de la civilisation humaine : naissance – développement – apogée (les souvenirs de la jeunesse du narrateur de la peste écarlate) – chaos (le 2013 du livre, date de la pandémie) – ténèbres (le 2073, année où se déroule l’action du livre) – renaissance.
L’édition BABEL, outre une traduction remarquable, propose une postface de Michel Tournier (rédigée en 1992) qui est des plus remarquables (« l’Absolu est un voyage sans retour« ).
La nouvelle provient de l’Institut Italien de Technologie de Gènes : l’équipe du professeur Enzo di Fabricio (responsable du département de Nanostructures) a réussi à capturer l’image d’une molécule d’ADN à l’aide d’un microscope électronique en transmission et d’un support en silicone hydrophobe.
L’équipe a développé un support ( formé de colonnes dites « nanopiliers » ) extrêmement hydrophobe, provoquant ainsi l’évaporation, pour ne laisser visibles que des brins d’ADN tendus. L’équipe a également percé de petits puits dans le fond du support « nanopilier » (voir la photographie du support ci-dessous), à travers lequel ils font pénétrer des faisceaux d’électrons pour obtenir leur capture en haute résolution.
Cette image est générée en utilisant la technologie de microscopie TEM pour « transmission electron microscopy » qui repose sur le principe de transmission d’un faisceau d’électrons à travers un support très mince. Plus précisément une image est formée à partir des d’interactions entres les électrons et le support; le résultat peut ensuite être imprimé sur un film photographique ou capturé via un détecteur numérique. Cette technologie offre une grande résolution indispensable pour visualiser un brin d’adn.
Sur l’image en en-tête on repère bien la forme hélicoïdale de la double-hélice d’ADN.
Outre l’aspect « inédit » de cette technique, elle ouvre également des perspectives intéressantes: on pourrait être ainsi en mesure d’observer (réellement) la façon dont une molécule d’ADN interagit avec diverses biomolécules.
Pour en savoir plus : l’article « Direct Imaging of DNA Fibers: The Visage of Double Helix » publié dans Nanoletters (American Chemical Society) en Novembre 2012
Lille, le 19 décembre 2012
 Le PPF Bioinformatique de l’Université Lille 1 organise une journée scientifique sur le thème de la métagénomique et des méthodes d’analyse bio-informatique et biostatistique associées : assemblage des génomes, classification et quantification des espèces, assignation taxonomique, reconstruction phylogénétique… Cette réunion s’inscrit dans la lignée des éditions 2009 et 2011 sur l’analyse des données NGS.
Le PPF Bioinformatique de l’Université Lille 1 organise une journée scientifique sur le thème de la métagénomique et des méthodes d’analyse bio-informatique et biostatistique associées : assemblage des génomes, classification et quantification des espèces, assignation taxonomique, reconstruction phylogénétique… Cette réunion s’inscrit dans la lignée des éditions 2009 et 2011 sur l’analyse des données NGS.
Les exposés se tiendront dans l’amphithéatre Butiaux, sur le campus de l’Institut Pasteur de Lille (métro ligne 2, station Grand Palais).
Programme
- 10h00 : café d’accueil
- 10h20 : présentation de la journée (David Hot)
- 10h30-11h30 : Nicolas Pons, INRA Jouy-en-Josas : The potential of the new science of metagenomics in curing human complex diseases
- 11h30-12h20 : Guy Perrière, LBBE, Lyon
- 12h20-13h45 : déjeuner
- 13h45-14h35 : Frédéric Mahé, TU Kaiserslautern : TARA OCEANS, exploring worldwide marine biodiversity
- 14h35-15h25 : Mihai Pop, University of Maryland : Towards Holistic Analyses of Metagenomic Data
- 15h25-15h40 : pause
- 15h40-16h30: Jean-Jacques Daudin, AgroParisTech : Bioinformatics and Statistical challenges from the analysis of NGS-Metagenomics experiments
- 16h30-17h20 : Sara Vieira-Silva, Vlaams Instituut voor Biotechnologie
Inscriptions
La participation est gratuite, mais nous vous demandons de vous inscrire pour permettre l’organisation des pauses et du déjeuner.
Merci d’envoyer un mail à mohcen.benmounah@lifl.fr avec les informations suivantes pour le 7 décembre.
--------------------------------------------------
19 decembre 2012 - journee Metagenomique
Nom:
Prenom:
Laboratoire :
Dejeuner : oui/non
--------------------------------------------------
Vous retrouverez l’intégralité de ces informations sur le site du ppf : http://www.lifl.fr/~touzet/PPF/metagenomique12.html
Le réseau régional d’ingénieurs en bioinformatique de Lille et le PPF bioinformatique vous convient à une conférence :
Mercredi 28 novembre 2012, 10h30-12h, Amphithéâtre de l’Institut de Biologie de Lille, 1, rue du Pr Calmette, LILLE.
Comment diversifier les données de séquençage à haut débit pour optimiser la bioinformatique ?

Les stratégies hybrides PacBio & Roche 454/Illumina HiSeq
– Dr Christophe Meynier et Sabrina Benoussaidh – GATC Biotech France & Belgique.
« Les nouvelles technologies de séquençage (NGS) ont permis d’améliorer drastiquement la caractérisation des génomes avec la génération de séquence en haut débit et ce à faible coût. Malgré cela, l’assemblage d’un génome reste souvent problématique et diffère en fonction des technologies utilisées. En effet la qualité d’un assemblage de novo est influencée par différents facteurs inhérent à la structure intrinsèque du génome et en parallèle à la longueur des fragments, au nombre de séquences et à la qualité de ces dernières. La technologie PacBio RS fournit les plus longues séquences jamais obtenues (3500 bases en moyenne) ce qui simplifie et améliore la création de contigs.
Des études ont montré qu’en corrigeant les séquences générées par le PacBio RS avec des données issues de secondes générations de séquenceurs (Roche 454, HiSeq), nous optimisons alors les avantages de chacune de ces technologies (couverture, fiabilité des bases et longueurs des séquences). En diminuant le nombre de contigs tout en augmentant leurs tailles, les chercheurs ont désormais les outils pour obtenir des assemblages de génomes plus rapidement, à moindre coûts ou pour obtenir des informations sur des génomes partiellement séquencés jusque-là disponibles uniquement manuellement (via PCR ou marche sur l’ADN). Notre présentation montre des résultats d’assemblages obtenus avec différentes technologies, les compare et démontre l’intérêt d’association de technologies ensemble. En outre, nous discuterons également des technologies Illumina HiSeq 2000 et Roche 454 au travers d’applications spécifiques telles que le reséquençage de génomes entiers, le séquençage de transcriptomes (RNAseq) et le ChipSeq. »
 DNA-Vision est devenue en 2011, le prestataire européen en génomique avec la plus grande puissance de séquençage (quatre systèmes Solid, deux HiSeq2000, un GAIIx, tous deux d’Illumina, et deux 454-de chez Roche). La petite société issue de l’Université Libre de Bruxelles (ULB) et fondée en 2004 a bien grandi et est actuellement dirigée par Laurent Alexandre. Ce dernier est le créateur du site e-toubib, Doctissimo, aujourd’hui détenteur de 80 % de DNA-Vision, suite à la vente de son site internet à Lagardère qui a permis à l’ex-chirurgien de devenir riche… engrangeant plus de 139 millions d’euros suite à la transaction. Il faut dire que le docteur en médecine, chirurgien urologue, énarque et diplômé en sciences politiques a été à l’origine de plusieurs sociétés dans le domaine médical : Benefit, Medcost, Clinics et enfin le site préféré des hypocondriaques, Doctissimo. C’est avec pour idée principale d’appliquer le séquençage, en profitant de sa vertigineuse baisse de coûts, au champ de la médecine personnalisée que le médecin essayiste, auteur de « la mort de la mort » est devenu président de la société DNAvision. Même s’il est vrai que, l’encore assez nouveau président de DNAVision, préparant l’entrée en bourse de son entreprise, parle de médecine personnalisée, la société belge est connue pour sa maîtrise des technologies haut-débit et à titre d’exemple, pour son expertise du séquençage appliqué aux études métagénomiques (vous pouvez lire : Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa, PNAS, avril 2010).
DNA-Vision est devenue en 2011, le prestataire européen en génomique avec la plus grande puissance de séquençage (quatre systèmes Solid, deux HiSeq2000, un GAIIx, tous deux d’Illumina, et deux 454-de chez Roche). La petite société issue de l’Université Libre de Bruxelles (ULB) et fondée en 2004 a bien grandi et est actuellement dirigée par Laurent Alexandre. Ce dernier est le créateur du site e-toubib, Doctissimo, aujourd’hui détenteur de 80 % de DNA-Vision, suite à la vente de son site internet à Lagardère qui a permis à l’ex-chirurgien de devenir riche… engrangeant plus de 139 millions d’euros suite à la transaction. Il faut dire que le docteur en médecine, chirurgien urologue, énarque et diplômé en sciences politiques a été à l’origine de plusieurs sociétés dans le domaine médical : Benefit, Medcost, Clinics et enfin le site préféré des hypocondriaques, Doctissimo. C’est avec pour idée principale d’appliquer le séquençage, en profitant de sa vertigineuse baisse de coûts, au champ de la médecine personnalisée que le médecin essayiste, auteur de « la mort de la mort » est devenu président de la société DNAvision. Même s’il est vrai que, l’encore assez nouveau président de DNAVision, préparant l’entrée en bourse de son entreprise, parle de médecine personnalisée, la société belge est connue pour sa maîtrise des technologies haut-débit et à titre d’exemple, pour son expertise du séquençage appliqué aux études métagénomiques (vous pouvez lire : Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa, PNAS, avril 2010).
Revenons au moins un instant sur le séquençage haut-débit appliqué à la médecine personnalisée, vu comme le fait de rendre accessible au soignant l’information génomique de son patient.
A une époque où le Dossier Médical Unique (DMP) a grand mal à devenir une évidence, à une période où l’accès universel aux soins est de plus en plus remis en question pour des raisons de disette financière, l’accès à une archive de plusieurs Gigaoctets de données par patient semble peu crédible à court terme. Ajoutés à cela les problèmes d’éthique, de confidentialité de données et de formation du personnel soignant à l’interprétation de ces données et vous comprendrez que le rêve d’un Laurent Alexandre aura du mal à toucher la médecine de masse mais pourra séduire les cliniques privées qui échappent à la problématique d’un accès universalisé aux soins.
On voit qu’à l’instar du tourisme spatial, la cible ne semble pas vraiment être le chômeur en fin de droit. La médecine pour pas grand monde… puisque si un séquençage coûte de moins en moins cher, l’interprétation des données générées et l’intégration de ces données dans un réel parcours médical est encore inaccessible aux 99 % des personnes les plus pauvres des 10 % des pays les plus riches.
Malgré tout, la médecine personnalisée dépasse cette vision de l’ADN. En effet, la médecine personnalisée comporte plusieurs approches :
– la détermination du risque (un génotypage haut-débit chez 23andme ou un séquençage chez un autre quidam pour vous dire que vous avez 2,03 % de chance de plus que la moyenne d’être atteinte de la maladie d’Alzheimer)
– la médecine stratifiée où seuls les patients répondant positivement à l’administration d’une solution curative, minimisant les effets secondaires de celle-ci, seront traités.
– la thérapie génique (un peu passée de mode) pour laquelle il existe trois types de thérapies : la transfection permanente d’ADN, la transfection transitoire d’ADN, les thérapies portant sur l’ARN.
 Si le séquençage multiparallélisé de l’ADN a tendance à se démocratiser au sein des laboratoires de recherche et de certains laboratoires en usant comme diagnostic, il semble qu’un pas de géant reste à franchir pour que celui-ci transforme complètement notre vision de la médecine. La médecine personnalisée n’est cependant pas à remettre en question… il s’agit juste de lui donner un horizon réaliste et réalisable, sans quoi cette notion sera à ranger, comme beaucoup d’autres avant, aux rayons des mots clés usés par la mode.
Si le séquençage multiparallélisé de l’ADN a tendance à se démocratiser au sein des laboratoires de recherche et de certains laboratoires en usant comme diagnostic, il semble qu’un pas de géant reste à franchir pour que celui-ci transforme complètement notre vision de la médecine. La médecine personnalisée n’est cependant pas à remettre en question… il s’agit juste de lui donner un horizon réaliste et réalisable, sans quoi cette notion sera à ranger, comme beaucoup d’autres avant, aux rayons des mots clés usés par la mode.
 « La réalité virtuelle » ou « la réalité augmentée », « l’art génératif » ou encore « l’art interactif », autant de déclinaisons possibles pour designer « l’art virtuel« . Cette forme d’art nouveau est à la croisée des chemins de l’informatique, de la technologie ou encore du graphisme.
« La réalité virtuelle » ou « la réalité augmentée », « l’art génératif » ou encore « l’art interactif », autant de déclinaisons possibles pour designer « l’art virtuel« . Cette forme d’art nouveau est à la croisée des chemins de l’informatique, de la technologie ou encore du graphisme.
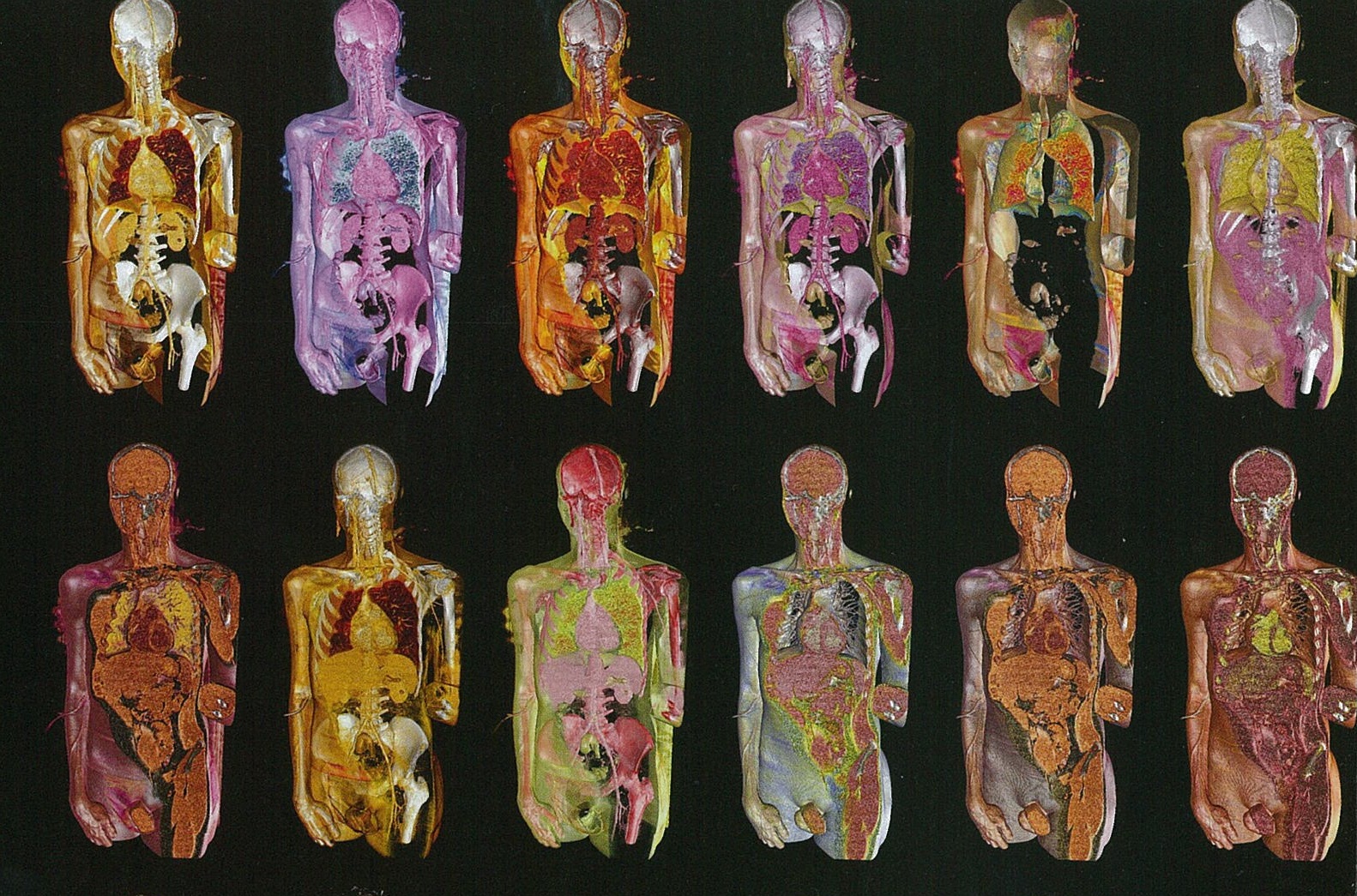
Parmi les plus médiatiques de la discipline, à la fois radiologue et photographe, Rodolphe von Gombergh détourne les techniques de l’imagerie médicale de pointe (ultra-sons, ondes électromagnétiques, rayons X et imagerie 3D) pour créer son propre univers et révéler ses secrets dans l’art de la trans-apparence ( Une des modalités du Bio Art évoqué au cours d’un article précédent).
Dès les prémisses de l’imagerie médicale, il se passionne pour la discipline et, fort d’une équipe de recherche et développement, contribue à l’explosion de l’imagerie médicale moderne. les procédés utilisés permettent de modéliser le corps humain en faisant apparaître les organes comme transparents. Cet explorateur des corps eut l’occasion d’exposer ses premières images médicales au musée Beaubourg. Dix ans plus tard, c’est au musée Guimet que seront livrées les découvertes des scanner des célèbres bouddhas du XI au XVII ème siécle (http://www.dailymotion.com/video/xqtdo2_virtual-art-ou-la-science-qui-se-mele-a-l-art_news).
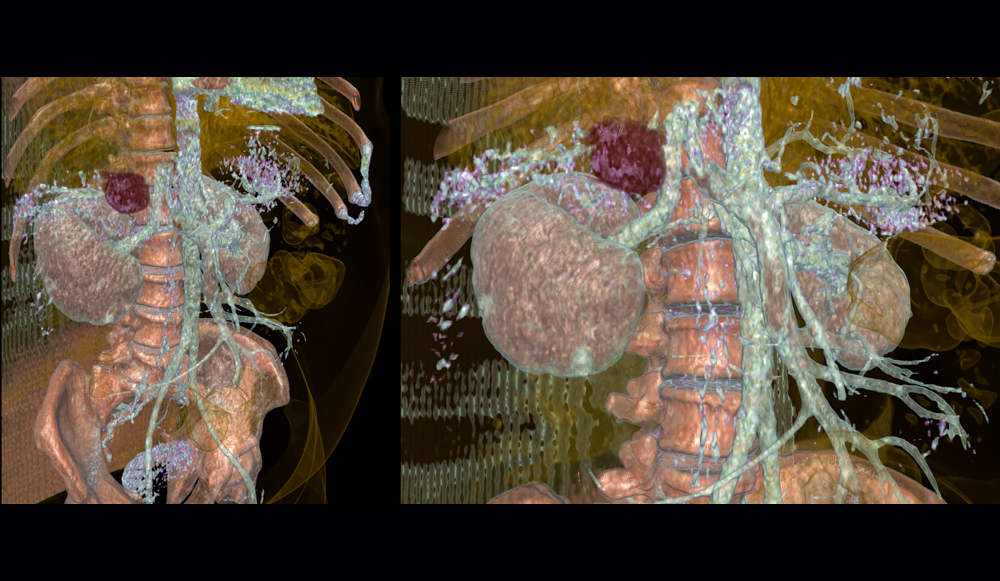
Sur le plan médical, il est aisé d’imaginer la puissance de ces méthodes non invasives pour le dépistage d’anomalies (exemple ci dessous présentant une tumeur de la surrénale), d’autant qu’elles ne nécessitent aucune anesthésie ni ne présentent de risque d’infection pour le patient…
 La paléogénétique, la science qui permet de remonter le temps, a trouvé sous la forme de séquenceurs haut-débit, sa DeLorean… en route vers l’Ardèche, dans l’antre de la grotte Chauvet. La grotte Chauvet, inventée en 1994, comporte 420 représentations d’animaux d’une incroyable diversité technique. C’est en partie les déductions menées à partir de l’étude de cette grotte, que l’on a remis en cause l’idée d’un art (pariétal) évoluant lentement et de manière linéaire. L’art peut être vu comme une suite d’apogées et de déclins, un concept s’opposant à l’idée d’un art co-évoluant avec le progrès technique et la diffusion du savoir-faire et des connaissances.
La paléogénétique, la science qui permet de remonter le temps, a trouvé sous la forme de séquenceurs haut-débit, sa DeLorean… en route vers l’Ardèche, dans l’antre de la grotte Chauvet. La grotte Chauvet, inventée en 1994, comporte 420 représentations d’animaux d’une incroyable diversité technique. C’est en partie les déductions menées à partir de l’étude de cette grotte, que l’on a remis en cause l’idée d’un art (pariétal) évoluant lentement et de manière linéaire. L’art peut être vu comme une suite d’apogées et de déclins, un concept s’opposant à l’idée d’un art co-évoluant avec le progrès technique et la diffusion du savoir-faire et des connaissances.
Les œuvres de la grotte Chauvet démontrent q’au début du Paléolithique supérieur, des artistes étaient capables d’une abstraction intellectuelle (une certaine conceptualisation) pour préparer la paroi calcaire et penser le dessin, tout en maitrisant des techniques complexes (estompes,perspective…). L’observation des traces humaines, à la grotte Chauvet comme dans beaucoup de lieux montrant une activité préhistorique, est accompagnée d’analyses de mesures physico-chimiques. Ainsi, avec d’autres méthodes, la datation radiométrique au carbone 14 a été employée pour permettre une estimation de l’âge absolu d’un échantillon organique… la grotte ardéchoise aurait hébergé les artistes du paléolithique autour de 31 000 ans avant notre ère. Assez nouvellement les techniques de génomique trouvent un champ d’application au niveau de la l’histoire des populations et viennent compléter les autres types d’observations pour nous reconstituer ce que le temps a détruit ou transformé.
Le carbone 14 génomique pourrait (en quelque sorte) être constitué par l’ADN mitochondrial. L’ADNmt est ici pris comme témoin (plus ou moins stable) des espèces disparues et sert de base aux études phylogénétiques, loin de Jurassic Park mais néanmoins enthousiasmant… mais est ce vraiment aussi simple ?
En effet, de par sa stabilité présumée au fil de l’évolution, l’ADNmt est devenu une cible de choix pour l’étude de la diversité des populations : a priori, les gènes déterminant la fonction respiratoire de la cellule sont moins la cible d’adaptations fréquentes à des changements environnementaux que, par exemple, ceux de l’immunité, situés, eux, dans l’ADN nucléaire. Je vous conseille vivement, à ce sujet, la lecture de l’article suivant, remettant en cause ce paradigme (cf. CNRS – le journal – mars 2007).
Mais revenons à l’utilisation du séquençage haut-débit pour remonter le temps. Le temps a fait son œuvre quant à la première étape de la préparation de la librairie, la fragmentation. « Dans les régions tempérées, ce sont les échantillons provenant de grottes qui sont souvent les mieux conservés (cf. figure ci-dessous). Dans les grottes, la température reste très stable, autour d’une valeur comprise entre 10 et 15°C. De plus, les grottes se creusent dans des environnements karstiques, riches en calcium et de pH neutre ou basique, qui sont des conditions propices à la conservation de la matière organique et de l’ADN en particulier. » – Extrait de la thèse de Céline Bon (CEA – page 50 – 25 juin 2012).
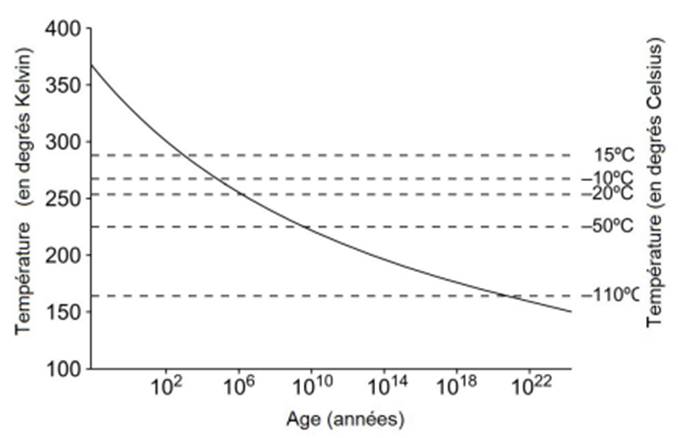
Temps de survie d’un ADN de taille supérieure à 100 pb
Le séquençage haut-débit devient réellement d’un grand intérêt dans l’étude du métagénome des coprolithes et des ossements retrouvés dans ces grottes. La capture d’écran ci-dessous vous mènera en cliquant dessus, sur une partie du site du CEA où une vidéo simple et efficace, reprend les propos de Jean-Marc Elalouf. Ce dernier aborde l’exceptionnelle conservation des ossements qui a permis aux biologistes d’en analyser l’ADN et ainsi de faire de nouvelles avancées sur l’étude des espèces éteintes et la phylogénie des ours des cavernes.
Un rapide, très rapide post pour vous faire part de la mise à disposition des diapositives (au format pdf) projetées lors des présentations du séminaire : 1 an d’expériences sur le désormais célèbre séquenceur de paillasse le PGM de Ion Torrent (Life Tech).
Les diapositives sont disponibles en suivant le lien http://www.pegase-biosciences.com/?p=506
Jeudi 13 Septembre 2012 de 9h00 à 12h30 dans l’amphi Buttiaux de l’Institut Pasteur de Lille
Après plus d’un an d’utilisation du séquenceur Ion PGM ™ , la plateforme PEGASE-Biosciences organise une demi-journée de retour d’expérience afin de dresser un bilan des possibilités offertes par cette technologie et d’échanger sur les applications du séquençage à haut-débit.
Vous pouvez télécharger le flyer en suivant ce lien.
Agenda :
9h00 – 9h10 Mot d’accueil – Introduction.
9h10 – 9h30 Stéphane Jankowski (Life Technologies) : Technologie Ion Torrent ™
9h30 – 10h00 Valérie Leclère (Laboratoire ProBioGEM, Univ Lille1) : « Séquençage de génomes entiers pour caractériser de nouvelles souches de Bacillus ».
10h00 – 10h30 Magali Chabé (BDPEE, CIIL – Institut Pasteur de Lille) : « Etude du transcriptome de deux espèces de Pneumocystis par RNA-sequencing de novo ».
10h30 – 10h50 Pause café.
10h50 – 11h20 Christophe Audebert (Gènes Diffusion) & Ségolène Caboche (IFR142, Lille2) : « Le séquençage haut-débit appliqué à la caractérisation d’un agent infectieux : exemple du séquençage d’Escherichia coli à l’origine d’une infection alimentaire ».
11h20 – 11h50 Charles Hébert (Pathoquest) : « Détection et identification de micro-organismes dans le plasma humain par approche métagénomique ».
11h50 – 12h20 David Hot (TAG, CIIL – Institut Pasteur de Lille) : « Identification de nouveaux transcrits (small RNAs) et étude des départs de transcription chez Bordetella pertussis par ‘differential RNA-seq’ ».
12h20 – 12h30 Conclusion – modalités du concours PEGASE.
Pour célébrer la création de “PEGASE-Biosciences” entre le laboratoire de Transcriptomique et Génomique Appliquée de l’IPL et la société Gènes Diffusion, la plateforme organise un concours* vous permettant de gagner un séquençage Ion Torrent ™ sur puce 318 (~1Gb de séquences).
Les places étant limitées, nous vous remercions de vous inscrire par email à l’adresse suivante :
stephane.jankowski@lifetech.com ou admin@pegase-biosciences.com
*Les modalités de ce concours vous seront présentées lors de cette journée.
Lien vers l’annonce du séminaire sur : http://www.pegase-biosciences.com/?p=464
Vous pouvez confirmer votre participation en scannant le QR Code suivant :
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages







