Ce post fait naturellement suite à celui dédié à la seconde génération de séquenceurs multi-parallélisés, et conserve la même approche, à savoir un tour d’horizon des technologies et une évocations des informations générales sur le sujet.
A l’instar du PGM de Ion torrent mis sur le marché depuis un an (10Mb – reads 100b – 06.2011 / 100Mb – reads 200b -11.2011 / 1Gb – reads 400b – prévu début 2012), la seconde génération de séquenceurs haut débit tend vers une production de reads de plus en plus longs et de moins en moins chère. Toutefois, on est en droit de se demander quelle sera leur pérennité face à la 3éme génération répondant à un cahier des charges assez similaire et la possibilité de bénéficier de nouvelles applications.
Le principe de la 3ème génération peut être symbolisé par le séquençage d’une molécule d’ADN sans étape de pré-amplification (contrairement à la génération actuelle type 454 Roche, SOLiD Life technologie, Ion Proton, PGM Ion torrent, HiSeq Illumina, …) en conservant l’incorporation de nucléotides, par cycles ou non ( dans ce dernier cas, le terme de « Séquençage d’ADN simple molécule en temps réel » est approprié).
Les technologies « SMS » pour « Single Molecule Sequencing » peuvent être regroupées selon trois catégories:
– Technologies de séquençage en temps réel impliquant la synthèse du brin d’ADN complémentaire via une ADN polymérase.
– Technologies de séquençage par détection des bases successives d’une molécule d’ADN au travers de nanopores.
– Technologies de séquençage basées sur des techniques de microscopie.
En combinant les dernières avancées dans la nanofabrication, la chimie de surface et l’optique, Pacific Biosciences (Pacbio RS) a lancé une plateforme technologique puissante appelée technologie de molécule unique en temps réel, ou « SMRT » pour « Single Molecule Real-time sequencing ». Parmi ses concurrents directs, Helicos Biosciences (Helicos) qualifié « tSMS » pour « True Single Molecule Sequencing ». Malgré le recours à une technologie analogue, la mention « Temps réel » auquel il échappe est simplement liée à une incorporation cyclique des nucléotides fluorescents.
D’autres technologies, à des degrés de développement plus ou moins avancé, sont dans les tuyaux et qui sait de Noblegen, Starlight, Cracker Bio, NABSys, Halcyon, ou autres… révolutionnera encore un peu plus cet univers du haut débit et suivra le chemin emprunté dernièrement par Oxford Nanopore …
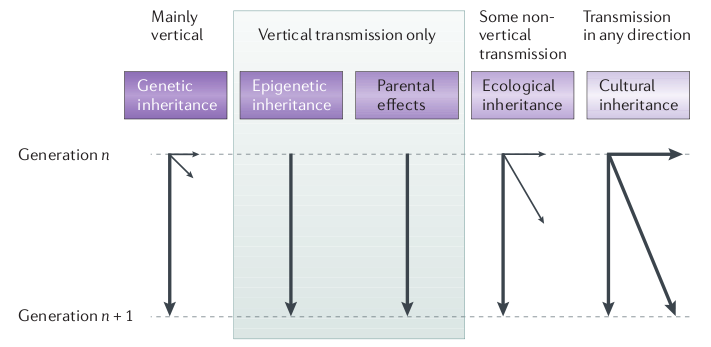
Pierre Barthélémy, journaliste scientifique au monde, a publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS et directeur de l’EDB) qui nous parle de la nécessité de s’extraire du dogme du « tout-génétique » pour s’intéresser plus précisément à d’autres mécanismes qui régissent l’hérédité, tel que l’épigénétique, l’hérédité culturelle et environnementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cette interview fait suite à la publication d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution parue dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’interroger sur la part réelle de la génétique dans les mécanismes de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post) qui aborde ces aspects de la transmission génétique et non génétique, entre générations.
L’hérédité culturelle est une composante en général peu discutée (notamment dans le domaine animal): quelle est la part de variation phénotypique qui peut être expliquée par une transmission sociale (ou transmission par apprentissage)? A noter que le rôle de la composante culturelle dans l’hérédité a été formalisé dans les années 1970, ça ne date donc pas d’hier, mais comme toutes les composantes non génétiques impliquées dans l’hérédité elle s’est retrouvée minimisée, voire ignorée, au profit de la composante génétique.
Quoi qu’il en soit, à l’heure de l’arrivée de la 3eme génération de séquenceur haut-débit et de la génération toujours plus astronomique de données génomiques, il est toujours bon de rappeler que le seul transfert de gènes n’explique qu’une proportion des mécanismes étudiés.
Pierre Barthélémy, journaliste scientifique à publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS) qui nous parle de la nécéssité de sortir du dogme du « tout-génétique » pour s’intérésser plus précisement à d’autre mécanisme qui régisse l’hériédité, l’épigénétique, l’hérédité génétique et environementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cet interview est le résultats d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution publié dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’intéroger sur la place réelle de la génétique dans les mécanisme de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post).
 Le représentant européen des séquenceurs haut-débit de troisième génération, Oxford Nanopore va communiquer à l’AGBT… dans quelques jours. La société en profitera pour annoncer la commercialisation de ses premières machines permettant de séquencer en 24 heures un génome humain pour moins de 1000 $ (soit le même prix qu’un génome humain séquencé par un Ion Proton en 2 heures sur une plateforme de deuxième génération).
Le représentant européen des séquenceurs haut-débit de troisième génération, Oxford Nanopore va communiquer à l’AGBT… dans quelques jours. La société en profitera pour annoncer la commercialisation de ses premières machines permettant de séquencer en 24 heures un génome humain pour moins de 1000 $ (soit le même prix qu’un génome humain séquencé par un Ion Proton en 2 heures sur une plateforme de deuxième génération).
Oxford Nanopore souhaite commercialiser sa machine (en direct) avant la fin 2012. Un réelle course a lieu, puisqu’à cette échéance sera commercialisé le Ion Proton. Nous assisterons donc avant fin 2012 a un chevauchement technologique où 2ème et 3ème générations de séquenceurs risquent de se télescoper… les scientifiques en cours d’élaboration de dossiers de demandes de financements devront (vite) choisir leur camp… : partir sur une technologie qui a d’ores-et-déjà fait ses preuves (Ion Proton) donc de 2ème génération ou une technologie de 3ème génération totalement innovante… mais dont on ne possède pas de retour indépendant sur la qualité des données générées. La « guerre » technologique est lancée entre le transfuge de Solexa (technologie des séquenceurs Illumina), John Milton, le responsable scientifique d’Oxford Nanopore et la biotech’ rock-star Jonathan Rothberg, responsable scientifique de Ion Torrent chez Life Technologies.
A Davos en Suisse (dans la patrie de Roche) au forum économique mondial, Jonathan Rothberg présente son séquenceur Ion Proton, une évolution de son PGM de Life Technologies. La dépêche AFP (mise en forme par 20minutes est disponible en cliquant sur l’image ci-dessous) est dithyrambique replaçant la machine de Life Technologies dans le contexte et le champ d’application de la médecine personnalisée… jouant au passage du violon de l’affect. Le Ion Proton, fort de la démonstration menée par son petit frère le PGM depuis un an (200 machines PGM en Europe !) s’appuie sur l’efficience du séquençage à l’aide de semi-conducteurs.
Il parait évident que la médecine personnalisée aura un futur au sein des pays développés… mais il ne faut pas oublier qu’il s’agit d’un futur que l’on augurait aux puces à ADN, il y a plus de 10 ans, sans réelle démocratisation au sein des cliniques. Avant toute chose, permettre de séquencer l’équivalent d’un génome humain ou de 2 exomes humains pour la somme soutenable de 1000 $ (en 2 heures) laisse entrevoir une accélération des recherches biomédicales mais aussi de toutes les autres ! Cet outil qui torpille un MiSeq (Illumina) sorti trop tard, diffusera dans les laboratoires… il remporte, malgré ses défauts d’ores et déjà connus, la bataille du séquençage 2ème génération. Le prochain pari (plus aléatoire) porterait plutôt sur le séquençage 3ème génération dont la promesse est une lecture d’une molécule d’ADN ou d’ARN (non amplifiée artificiellement) dans son « état » originel (méthylé ou non par exemple). La 3ème génération unifie génomique et épigénétique.
Le Ion Proton devrait être livrable à partir d’octobre 2012, quelques chanceux pourront malgré tout réaliser des runs dès le milieu de l’année (il faut en acheter un minimum de 4 pour être un parmi ces privilégiés).
Un Ion Proton devrait avoir un coût proche de 140 k€ auxquels il vous faudra ajouter 65 k€ de serveur informatique.
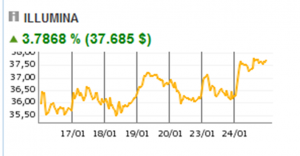
La société suisse Roche tente de phagocyter la société californienne mastodonte, Illumina. Roche est prête à sortir un minimum de 5,7 milliards de USD de ses coffres suisses pour lancer une OPA inamicale – offre lancée le 24 janvier 2012 – sur la société leader de la biologie haut-débit. Illumina a été approchée par la société Roche afin de trouver un « terrain » d’entente, sans succès. Roche propose une offre valorisant l’action de 43 % par rapport à la valeur des cours des 3 derniers mois.
« Ensemble, les capacités de la division Diagnostics de Roche et d’Illumina, l’un des principaux fournisseurs de systèmes intégrés pour le séquençage de l’ADN, renforceront la position de Roche dans les secteurs Séquençage et Puces à ADN caractérisés par une demande croissante de solutions génétiques/génomiques », explique Roche, qui souligne qu’il « n’a pas ménagé ses efforts en vue de parvenir à une transaction négociée avec Illumina », mais que celle-ci « n’a pas voulu s’engager dans une discussion de fond ».
En temps de crise, les actionnaires ont bien souvent besoin de liquidité – valorisée qui plus est – il ne fait nul doute que cette OPA arrivera à ses fins… il ne s’agit pas nécessairement pour les scientifiques d’une excellente nouvelle, tant la concentration aux mains de peu se ressent par le moins de développement et des coûts croissants à supporter au niveau des laboratoires… Nous assistons à la création, au niveau des sociétés de biotechnologie, d’un monde bipolaire : Life vs Roche !
Le communiqué de presse de Roche concernant cette OPA est disponible ici.
 Chaque année depuis 13 ans, l’AGBT (Advances in Genome Biology and Technology) rassemble les fournisseurs de technologies de pointe, quelques « maîtres » en la matière venant dispenser des cours du soir (sorte de « master-class ») et beaucoup de scientifiques essentiellement Nord-Américains. En effet, cette grande messe de la biologie haut-débit a lieu à Marco Island, île de Floride, lieu de villégiature pour retraités liftés percevant leurs fonds de pension sous forme de rente viagère.
Chaque année depuis 13 ans, l’AGBT (Advances in Genome Biology and Technology) rassemble les fournisseurs de technologies de pointe, quelques « maîtres » en la matière venant dispenser des cours du soir (sorte de « master-class ») et beaucoup de scientifiques essentiellement Nord-Américains. En effet, cette grande messe de la biologie haut-débit a lieu à Marco Island, île de Floride, lieu de villégiature pour retraités liftés percevant leurs fonds de pension sous forme de rente viagère.
La Côte Est américaine accueille les grandes entreprises de biotechnologies de la côte opposée pour démontrer que la Californie avec des sociétés telles que Illumina, Life technologies, Roche, Pacific Biosciences, Agilent… reste le meilleur fournisseur de technologies de pointes pour les gros centres de recherches situés sur la Côté Est (NIH, MIT-Broad Institute, Yale…). La Côte Est héberge les deux tiers des séquenceurs haut débit nord-américains. Depuis quatre ans, il faut bien le dire l’AGBT consacre le séquençage haut-débit, technologie qui vampirise la plupart des présentations (n’oublions pas que la jeune société PacBio sponsor principal, vend des séquenceurs…) et sessions « workshop »… au détriment de technologies plus « anciennes » (les puces à ADN par exemple). On le sait, ce type de rassemblements adoube la nouveauté et consacre ce que, parfois, hâtivement, on nomme : innovation.
Bien que ces rassemblements endogames sont critiquables, ils constituent une tribune pour les principaux axes de développements des technologies appliquées au vivant. L’AGBT est un tremplin dont se servent un grand nombre de firmes californiennes pour annoncer des lancements de machines à générer de la séquence. Cet évènement très américanocentré n’a pas de réel pendant européen, malgré les différentes formes prises par des rassemblements tels que le « carrefour des biotechnologies » remplacé par l’Eurobio-event. L’Eurobio-event sorte de biennale biotech’ où l’on trouve rassemblés beaucoup de vendeurs de robotiques, de pipettes… beaucoup de conférences traitant du business, de l’investissement dans les biotechnologies plus que des biotechnologies en tant que technologies. Assez peu de science dans Eurobio-event, beaucoup de business.
L’AGBT oscille quant à elle, entre un monde de scientifiques avide de nouvelles méthodes visant à l’exploitation rationnelle des flots de données issues de la biologie haut-débit (ce que représente l’illustration ci-dessus) et un monde de vendeurs de solutions appliquées à la recherche en biologie – quand les uns partagent un même « problème » et réfléchissent à une solution optimale pour sortir du sens biologique d’un amas de données, les autres vendent les générateurs de « problèmes » en misant sur la communauté scientifique mondiale pour apporter la réponse aux nouveaux problèmes générés. Il s’agit de trier l’information émanant de ce type de rassemblements en n’oubliant pas que cette vitrine – telle que peut l’être l’AGBT ou tout autre évènement de cette ampleur – peut parfois être un mirage scientifique plus qu’un virage technologique…
L’AGBT cette année 2012 aura lieu du 15 au 18 février sur l’île Marco Island : http://agbt.org/about.html, on y parlera de la technologie de Pacific Biosciences (des retours sur expériences sont prévus, avec leur 15 % de taux d’erreur de séquençage, il s’agit de LA déception technologique de l’année, mais PacBio est aussi LE sponsor principal de la réunion, quel suspens !), on y parlera aussi du futur Ion Proton…
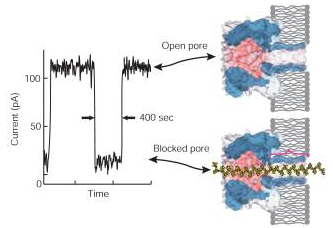
 Depuis la publication de 2006, d’Astier and al., J. Am. Chem. Soc., la communauté scientifique attendait la révolution du séquençage 3ème génération que constituait la géniale idée d’Oxford Nanopore : à savoir l’alliance des nanotechnologies, du génie génétique le tout à des fins de séquençage (et plus si affinité). Ainsi, un pore d’alpha-hémolysine (d’environ 1 nanomètre) ou la porine A de Mycobacterium smegmatis ou bien encore du graphène ont été investigués pour devenir le pore idéal dans lequel, après clivage par une exonucléase des liaisons phosphodiester, les bases azotées dont l’enchaînement constitue le séquençage passe afin d’être analysées (par analysées on entend lues, la lecture étant basée sur la différence de conductance électrique liée à la nature chimique des bases azotées). Le pari, pour Oxford Nanopore réside dans le fait de trouver un mode de détection suffisamment sensible pour analyser puis par puis un signal généré par le passage d’une base au travers de pores (autrement appelés trous). La résolution de la technologie constituait un défit, sa faiblesse provenait (espérons que ce verbe se conjugue aujourd’hui au passé) de la rapidité de passage des bases à travers le pore (1 à 5 µs / base)…
Depuis la publication de 2006, d’Astier and al., J. Am. Chem. Soc., la communauté scientifique attendait la révolution du séquençage 3ème génération que constituait la géniale idée d’Oxford Nanopore : à savoir l’alliance des nanotechnologies, du génie génétique le tout à des fins de séquençage (et plus si affinité). Ainsi, un pore d’alpha-hémolysine (d’environ 1 nanomètre) ou la porine A de Mycobacterium smegmatis ou bien encore du graphène ont été investigués pour devenir le pore idéal dans lequel, après clivage par une exonucléase des liaisons phosphodiester, les bases azotées dont l’enchaînement constitue le séquençage passe afin d’être analysées (par analysées on entend lues, la lecture étant basée sur la différence de conductance électrique liée à la nature chimique des bases azotées). Le pari, pour Oxford Nanopore réside dans le fait de trouver un mode de détection suffisamment sensible pour analyser puis par puis un signal généré par le passage d’une base au travers de pores (autrement appelés trous). La résolution de la technologie constituait un défit, sa faiblesse provenait (espérons que ce verbe se conjugue aujourd’hui au passé) de la rapidité de passage des bases à travers le pore (1 à 5 µs / base)…
Une attente à émouvoir un James Dewey Watson, plusieurs publications en guise de démonstration de la faisabilité d’un tel séquençage, des appels de fonds ayant abouti à l’intégration d’Illumina au niveau du capital d’Oxford Nanopore (investissement en deux fois d’abord de 28 millions $ puis de 41 millions) ont maintenu en haleine les scientifiques avides de séquençage 3ème génération.
Oxford Nanopore est l’un des seuls acteurs européens, dans le monde du séquençage haut-débit où l’hégémonie californienne est écrasante… Alors que les Californiens (PacBio, Life, Illumina) communiquent à grand renfort d’annonces choc, Oxford Nanopore garde une part de mystère (aucune spécification réellement disponible à ce jour), nourrissant notre curiosité avec quelques publications scientifiques et quelques animations dont certaines vous sont présentées ci-dessous.
http://vimeo.com/18630569
Cette première animation reprend les différentes modalités de séquençage
http://vimeo.com/20289048
Avec la vidéo ci-dessus et celle ci-après, Oxford Nanopore réinvente le magnétoscope VHS à visée séquençage haut-débit…
http://vimeo.com/19288315
Finalement, le message à peine subliminal de ce court article : Oxford Nanopore est une société à surveiller de près… de plus en plus près.
Note : si les liens vers les vidéos ne fonctionnent pas essayer toujours en suivant celui-ci…
La HRM est une technique accessible (en temps/finance) sous diffusée. Loin d’avoir l’ambition de réparer cette injustice (à mes yeux), cet article vise à aborder une technique analytique de biologie moléculaire survolée, méconnue et snobée à l’heure de la biologie haut-débit.
La HRM (High Resolution Melt, Courbe de Fusion à Haute-Résolution) permet de distinguer des amplicons (produits PCR) variant d’un minimum de 1 base… (avec plus ou moins de difficulté selon que la mutation concernée est une transversion A/T ou un INDEL par exemple). Cette technique est l’alliance de machine qPCR permettant un contrôle fin de la température (pour ce qui nous concerne nous sommes les heureux possesseurs d’un RotorGene 6000, de Corbett dorénavant vendu sous le nom de Rotor-Gene Q, Qiagen) et d‘agents intercallants de l’ADN saturants et moins « toxiques » pour la PCR comme peut l’être l’EvaGreen. Au rayon des molécules fluorescentes pour cette application vous pourrez trouver le LC Green et LC Green Plus , le ResoLight, Chromofy, SYTO 9 et le SybrGreen I bien sûr ! (de toute évidence sur la plateforme RotorGene, cette molécule permet de bons résultats). Ainsi que le montre les courbes ci-dessous, après amplification, les produits de PCR sont chauffés jusqu’à complète dénaturation par pas de température les plus fins possibles, plus ceux-ci sont petits, plus la technique est résolutive. Le point d’inflexion (l’endroit où la dérivée s’annule) reporté en abscisses est le Tm du produit PCR préalablement amplifié. Or le Tm d’un produit PCR est dépendant de sa concentration, de la concentration en sels du milieu dans lequel il est solubilisé mais surtout de sa composition en bases azotées.
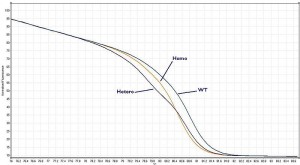
L’hétéroduplexe (la forme hétérozygote) montrera une courbe avec 2 points d’inflexions (ainsi que vous pouvez l’observer sur la courbe ci-dessus). Les 3 populations : homozygotes AA et BB, ainsi que la forme hétérozygote AB se distinguent entre elles.
Au niveau des stratégies de design puis d’analyse, un document plutôt bien réalisé est disponible ici. Les plateformes permettant de réaliser de « vraies » courbes HRM possèdent un logiciel permettant une normalisation qui facilite grandement l’interprétation des résultats.
Les applications de cette techniques sont évidentes :
– le génotypage à bas débit et très bas coût : de simples amorces suffisent (nul besoin de sondes internes doublement marquées) un mastermix contenant un fluorochrome et le tour est joué (il sera judicieux d’avoir sous la main des échantillons références homozygote des deux formes, au minimum). Bien designé, ce diagnostic de génotypage permet une discrimination des substitutions A/T (SNP de classe 4) les plus difficiles à distinguer compte tenu de la faible différence de Tm (autour des 0,2 °C) entre les deux formes.
– le criblage (screening) de mutants : cette application permet de réaliser un premier filtre en vue d’un séquençage (par exemple) sur des zones courtes de l’ADN potentiellement impliquées dans un phénotype particulier. La reproductibilité et la sensibilité de la technique est correcte comme le montre l’article Tù Nguyen-Dumont et al., BMC 2011
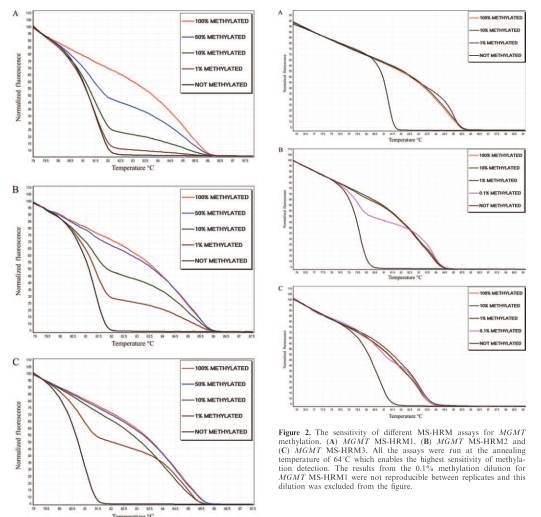
– méthylation de l’ADN : recherche de base 5-méthylcytosine (à l’aide d’un protocole bi-sulfite permettant de « remplacer » les cytosines non méthylées par des uraciles) à l’instar de ce que développe la publication dans NAR de Tomasz K. Wojdac et Alexander Dobrovic. Cette étude montre que l’intermédiaire de la MS-HRM (Methylation-Sensitive High Resolution Melting) il est possible de distinguer des zones méthylées à 0,1 % (ainsi que les courbes tirées de l’article sus-cité montrent pour des sensibilités descendant jusqu’à 1% d’amplicons méthylés, visibles ci-dessous).
En conclusion, la HRM est une technique largement méconnue et sous-employée, relativement simple à développer. Il est nécessaire de normaliser la concentration des échantillons (afin que ceux-ci aient des courbes d’amplification franchissant le seuil autour des 25 Ct). Dans l’idéal, la concentration en sels ne doit pas varier entre les échantillons… appuyé sur un bon design… et le tour est joué. De plus amples informations sont disponibles sur le site gene-quantification.info (la référence en matière de qPCR) ainsi que sur le site highresolutionmelt.co.uk.
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
 L’exome humain, l’ensemble des exons ou encore l’ensemble de ce qui, encodé sous forme d’ADN, peut être traduit en protéines représente environ 1,2% genome humain, 50 Mb et inclus 85 % des mutations référencées.
L’exome humain, l’ensemble des exons ou encore l’ensemble de ce qui, encodé sous forme d’ADN, peut être traduit en protéines représente environ 1,2% genome humain, 50 Mb et inclus 85 % des mutations référencées.
Emilie Lalonde et al., montre l’efficacité du séquençage d’exomes dans un article paru dans Human Mutation de 2010. La démonstration est faite au niveau de la découverte rapide des mutations associées au syndrome de Fowler, impliqué dans la prolifération anarchique des vaisseaux du cerveau qui empêche son développement. Etudiant les exons de deux patients sans lien de parenté cette équipe de Montréal a démontré l’efficacité de la capture d’exons (SureSelect, Agilent) suivi d’un séquençage haut-débit sur plateforme Illumina GAIIx (en moyenne pour les 2 expériences ont été générés 70 millions reads de 76 pb, environ 5 Gbases de séquences brutes).
Techniquement le séquençage d’exons est relativement simple : il implique une capture par hybridation spécifique (NimbleGen, SureSelect de Agilent sont les plus employés), une purification de ces exons ou parties d’exons capturés et le séquençage haut-débit de ceux-ci… L’article de Majewski et al., disponible ici (J. Med. Genet. 2011) cerne avec brio le formidable potentiel de ces nouvelles études, avec son « qu’est ce que le séquençage d’exons peut faire pour vous ? ». Il est assez aisé de comprendre que le séquençage des régions codantes est plus accessible au niveau budget et souvent suffisant pour répondre à beaucoup de questions de cliniciens travaillant à la recherche des causalités de maladies rares. Le schéma ci-dessous, tiré de Leslie G Biesecker, Nature Genetics 42, 13–14 (2010), synthétise la procédure permettant d’aboutir rapidement à la notion de « gènes candidats » (ou plutôt de mutations causales). Les mutations témoins sont accessibles sur les bases de données mondiales dbSNP ou bien encore par l’intermédiaire du projet 1000 Génomes… dont nous avons parlé dans de précédents articles.

L’article montre à quel point l’investigation visant à la découverte des causalités d’une maladie rare, par exemple, a été totalement bouleversée. Ces technologies couplant capture d’exons et séquençage des régions codantes capturées ont d’ores et déjà montré la preuve de leur efficacité. L’article nous rappelle que le génome humain réalisé en 2001 a coûté plus de 2,7 milliards USD, qu’en 2008 un séquençage humain complet coûtait 1,5 millions de USD et qu’aujourd’hui ce même séquençage approche les 10 000 USD…
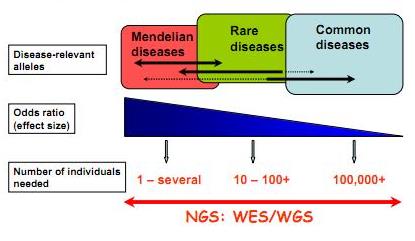
Feuille de route pour l'utilisation du séquençage haut-débit appliqué à l'identification des variations génomiques à l'origine de maladies "génétiques"
Ainsi que le montre Majewski et al. par le schéma ci-dessus plus la maladie que l’on cherche à élucider (si l’on peut s’exprimer ainsi) est la résultante d’une mutation qui a un effet fort, moins la cohorte à séquencer sera importante. En d’autres termes, pour ce qui concerne les maladies multi-factorielles (Alzheimer par exemple), le nombre d’individus dont les exons sont à séquencer sont importants… pour ce type de problématique les GWAS (Genome Wide Association Studies) par l’intermédiaire de scan de régions polymorphes anonymes sur la base de supports tels que les puces Illumina (puces permettant de scanner jusqu’à 4,3 millions de SNPs humains) restent compétitives et d’une puissance comparable. Dans le cas des maladies complexes d’autres stratégies sont développées telles qu’un premier crible sur une grande quantité de cas/témoins sur la base de puce haute densité de SNPs et un focus-séquencing des régions génomiques montrant une association potentielle avec le phénotype étudié…
Le séquençage haut-débit devenant de plus en plus accessible financièrement et par les méthodologies (pipeline d’analyses) de plus en plus disponibles permet d’envisager de nouvelles approches dans la découverte de cibles thérapeutiques potentielles. Ceci étant, l’accessibilité à des quantités de génomes de plus en plus disponibles (sans réel contrôle des organismes d’état) n’est pas sans soulever des problèmes d’éthique profonds et quelque peu négligé dans le débat scientifique actuel.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages