Le PPF Bioinformatique de l’Université Lille 1, l’IFR 142 Médecine Cellulaire et Moléculaire de l’Institut Pasteur de Lille et l’IFR 114 Médecine Prédictive et Recherche Thérapeutique organisent une journée scientifique sur le thème de l’analyse bio-informatique des données produites par les technologies de séquençage à haut débit, dans la lignée de l’édition de 2009. Cette année, les sujets abordés couvriront plus particulièrement les données de type RNA-seq, ChIP-seq,…
Les exposés se tiendront dans l’amphithéatre Butiaux, sur le campus de l’Institut Pasteur de Lille (métro ligne 2, station Grand Palais).
Cette journée sera jumelée au workshop Algorithmique, combinatoire du texte et applications en bio-informatique, qui aura lieu à partir du lendemain au même endroit.
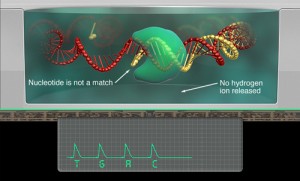
Pour donner suite aux demandes assez nombreuses de détails concernant le principe de fonctionnement du séquençage multiparallélisé PGM (Ion Torrent / Life Technologies).
Sur le lien suivant vous trouverez une vidéo de principe de la technologie Ion Torrent (Life). En supplément, la publication de juillet 2011 de Rothberg et al. dans Nature (pour quelques informations supplémentaires). Il est à noter pour les connaisseurs que Jonathan Rothberg est l’un des fondateurs de la la société 454 Life Sciences (ayant donné naissance au séquenceur haut-débit le plus utilisé dans un passé proche, utilisant le principe du pyroséquençage), technologie vendue à Roche et le créateur de la société Ion Torrent avant que celle-ci ne soit rachetée 350 millions de dollars par Life Technologies.
 pour but, à travers la mutualisation de ressources informatiques des acteurs du projet, de proposer l’utilisation d’applications bioinformatiques déployées sur grille de calcul au plus grand nombre.
pour but, à travers la mutualisation de ressources informatiques des acteurs du projet, de proposer l’utilisation d’applications bioinformatiques déployées sur grille de calcul au plus grand nombre.
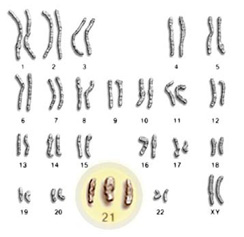 L’étude de Fan et al publiée en 2008 et que nous avions évoquée au travers d’un article précédent, décrivait l’étude de faisabilité d’un diagnostic prénatal non invasif à partir de cellules d’origine fœtale provenant d’une simple prise de sang maternel.
L’étude de Fan et al publiée en 2008 et que nous avions évoquée au travers d’un article précédent, décrivait l’étude de faisabilité d’un diagnostic prénatal non invasif à partir de cellules d’origine fœtale provenant d’une simple prise de sang maternel.
Ces travaux ont fait l’objet de controverses et même si la détection de la trisomie 21 est rendue possible , cela ne l’ était déjà plus pour les trisomies 18 ou 13, par exemple. En cause, la méthode d’analyse employée : l’ensemble des séquences sont alignées par rapport à un génome de référence et l’aneuploïdie foetale est détectée par une surreprésentation du chromosome correspondant, au sein du jeu de données. La sensibilité et l’efficacité de la méthode sont ici directement liées à la profondeur de séquençage. Cette approche ne permet pas de prendre en compte les biais aléatoires ou systématiques liés aux techniques de séquençage, facteurs primordiaux pour cette application.
Dernièrement, les entreprises Sequenom et Verinata Health ont publié des travaux (Sehnert et al.,2011) portant sur le dépistage d’aneuploïdies via le séquençage à haut débit, à partir de cellules d’origine fœtale issues d’une prise de sang maternel : 100% des cas de trisomies 21, 18 et autres anomalies ont été décelés ( soit 27 caryotypes anormaux parmi 48 patients ).
Cette efficacité repose sur une optimisation de l’analyse bioinformatique et notamment sur une considération pour les variations intra et inter-run qui se situent bien souvent à la frontière avec les faibles modifications de la distribution des séquences entre un cas sain et un cas d’aneuploïdie. L’algorithme développé utilise des valeurs de chromosome normalisées établies sur la base d’un ensemble de données de séquençages provenant d’échantillons parmi lesquels certains sont connus comme ayant un caryotype anormal.
En podcast de « clinical chemistry », le Dr. Richard P. Rava, co-auteur de l’article proposé précédemment, revient sur des points de détails et explications concernant le développement de l’algorithme optimisé pour les détections d’aneuploïdies.
Une revue intéressante et qui se veut exhaustive sur les conséquences de la généralisation des technologies de séquençage et les solutions/adaptations possibles, on y retrouve pèle-mêle :
– Un listing à jour (2011) des différentes plateformes dédiées à la génération de données de séquençage (Illumina, Roche, Life Technologie pour ne citer qu’eux…) et leurs spécificités;
– La description de quelques stratégies de NGS : identification de variants, séquençage d’éxome, séquençage sur des régions précises…
– Les problématiques en bioinformatiques : stockage et analyse de données, développement de solutions logicielles adaptées…
– Les différentes analyses ainsi que des listes de logiciels pour répondre aux besoins: assemblage denovo et sur génome de référence, annotation et prédiction fonctionnelle, autant open-source que sous licence payante.
 La PCR est l’un des outils de base dans la boîte du biologiste moléculaire. Devenue accessible, totalement démocratisée dans les laboratoires, elle demande cependant une certaine exigence dans son développement.
La PCR est l’un des outils de base dans la boîte du biologiste moléculaire. Devenue accessible, totalement démocratisée dans les laboratoires, elle demande cependant une certaine exigence dans son développement.
Dans un premier temps il est essentiel de définir à quel type de PCR nous avons à faire :
– PCR de détection (diagnostic ?) : celle qui vous permet de croire que vous avez dans votre extraction l’agent infectieux que vous tâchez de cibler
– PCR de monitoring : celle qui vous suffit pour admettre que vous validez ou non l’expérience que vous désirez suivre, celle qui vous dit que vous avez contaminé votre extraction d’ARN en ADN génomique, par exemple
– PCR de production : celle qui vous permet d’obtenir le plus de produit spécifique afin d’exploiter ces quelques centaines de paires de bases pour en faire ce que vous avez imaginé (reporters à spotter sur puce ADN ou insert pour un clonage)
– PCR quantitative : celle qui se résume à estimer du mieux possible la quantité d’acides nucléiques ciblés présents initialement dans une PCR (cDNA, cible pathogène…).
Il est fondamental de reconnaître à quel genre s’apparente la PCR dont vous avez designé les amorces avec le plus grand soin. Peut être parce que les objectifs visés sont réellement différents voire opposés.
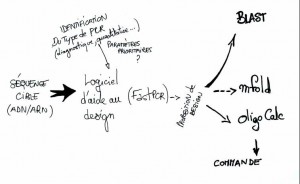
Les grandes étapes aboutissant à un design sont résumées ci-dessus. Il convient d’extraire en format FASTA, la séquence qui vous servira de matrice PCR, le logiciel d’aide permettra d’écarter les amorces de qualités douteuses (auto-appariées, peu spécifiques…), la proposition de design sera soumise dans un Blast (dont les paramètres dépendront du type d’application de votre PCR), dans le cas d’une qPCR SybrGreen il peut être intéressant d’utiliser un mFold sur l’amplicon que vous souhaitez générer, enfin l’ulitsation d’un autre service web tel que OligoCalc permettra de valider les paramètres de vos amorces en confrontant ceux-ci à ceux que vous aurez obtenus en sortie de logiciel d’aide au design.
Commençons par aborder les paramètres d’importances de cette technique de biologie moléculaire : la spécificité, la sensibilité, l’efficacité. Quand la PCR de détection cumulera sensibilité et spécificité, la PCR de monitoring devra être efficace et simple à mettre en place (quelques heures de développement entre ces deux variantes…). La première étape, la plus essentielle, consistera à designer des amorces (nous parlerons des sondes ultérieurement). Avant cela il vous faudra vous doter d’une stratégie de design et peut être d’un (le mieux serait plusieurs) logiciel d’aide au design d’amorces. Parmi tous les logiciels disponibles, rien ne sert de casser le cochon, il existe beaucoup de gratuiciels acceptables pour la grande majorité des applications. Parmi ceux-ci, notons :
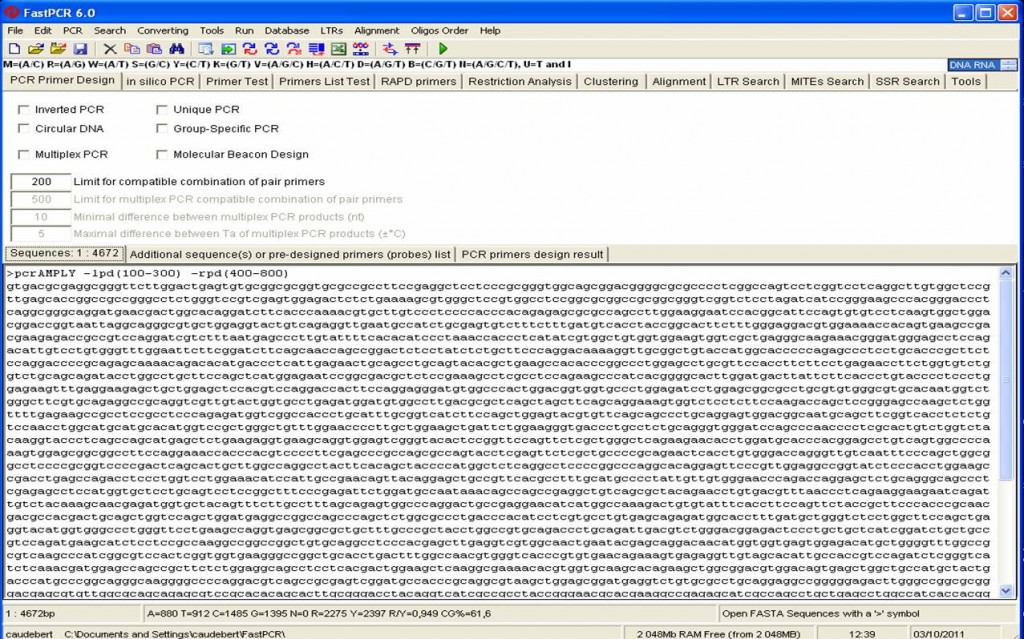
– FastPCR : quelques bugs, mais globalement cette solution à notre préférence pour sa modularité. Le logiciel gère les PCRs multiplexes, propose un grand nombre d’outils intégrés de manipulations de séquences. Il s’agit de l’une des seules solutions gratuites qui est l’équivalent d’une solution payante (mode d’emploi FastPCR disponible). Le logiciel quant à lui est disponible ici (dans sa version antérieure, plus stable, dont nous voyons une capture d’écran ci-dessous).
Parmi les critères généraux, les paramètres de design transposables à tout type de PCR sont les suivants :
– Longueur des oligonucléotides = 16 à 26 nt
– Taux de GC = 40 à 70% (idéalement autour des 55 %)
– Tm des oligonucléotides ~ 60°C (si vous développez une PCR diagnostique la température d’amorçage = Tm, si vous souhaitez produire il faudra diminuer de 2°C la température d’amorçage par rapport au Tm des amorces)
Du GC%, de la concentration en amorces et de la concentrations en cations dépend le Tm (définie comme la température pour laquelle 50 % d’une séquence donnée est sous forme double hélice et 50 % est sous forme simple brin). C’est donc ces paramètres qu’il faut intégrer lors du design. Bien maîtrisé, un design permettra de gagner un temps précieux lors des phases de mise au point et d’optimisation d’une PCR. Ceci étant particulièrement vrai pour ce qui concerne la mise au point d’une PCR quantitative.
D’autres paramètres évidents concernent la séquence ciblée en tant que telle. Sachant qu’en fonction de celle-ci la marge de manœuvre est plus ou moins grande.
– maximiser autant que possible les zones de grandes complexité linguistique (celle avec le moins d’homopolymères, FastPCR tient compte de cette notion pour designer des amorces et se sert de ce paramètre pour calculer son indice de qualité de design)
– en 3′ des amorces, sur les 5 dernières bases, autant que possible, rassembler un maximum de bases A ou T (3 A ou T sur ces 5 dernières bases) afin d’en diminuer le Tm en 3′ et ainsi d’accroître la spécificité des amorces designées
Selon le type de PCR à laquelle nous avons à faire les contraintes de design peuvent être radicalement différentes :
– ainsi, vous souhaiterez designer dans 2 exons différents lorsque vous souhaiterez suivre la contamination en ADNg d’une extraction d’ARN par exemple
– ainsi vous souhaiterez designer sur une région à cheval sur 2 exons (sur la base donc d’une séquence cDNA, ARN) pour les applications de qPCR liées à la mesure du niveau de modulation différentielle de votre ARN cible
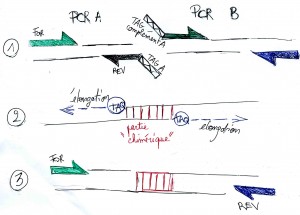
Truc et astuce pour diminuer vos coûts de séquençage d’amplicons en SANGER : la PCR hybride !
1/ Réalisation de PCR en tubes indépendants, l’amorce reverse (PCR A) et l’amorce forward (PCR B) des 2 PCR auront des séquences Tag synthétiques complémentaires
2/ La deuxième étape consistera en une élongation unidirectionnelle. Les 2 amplicons synthétisés lors de la PCR 1 s’apparieront grâce à leur séquence Tag complémentaire. Le produit de la PCR2 sera un hybride PCR A-séquence chimérique – PCR B
3/ Une dernière PCR, prenant la Forward de PCR A et la Reverse de PCR B seront utilisées… le produit néosynthétisé pourra être séquencé (2 ou plus pour le prix d’un, en outre bien optimisée ce type de PCR peut permettre d’amplifier des « signaux faibles »).
 On a (presque) oublié la vocation première de Louis Ferdinand Destouches plus connu sous son nom de plume L.F.Céline… un médecin occasionnel devenu homme de lettres au style révolutionnaire. L’un des premiers à faire incorporer le langage de la rue dans des romans lucides, engagés ou nihilistes. Il a su jouer sur des sonorités, il a ajouté la musique dans le roman traditionnel. L’un de ses tous premiers écrits est disponible aux éditions Gallimard collection Imaginaire : Semmelweis (préface inédite de Philippe Sollers), 1999, 128 p. Cette édition comporte une bibliographie sur Semmelweis établie par Jean-Pierre Dauphin et Henri Godard, ainsi que différents textes parus après la soutenance de la thèse de Louis Ferdinand en 1924. Dès l’écriture de sa thèse LFC affiche son style : sonore, provoquant, sans concession. Cette thèse est d’ores et déjà un objet littéraire à part entière (lors de sa soutenance Céline à 30 ans). Le sujet de cette biographie analytique : « la vie et l’oeuvre de Philippe Ignace Semmelweis (1818-1865) » est singulier à plusieurs niveaux : l’histoire d’un confrère (de LFC) « scientifique » face à une conviction dont il n’arrive pas à tirer une démonstration, l’histoire d’une opposition au conservatisme, un homme fort de ses déductions dont les paires rejettent les conséquences (un Galilée de la médecine). Dans sa thèse Céline décrit la valse macabre de la fièvre puerpérale qui passe des salles de dissection aux salles d’accouchements. Semmelweis convaincu – sans arriver à le démontrer formellement- que le vecteur de ces infections étaient les mains souillées des étudiants qui passaient de salles mortuaires aux salles d’accouchement. Céline peint un Semmelweis comme un obstétricien maniaque, déterminé, persuadé d’être dans le vrai quand, après observation, il essaie de persuader ses confrères de se laver les mains au chlorure de chaux -une idée de l’aseptie avant l’invention du mot microbe (par Charles Sédillot en 1878, 13 ans après la mort de Semmelweis). Une biographie tragique où l’obscurantisme est un des éléments poussant Semmelweis vers la folie. Quelques décennies plus tard, Louis Pasteur connaîtra un autre sort.
On a (presque) oublié la vocation première de Louis Ferdinand Destouches plus connu sous son nom de plume L.F.Céline… un médecin occasionnel devenu homme de lettres au style révolutionnaire. L’un des premiers à faire incorporer le langage de la rue dans des romans lucides, engagés ou nihilistes. Il a su jouer sur des sonorités, il a ajouté la musique dans le roman traditionnel. L’un de ses tous premiers écrits est disponible aux éditions Gallimard collection Imaginaire : Semmelweis (préface inédite de Philippe Sollers), 1999, 128 p. Cette édition comporte une bibliographie sur Semmelweis établie par Jean-Pierre Dauphin et Henri Godard, ainsi que différents textes parus après la soutenance de la thèse de Louis Ferdinand en 1924. Dès l’écriture de sa thèse LFC affiche son style : sonore, provoquant, sans concession. Cette thèse est d’ores et déjà un objet littéraire à part entière (lors de sa soutenance Céline à 30 ans). Le sujet de cette biographie analytique : « la vie et l’oeuvre de Philippe Ignace Semmelweis (1818-1865) » est singulier à plusieurs niveaux : l’histoire d’un confrère (de LFC) « scientifique » face à une conviction dont il n’arrive pas à tirer une démonstration, l’histoire d’une opposition au conservatisme, un homme fort de ses déductions dont les paires rejettent les conséquences (un Galilée de la médecine). Dans sa thèse Céline décrit la valse macabre de la fièvre puerpérale qui passe des salles de dissection aux salles d’accouchements. Semmelweis convaincu – sans arriver à le démontrer formellement- que le vecteur de ces infections étaient les mains souillées des étudiants qui passaient de salles mortuaires aux salles d’accouchement. Céline peint un Semmelweis comme un obstétricien maniaque, déterminé, persuadé d’être dans le vrai quand, après observation, il essaie de persuader ses confrères de se laver les mains au chlorure de chaux -une idée de l’aseptie avant l’invention du mot microbe (par Charles Sédillot en 1878, 13 ans après la mort de Semmelweis). Une biographie tragique où l’obscurantisme est un des éléments poussant Semmelweis vers la folie. Quelques décennies plus tard, Louis Pasteur connaîtra un autre sort.
 Il est de nos jours surprenant et presque déstabilisant, de lire une thèse de médecine, de moins de 80 pages qui constitue un objet littéraire abouti, une observation scientifique et humaine, argumentée et subtile. Seul contre tous, Semmelweis a maintenu sa position. Quant à lui, Céline par la voix de Bardamu -médecin anti-héros du Voyage au bout de la nuit- donne une définition de la médecine libérale : « larbin pour les riches, voleur pour les pauvres. »
Il est de nos jours surprenant et presque déstabilisant, de lire une thèse de médecine, de moins de 80 pages qui constitue un objet littéraire abouti, une observation scientifique et humaine, argumentée et subtile. Seul contre tous, Semmelweis a maintenu sa position. Quant à lui, Céline par la voix de Bardamu -médecin anti-héros du Voyage au bout de la nuit- donne une définition de la médecine libérale : « larbin pour les riches, voleur pour les pauvres. »
HEALTH MAP : WHO is watching you !
( -L’OMS vous regarde !-)
Encore une application liée à Google Map.
Grossièrement, à l’aide de :
contributions « volontaires » (par l’intermédiaire d’une application sur smart phone : Outbreak Near Me – « épidémie près de chez moi », cf. capture d’écran ci-contre) vous pouvez ainsi « dénoncer » votre voisin qui a la grippe (à vous la charge du diagnostic…), et le cas sera (peut-être) référencé, associé à une date et une géolocalisation puis reporté sur la planisphère) – simple efficace un tantinet intrusif.
en scrutant de manière automatisée à large échelle le contenu des réseaux sociaux, les flux de nouvelles locales, les dépêches, les flux d’informations informelles (SMS, Twitter…), et en appliquant une « analyse sémantique » automatisée du contenu. Cet outil est capable, en se référant à un dictionnaire des localités et des maladies, d’attribuer un cas relaté d’une maladie donnée à une géolocalisation que l’on souhaite la plus précise possible. Un outil basé sur le principe de détection sémantique contextuelle (cf. bibliographie en accès gratuit dans PubMed)
L’objectif ? Suivre en temps proche l’évolution d’une pandémie, observer avec un temps d’avance l’arrivée de la grippe saisonnière par exemple… avec plus de 2300 lieux, 1100 « maladies » dans le dictionnaire de référence… la vigie automatisée a du travail !
 Le projet peut paraître séduisant. Il constitue un fantastique système de surveillance (un de plus, dirons les septiques) pour l’OMS. Même si l’information est bruitée (suite à un mauvais diagnostic, une information journalistique n’est pas une information scientifique) ou comporte des erreurs dans le couple d’attribution maladie / lieu, l’outil a misé sur la vélocité dans son aptitude à remonter de l’information que l’on souhaite la plus pertinente possible. Il est en perpétuelle amélioration. Sur Health Map, vous pouvez suivre jour après jour, tels des boutons de varicelles, des spots en relation avec votre agent infectieux favori s’illuminent sur la carte mondiale. Outre l’outil (une application de la fouille de texte, encore une) le site internet est très ergonomique et utile notamment pour sa section : « The Disease Daily« . Une critique possible : le continent africain fait les frais de son manque de couverture par les systèmes d’informations et de communication, la Chine semble, quant elle, assez épargnée par les pandémies… l’outil sémantique intègre t’il le cantonais ou le mandarin… ? A vous de juger.
Le projet peut paraître séduisant. Il constitue un fantastique système de surveillance (un de plus, dirons les septiques) pour l’OMS. Même si l’information est bruitée (suite à un mauvais diagnostic, une information journalistique n’est pas une information scientifique) ou comporte des erreurs dans le couple d’attribution maladie / lieu, l’outil a misé sur la vélocité dans son aptitude à remonter de l’information que l’on souhaite la plus pertinente possible. Il est en perpétuelle amélioration. Sur Health Map, vous pouvez suivre jour après jour, tels des boutons de varicelles, des spots en relation avec votre agent infectieux favori s’illuminent sur la carte mondiale. Outre l’outil (une application de la fouille de texte, encore une) le site internet est très ergonomique et utile notamment pour sa section : « The Disease Daily« . Une critique possible : le continent africain fait les frais de son manque de couverture par les systèmes d’informations et de communication, la Chine semble, quant elle, assez épargnée par les pandémies… l’outil sémantique intègre t’il le cantonais ou le mandarin… ? A vous de juger.
Partenaires, collaborateurs & supports du projet : GOOGLE.ORG, NATIONAL INSTITUTES OF HEALTH RESEARCH / NATIONAL LIBRARY OF MEDICINECENTERS FOR DISEASE CONTROL AND PREVENTION, CANADIAN INSTITUTES OF HEALTH RESEARCHINTER, NATIONAL SOCIETY FOR INFECTIOUS DISEASES / PROMEDHEALTH AND HUMAN SERVICES, FLU.GOV, INTERNATIONAL SOCIETY FOR DISEASE SURVEILLANCE, INTERNATIONAL SOCIETY FOR TRAVEL MEDICINEGEOSENTINE, NEW ENGLAND JOURNAL OF MEDICINE, WILDLIFE CONSERVATION SOCIETY
 Il existe un endroit, un immeuble abritant un plateau technologique où les capacités de séquençage dépassent l’imaginable. Le pari est osé. Ce centre est confondu avec le BGI (Beijing Genomics Institute) alors qu’il se situe à un petit 2000 kms par route. Disons que Shenzhen, la quatrième ville du pays-continent en nombre d’habitants, se situant en bordure de Honk Hong, a un statut de zone économique spéciale. Depuis 1980 les entreprises étrangères sont autorisées à investir dans ce lieu propice… et c’est ainsi que cette localité a vu s’ouvrir le premier Mac Donald’s en Chine en 1990 (quelle chance !).
Il existe un endroit, un immeuble abritant un plateau technologique où les capacités de séquençage dépassent l’imaginable. Le pari est osé. Ce centre est confondu avec le BGI (Beijing Genomics Institute) alors qu’il se situe à un petit 2000 kms par route. Disons que Shenzhen, la quatrième ville du pays-continent en nombre d’habitants, se situant en bordure de Honk Hong, a un statut de zone économique spéciale. Depuis 1980 les entreprises étrangères sont autorisées à investir dans ce lieu propice… et c’est ainsi que cette localité a vu s’ouvrir le premier Mac Donald’s en Chine en 1990 (quelle chance !).- Son premier séquençage de novo du génome humain et de mammifère (le panda géant, séquencé sur la plateforme GA de Illumina) à l’aide de technologies de séquençage haut-débit à reads courts (publications de 2010 : Nature et Genome Reasearch)
- Son séquençage du premier homme pré-historique (à partir d’un prélèvement de cheveux conservé au muséum national du Danemark) (publication : Nature, 2010)
- Son séquençage du premier génome diploïde d’un individu asiatique dans le cadre du projet de Yan Huang (publication : Nature 2008)
- Sa construction d’une carte pan-génomique humaine, avec un ajout de 19 à 40 Mbases absentes de la séquence de référence humaine (publication : Nature Biotechnology 2010)
- Sa contribution à hauteur de 10% à l’information des séquences pour le projet HapMap humain
- Sa contribution à hauteur de 1% au Projet génome humain de référence (seul institut au monde en développement à contribuer au projet)
- Sa démonstration de la faisabilité du séquençage du microbiome du tube digestif humain, estimé à 150 fois plus grand que le génome humain (publication : Nature 2010)
- Son action clé dans le projet sino-britannique du projet de séquençage du génome de poulet
- Le BGI avec Shenzhen est le principal centre de séquençage dans le projet 1000 génomes, elle a été la première institution chinoise à séquencer le virus du SRAS, quelques heures seulement après le premier séquençage du virus par des Canadiens, et a été un acteur clé dans l’analyse de l’épidémie à E. coli O104:H4
Les projets 10 000 génomes microbiens, 1000 génomes eucaryotes sont souvent qualifiés de pharaoniques. Cependant avec cette quantité de machines, avec l’argent disponible pour le consommable soit plus de 1,5 milliards de USD sur 10 ans, avec de la main d’oeuvre qualifiée disponible et un « pipe-line » d’assemblage et d’analyse développé… ces deux projets devraient être achevés comme prévu (cette notion a cours en Chine).
Souvent les projets pleuvent, mais les financements un peu moins. Avec des débits (calculés selon les spécifications des fournisseurs de séquenceurs) de plusieurs génomes humains / jour, avec l’équivalent de plus de 100 millions d’USD d’investissements en unités de séquençages, il reste du temps machine à occuper.
Un virage économico-scientifico-politique a été amorcé par le gouvernement chinois. Ceci peut être résumé par l’équation suivante : investissement massif d’argent avec objectifs scientifiques à court et moyen terme, rentabilisation quasi- immédiate de l’investissement matériel en l’ouvrant à des prestations de service, assommer la concurrence en occupant le terrain des publications scientifiques et en proposant des prix de séquençage cassés (cf. publicité ci-dessus, source site web du BGI Europe) en ce qui concerne les prestations de service (Shenzhen, zone économique spéciale n’est pas une implantation géographique choisie au hasard… la génomique aux portes de Macao et de Honk-Hong).
Forts de cette mécanique bien huilée, les scientifiques chinois trustent les bonnes feuilles de Nature… investissement financier avec un fort impact bibliométrique. Le gouvernement français s’est inspiré des bonnes recettes chinoises : des investissements publics colossaux dans des sciences technophages, une collaboration réelle ou feinte des secteurs public et privé, un retour sur investissement rapide sous forme de publications. Après le grand emprunt, le gouvernement français soucieux de cumuler rayonnement scientifique international et relance de la croissance économique, a injecté quelques deniers en Equipex et Labex. La science comme moteur de croissance économique. Attention au retour sur investissement car, quand les bourses se contractent le créancier devient plus soucieux du rendement de ses deniers.
En conclusion, si le modèle chinois semble vertueux sur le plan du développement scientifique et technologique, il est basé avant tout sur un modèle économique où le dumping social, où le rendement bibliographique à court terme deviennent pierres angulaires de la techno-science. La technologie doit servir la science, l’inverse n’a pas de sens. La Chine a l’ambition de devenir une bibliothèque d’Alexandrie numérique avec son projet « library of digital life« . Après avoir conquis l’espace, les marchés mondiaux, après être devenu le créancier des Etats Unis, la Chine souhaite devenir la puissance scientifique (c’est surtout exact concernant les sciences de la vie) qui éclaire le monde.
 Il s’agit d’un run sans valeur biologique qui a été réalisé dans le cadre d’une validation proposée par le fournisseur (Life) pour le « label CSPro ». Ce run consiste notamment, en un re-séquençage de E. coli DH10B.
Il s’agit d’un run sans valeur biologique qui a été réalisé dans le cadre d’une validation proposée par le fournisseur (Life) pour le « label CSPro ». Ce run consiste notamment, en un re-séquençage de E. coli DH10B.Ce type de données peut (vous) permettre d’évaluer la technologie (taux d’erreur, profondeur…) de séquençage haut-débit d’une part. D’autre part ces données peuvent servir afin d’évaluer les logiciels d’assemblage tels que ceux que nous possédons (DNastar, CLC genomic workbench, Partek…) pour des reads issus de PGM (Ion Torrent).
- Accès au jeu de données librairie DH10B au format sff (compressé)
- Accès à la référence, génome annoté de DH10B
- Accès au rapport de run au format pdf
- Accès au Library Sequence (FASTQ)
Vous trouverez ci-dessous une partie du rapport de run relative à la qualité des reads générés et alignés sur le génome de référence à l’aide de la suite Ion Torrent version 1.4.1
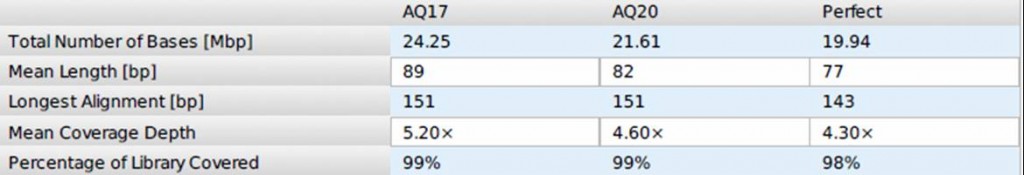
Ce run a été réalisé sur une puce 314, consommable fourni pour 10 Mbases de séquençage brut (ici nous dépassons les spécifications, 32.99 Mbases séquencées, même si le présent run est considéré comme assez moyen par rapport aux résultats précédemment obtenus). Malgré tout, il est possible de couvrir un petit génome à plus de 4X à des coûts imbattables (pour l’instant)!
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages