Le contenu scientifique a bien souvent besoin d’être traduit et la démarche du chercheur nécessite d’être expliquée (nous ne sommes pas très loin des notions d’éthique). La vulgarisation peut parfois être un outil permettant d’éveiller l’opinion qui, de ce fait peut s’en faire une, ou à rebrousse poil, elle peut être une méthode visant à la rassurer. Nous pouvons lire et relire les méthodes employées par Edward Bernays pour transformer le citoyen en consommateur.
Le contenu scientifique a bien souvent besoin d’être traduit et la démarche du chercheur nécessite d’être expliquée (nous ne sommes pas très loin des notions d’éthique). La vulgarisation peut parfois être un outil permettant d’éveiller l’opinion qui, de ce fait peut s’en faire une, ou à rebrousse poil, elle peut être une méthode visant à la rassurer. Nous pouvons lire et relire les méthodes employées par Edward Bernays pour transformer le citoyen en consommateur.
La biotechnologie alimente les peurs par ce qu’elle peut engendrer dans un laboratoire et qui échapperait au contrôle citoyen dès lors que l’invention devient produit, par exemple. Cet article est l’occasion d’écouter un exemple de vulgarisation scientifique : l’émission de Là-bas si j’y suis (excellente émission du service public, sur France Inter), du vendredi 15 décembre 2006, » Leçon de choses : Les OGM « .
Dans cette émission Christian Vélot explique avec un magnifique esprit de synthèse ce que sont les OGM, dans son Laboratoire de recherche en Génétique Moléculaire, à Orsay. Christian Vélot est un lanceur d’alerte, un exemple de libre chercheur, libre penseur qui a réussi à faire sortir la science de son laboratoire pour l’amener dans le champ du débat… une nécessité absolue surtout s’agissant des applications biotechnologiques. L’intervention de Christian Vélot, dans l’émission de Daniel Mermet est un modèle du genre, en effet bien que techniquement imparfaite, elle incite l’auditeur à prendre du recul et à aller se documenter sur le sujet en le dépassionnant, en évitant l’ornière des sempiternels poncifs. De la connaissance naît la curiosité qui enrichit l’argumentation, essence de toute opinion et l’accès à ces arguments est la voie vers la décision et donc vers… la démocratie.
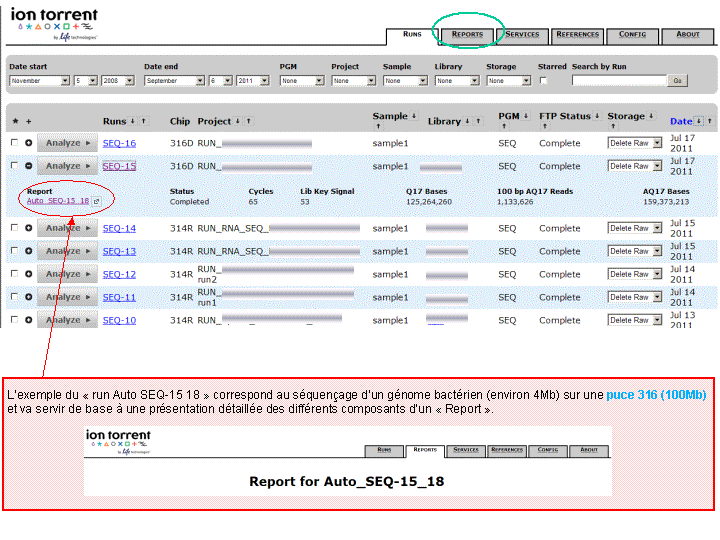
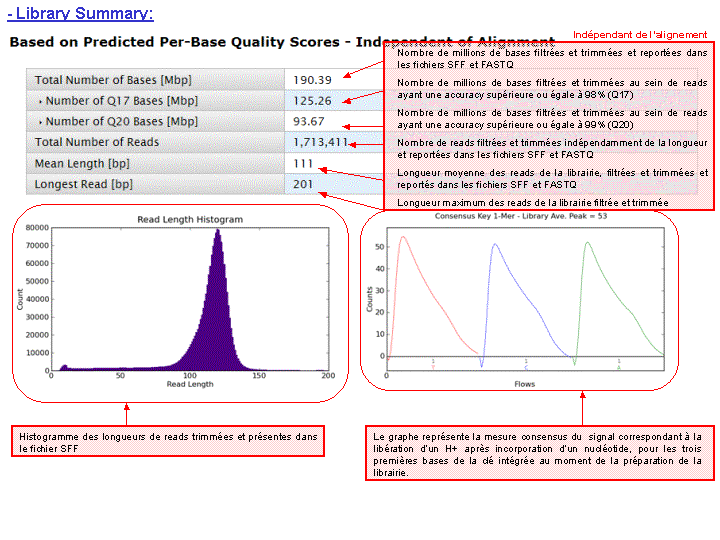
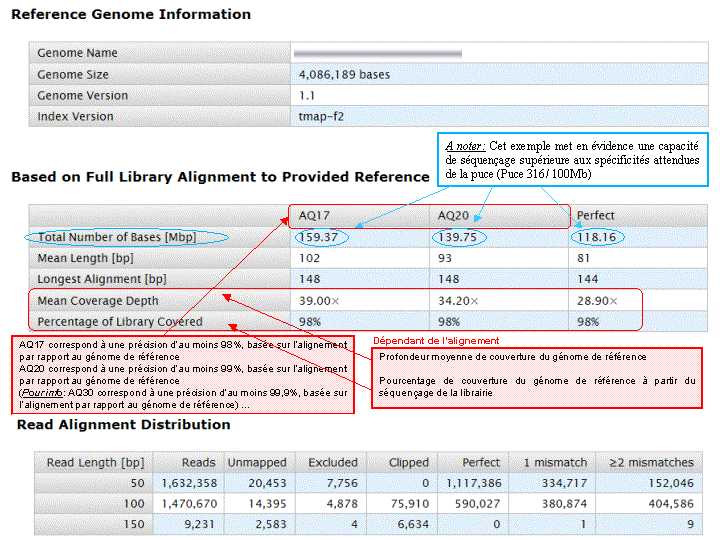
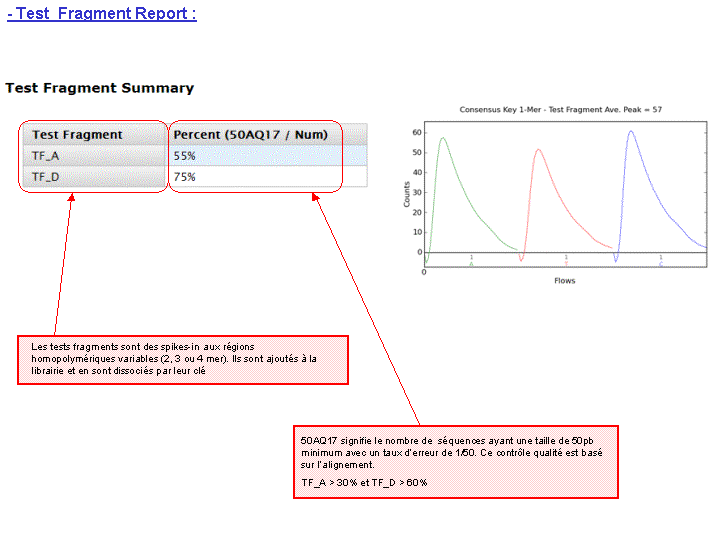
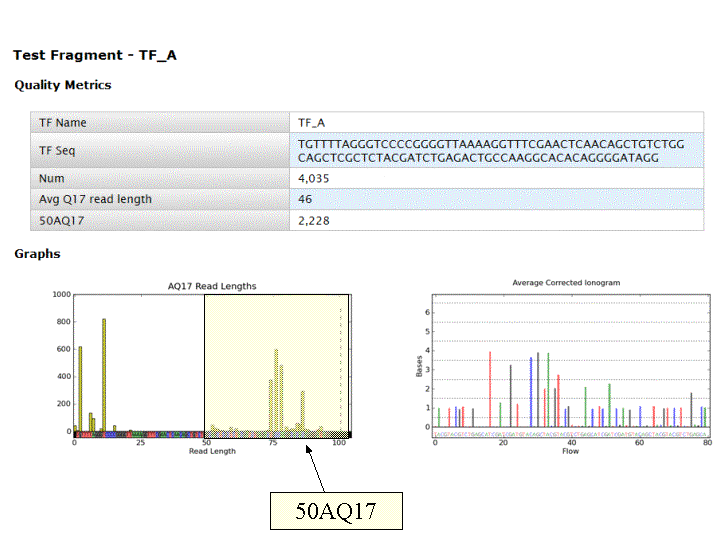
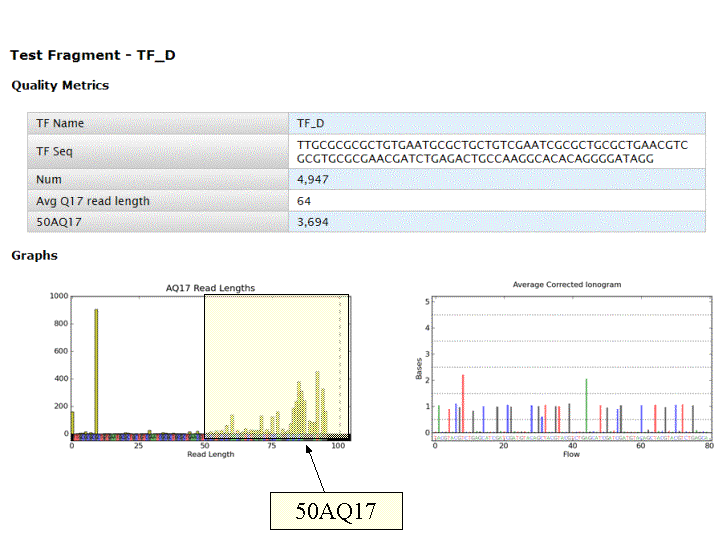
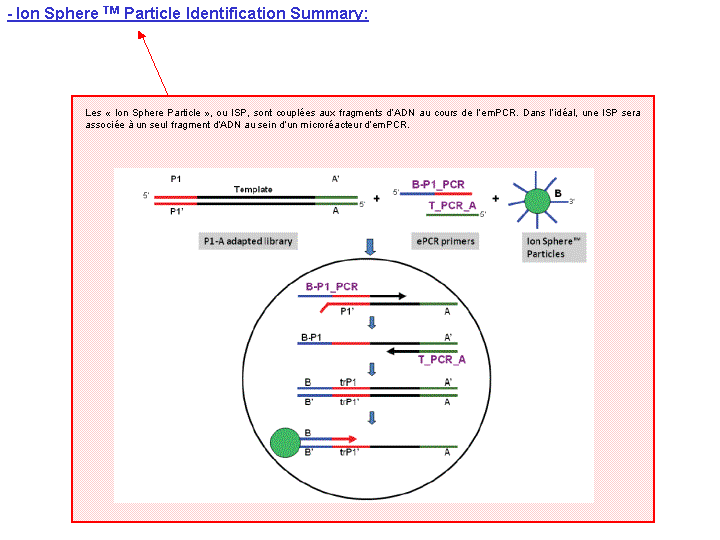
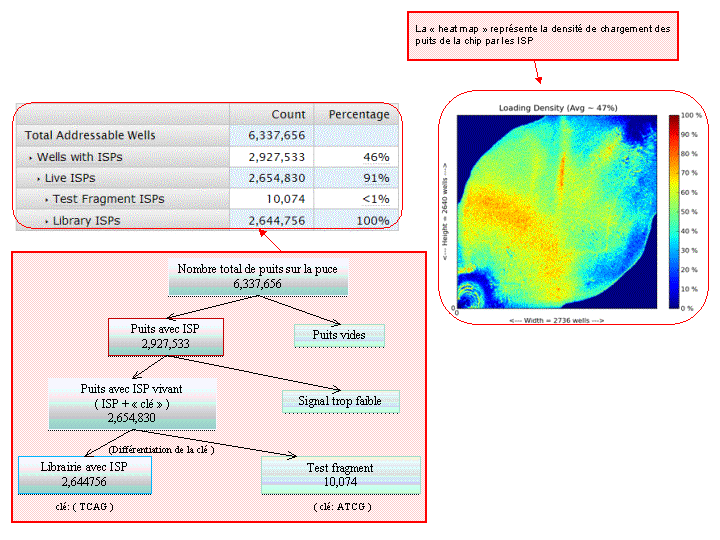
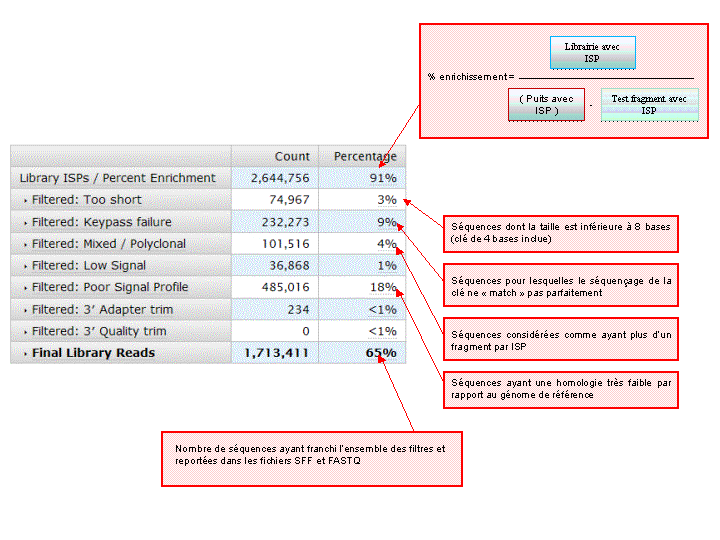
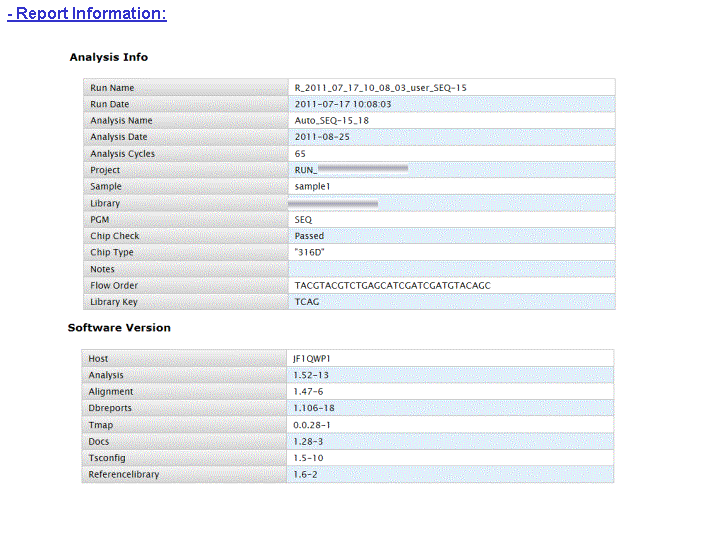
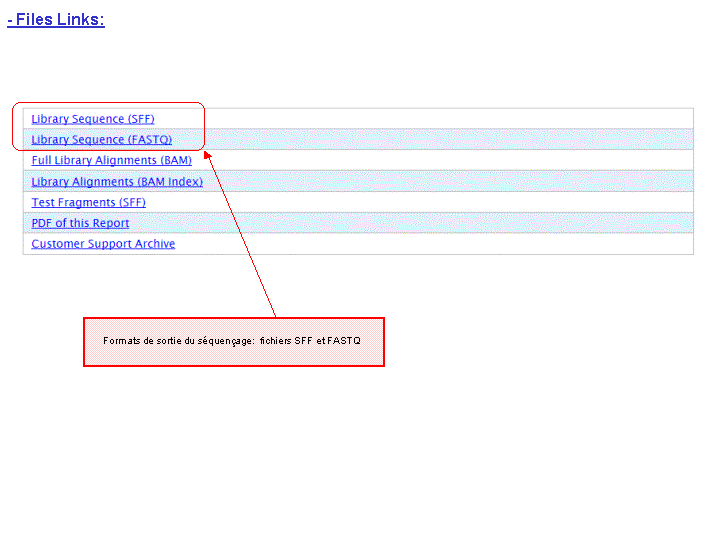
A l’issue d’un « run » de séquençage Ion Torrent (PGM), l’ensemble du signal brut (ionogramme) est converti en séquences et stocké au niveau du serveur. Pour chaque « run de re-séquençage», un alignement préliminaire est réalisé sur la base du génome de référence mentionné lors de l’initialisation du PGM. Cette information est reprise au travers d’un rapport qui comporte également un ensemble de paramètres, que l’on se propose de détailler ci-dessous :
Le rapport se divise en 5 sections:
Le ppf (Portail du Plan Pluriformation) bioinformatique de Lille 1 organise le 20 septembre prochain sur le campus de Lille 1 une journée scientifique sur la fouille de texte pour la biologie (extraction d’information en génomique, inférence des interactions géniques, …).
Le programme et les détails pratiques sont disponibles sur la page http://www.lifl.fr/~touzet/PPF/fouilletexte11.html
Il convient tout d’abord de distinguer deux types de bioinformaticiens :
-Ceux qui sont intégrés dans une unité de recherche : ils sont souvent spécialisés sur une thématique ou même un projet précis : généralement isolés, non permanent, impliqués et informés des problématiques biologiques
-Ceux qui font partie d’une unité de bioinformatique : en émulation avec d’autres bioinformaticiens, moins précaires, plus loin des problématiques biologiques
Ce document se veut une libre opinion sur la bonne intégration d’un, ou d’une équipe, de bioinformaticiens au sein d’un groupe de recherche et les clés pour la bonne réussite de celle-ci. Il est en grande partie inspiré de l’éditorial « On the organization of bioinformatics core services in biology-based research institutes » agrémenté de commentaires plus personnels.
Pour le bien-être interne et externe d’une plateforme de bioinformatique, il convient de :
-Bien séparer les tâches dîtes de prestation et les activités de recherches.
-Scinder les sujets traités par tâches bien définies : par exemple, support en développement et maintenance de base de données, analyse statistique de données haut-débit, support pour le déploiement d’applications sur environnement HPC, analyse de donnée NGS…
-Mettre en place un comité d’utilisateurs incluant des utilisateurs biologistes/bioanalystes chargés d’établir les priorités
-Mettre en place des outils pour suivre l’activité bionformatique de la façon la plus transparente possible
-Mettre en place au maximum des projets transversaux pour favoriser l’interface entre biologiste et bioinformaticiens, les interactions pluridisciplinaires sont les plus enrichissantes et souvent les plus productives. Comme toutes disciplines, la bioinformatique peut devenir endogame, particulièrement dans ou le cadre où l’on parle de bioinformatique comme une science à part entière, et non comme un outil au service de problématiques biologiques : une équipe de bioinformatique peut très bien travailler sur des sujets « porteurs » (donc plus facile à financer) sans interagir, ou seulement sporadiquement, avec des biologistes pour valider le bien-fondé du travail. C’est une des dérives du système d’évaluation actuel, pour plus d’information, lire la libre opinion publiée dans ce blog : Chemins de traverses : la recherche d’excellence ou quand les mots perdent leur sens.
-Mettre en place des formations pour les biologistes : collaborer avec des personnes connaissant un minimum les problématiques bioinformatiques est plus efficace
-Dans le même esprit, nommer un bioinformaticien responsable pour le support utilisateurs pour assister les biologistes dans leurs tâches quotidiennes de bioanalyse est très utile pour entretenir de bonne relation avec les laboratoires de recherche
-Pour les bioinformaticiens intégrés dans une unité, il est primordial de trouver des structures permettant de rencontrer et de partager avec d’autres bioinformaticiens, à défaut, passer quelques heures par semaine à effectuer de la veille technologique peut être un palliatif.
Pour conclure, une équipe de bioinformaticiens doit communiquer un maximum avec leurs partenaires biologistes afin de favoriser une saine interaction, un bioinformaticien, seul, entourer de biologistes se doit de rester à la page, idéalement en partageant et en communiquant sur ces projets à d’autres bioinformaticiens.
 Cet article est le prolongement sous forme de fiche technique, de notre article abordant le bio-art.
Cet article est le prolongement sous forme de fiche technique, de notre article abordant le bio-art.
Commençons par quelques définitions :
fingerprinting = signature moléculaire exploitant les polymorphismes individuels
fingerprinting = (exploitation des polymorphismes pour singulariser un prélèvement) + (séparation sur gel) + (visualisation par hybridation de sondes spécifiques marquées)
L’ensemble du génome humain est commun à tous aux mutations près. Ces mutations se produisent au hasard et se produisent à une moyenne proche de 1 ‰.
Lorsque l’on digère le génome humain (3,5 x 109 bp pour un génome haploïde) par une enzyme de restriction dont le site est à 6 bp (probabilité moyenne de coupure (1/4)6=1/4096 soit 1 site tous les 4 kbp), on obtient de l’ordre de 106 fragments qu’on ne peut examiner un à un pour déterminer la taille d’un fragment particulier. Il faut un moyen de repérer la région où la mutation est susceptible de s’être produite. Une caractéristique unique d’une région particulière d’ADN est sa séquence (à condition de n’être pas de l’ADN répété). Ainsi, après séparation des fragments selon leur taille, un blotting (ou buvardage) sera utilisé pour réaliser la signature moléculaire à l’aide de sondes marquées par un radio-isotope ou avec une molécule « traçante ».
Le southern blot est une technique permettant, après une phase de séparation sur gel, de visualiser spécifiquement des loci ciblés. Cette technique a été mise au point par E. M. Southern qui a combiné électrophorèse, transfert et hybridation avec une sonde marquée pour permettre une détection spécifique d’un locus cible (1975).
Après électrophorèse …
1. Le gel d’électrophorèse est immergé dans une solution alcaline permettant la dénaturation de l’ADN
2. Une membrane de nitrocellulose (ou de nylon) imprégnée d’une solution saline concentrée est appliquée directement sur le gel, un poids la maintient en sandwich entre une couche épaisse de substance absorbante et le gel. Trois moyens sont disponibles pour attirer la solution saline à travers le gel :
– par capillarité
– par électrophorèse
– par succion sous vide
3. La membrane est alors chauffée, dans le cas de nitrocellulose, ou exposée au rayonnement ultra-violet pour le cas du nylon, afin de fixer de manière permanente l’ADN sur la membrane. Une étape de préhybridation permettant de traiter les sites laissés libres, ils seront saturés par une solution contenant une protéine telle que de la sérum albumine et / ou traités à l’aide d’un détergent comme le laurylsulfate de sodium.
4. Une sonde spécifique marquée complémentaire de l’ADN cible est mise en contact de la membrane sur laquelle il a été transféré.
5. Une phase de « visualisation » de l’hybridation aura lieu soit par autoradiographie dans le cas de l’emploi d’une sonde marquée à l’aide d’un radio isotope (phosphore 32 par exemple) ou par développement d’un film photographique impressionné après réaction de chimioluminescence.
Cette technique est de moins en moins utilisée dans les laboratoires d’identification (police scientifique, laboratoires vétérinaires), elle a été remplacée par l’électrophorèse capillaire après une PCR sur des régions flanquantes de microsatellites (cette technique, beaucoup plus automatisable et standardisable a supplanté le SouthernBlot). Aujourd’hui cette technique tend à être remplacée par des panels de SNPs.
Deux techniques analytiques de biologie moléculaire dérivent du Southern Blot par goût du jeu de mot, elles ont été nommées :
– Northern Blot, technique analytique des ARN (James Alwine, David Kemp, and George Stark at Stanford University, 1977)
– Western Blot, technique analytique des protéines (W. Neal Burnette, 1979-1981)
Le quatrième point cardinal, le Eastern Blot, beaucoup plus marginal consiste en l’analyse des modifications post traductionnelles des protéines.
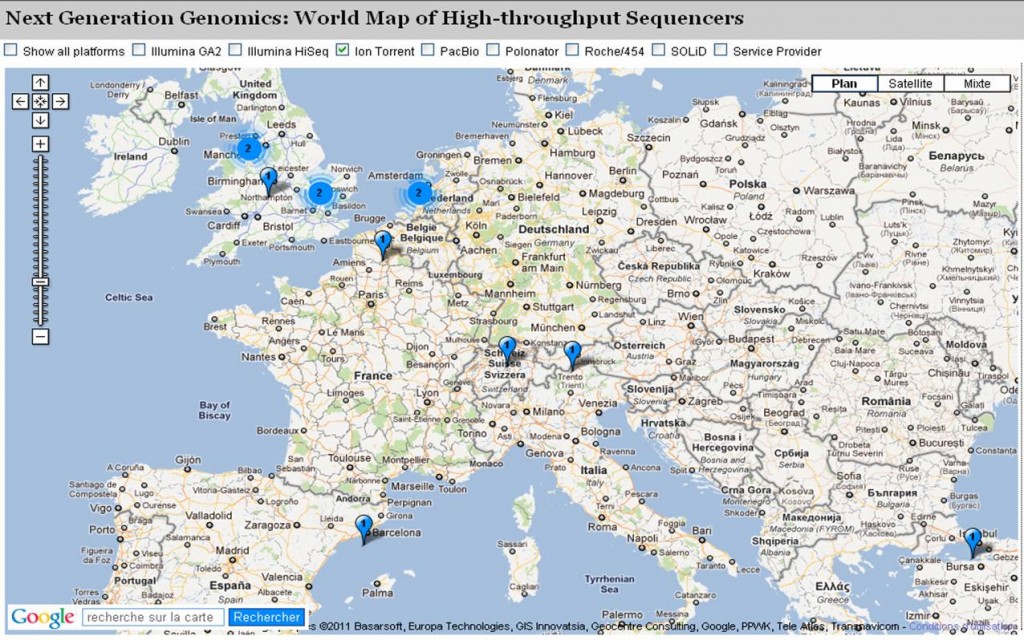
Il s’agit d’une application rendue possible grâce à Google Map et hébergée sur un blog du Pallen Research Group. Cette application permet de connaître (en partie, car il s’agit d’un recensement sur déclaration volontaire des utilisateurs) des lieux où sont implantées les différentes technologies de séquençage haut-débit.
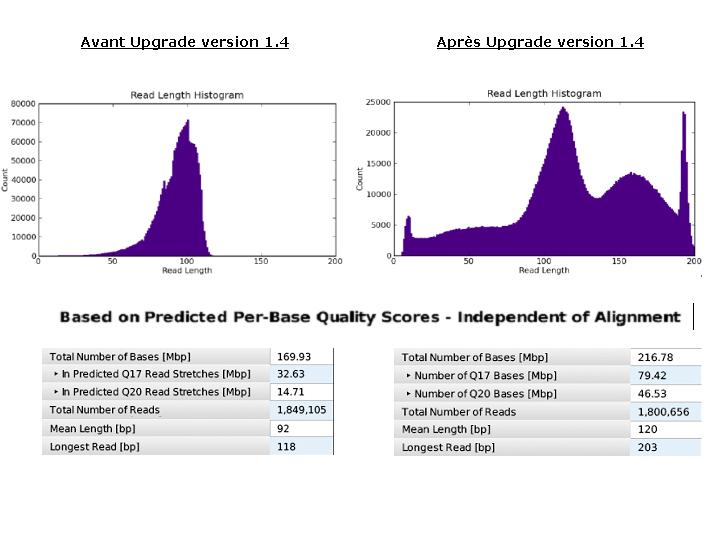
L ’évolution de la technologie Ion torrent permet depuis ces derniers jours, d’accéder au format de puce « 316 » succédant à la « 314 ». Ces puces multiplient par 10 la quantité de données générées à l’issue d’un « run » de séquençage aboutissant ainsi à 100 Mb.
De manière à bénéficier de la capacité totale de ces puces, une mise à jour du serveur, ainsi qu’une configuration du PGM en version 1.4 sont nécessaires et ont été réalisées ce jour. Ces modifications se traduisent par une amélioration de l’algorithme d’analyse (basecalling, alignement, …) optimisant le nombre de reads considérées et traitées. Le développement apporté au niveau des « runs » permet désormais d’intégrer des « reads » de plus grande taille (jusqu’à 200b environ) directement lié à une amélioration de la précision de séquençage.
Techniquement, quelques modifications au niveau du PGM ont conduit au remplacement de certaines tubulures (diamètre supérieur aux précédentes) : Cet épisode fut l’occasion de découvrir les entrailles de la machine…
Depuis une petite dizaine d’années, une partie du monde de la recherche s’indigne souvent en silence contre un outil de mesure : les principes de l’évaluation scientifique.
Le pouvoir politique français garant du bon usage des fonds publics alloués à la recherche a calqué son appareil de mesure sur ce qui a déjà cours dans le monde anglo-saxon (l’AERES s’est inspiré notamment de l’ERIH, l’European Reference Index for the Humanities). Ainsi, ont fleuri un grand nombre de facteurs dont l’objectif est d’objectiver l’impact d’une recherche sur une société qui la finance ou plutôt d’évaluer la diffusion d’une innovation, d’une découverte (et non son impact réel sur la société).
Ci-dessous un exemple des items tirés de la « fiche de données bibliométriques » qu’un chercheur remplit en vue de satisfaire aux exigences lui permettant de voir sa carrière progresser (dossiers de contractualisation de la vague C, CNRS):
– Nombre d’articles réf Web of Science sur 2004-2007
– Nombre total d’articles réf. Web of Science, Npa
– Nombre total de citations Nc,totb
– Nombre total de citations hors autocitations
– Nombre de citations par articles Nc,tot/Npd
– Facteur h
– Facteur h relatif, h/Npf
– Facteur d’impact max. de la discipline
– Facteur d’impact moyen de la discipline
– Facteur d’impact max. pour le chercheur
– Facteur d’impact moyen pour le chercheur
Pour paraphraser un chercheur imminent, Pierre Bourdieu, parmi les bons élèves d’une classe il y a ceux qui ont compris comment les notes sont décernées et ceux qui ont compris…
Cet outil de mesure certainement nécessaire peut apparaître comme critiquable à plusieurs titres :
– l’instrument de mesure peut modifier la variable qu’il tente de mesurer (ainsi des pratiques telles que le « name-droping » des auteurs associés à une publication, les citations de convenances etc.)
– l’évaluation telle qu’elle est menée engendre des modes à l’instar de ce que relate la nouvelle de Léo Szilard, La fondation Mark Gable. Léo Szilard s’amusait des solutions qui engendreraient un retard du progrès scientifique… le présent peut par certains égards supplanter son ironie.
– ce type d’évaluation basée sur la bibliométrie engendre que « l’objectif principal des scientifiques n’est plus de faire des découvertes mais de publier autant que possible», de sorte que « l’utilité, la qualité et l’objectivité des articles se sont dégradées », c’est ce que de manière synthétique l’article du Monde Diplomatique, (comment devenir le chercheur du mois) tend à démontrer.
Par dessus tout, l’évaluation induit la mise en concurrence systématique de tous contre tous. Cette conscience même de la mise en concurrence entre équipes travaillant sur une même thématique incite ces mêmes équipes à attirer vers elles les fonds alloués à la recherche pendant que d’autres tentent de justifier leur propre existence… autant d’énergie qui n’est pas consacrée à la recherche en tant que telle : beaucoup de temps pour remplir des formulaires, beaucoup de temps pour communiquer, de moins en moins de temps pour réfléchir, développer et réaliser le travail de fond…
En définitive, le bon élève assimile et critique le système mais peut rapidement finir par exploiter les vices de l’outil de mesure : la notation. Ayant compris, il se plie aux exigences des critères de la recherche d’excellence… il remplit des formulaires, cherche comment il peut financer sa recherche, en n’oubliant pas les mots clés attendus. Pour prétendre à ces financements, il lui faudra justifier d’une certaine visibilité donc publier autant que possible… la publication devient l’alpha et l’oméga du chercheur… Ceci ayant pour effet, la dilution de l’information… et donc une perte de temps à rechercher l’information réellement pertinente.
Le constat peut paraître sans nuances mais il convient de l’inscrire dans une évolution du monde où la justification prend le pas sur la réflexion et l’action. Même s’il apparaît nécessaire d’évaluer ce que deviennent les fonds publics, il semble qu’un mauvais outil de mesure peut pervertir tout un système. C’est ainsi que l’on se gargarise à l’envi des terminologies novlangues où « l’excellence » remplace la science.
« Evaluer tue » – logo retravaillé de : http://forumpsy.wordpress.com/
L’accroissement des capacités technologiques en biologie moléculaire à pour conséquence la possibilité de séquencer/génotyper mais aussi stocker toujours plus de données constituant une manne d’informations sans limite pour qui sait en extraire du sens. C’est le travail du bioinformaticien de proposer à la fois un support technique et analytique au biologiste en même temps que la capacité en terme de ressources informatiques pour répondre aux besoins.
Ce premier post sur la place du bioinformaticien se focalise sur l’interaction avec le biologiste au sein d’un projet.
On peut dissocier un projet en trois phases :
– La préparation : identifier la question biologique, étudier la faisabilité et les coûts, mettre en place la méthodologie…
– La réalisation : génération des données biologiques et analyse bioinformatique
– La finalisation : interprétation des résultats, mise en production ou introduction d’une nouvelle question biologique
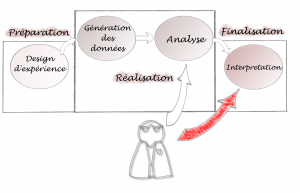
Le schéma ci-dessous montre un cheminement possible de l’intervention bioinformatique :
L’intervention du bioinformaticien est ici minimisée : il n’intervient qu’à partir de la phase analytique et dans l’interprétation des résultats (pour aider le biologiste à critiquer les résultats générés). Ceci pose plusieurs problèmes : les outils bioinformatiques doivent s’adapter, à tout prix, à la méthodologie mise en place précédemment. Si on constate que les méthodes d’analyses doivent être modifiées une fois la génération de données effectuées, la bioinformatique peut devenir ce qu’on appelle communément un goulot d’étranglement dans le projet…
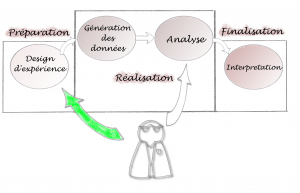
Au contraire, Le schéma ci-dessous nous parait plus judicieux :
Ici, l’intervention bioinformatique est optimisée : le bioinformaticien est totalement impliqué dans la préparation du projet. Et, au contraire, en fin de projet, les interventions sont minimisées car la méthodologie mise en place est adaptée pour une bonne compréhension par le biologiste. L’idéal et de fournir les outils nécessaires pour que le biologistes puisse lui-même exécuter ses analyses.
Pour résumer, l’ interaction doit se faire en amont et en aval de l’analyse et l’on se doit de minimiser les interventions pendant la phase d’analyse. En aval pour conseiller sur les méthodologies à utiliser, en amont pour contrôler les erreurs éventuelles rencontrées pendant la phase d’analyse (exécuter par le biologiste).
Aujourd’hui (20 juillet 2011) un graphiste de Google (c’est l’été, il s’agit certainement d’un stagiaire féru d’histoire des sciences) s’amuse à commémorer le 189 ème anniversaire de la naissance d’un moine morave qui reste comme le père fondateur de la génétique…
« Mendel est un contemporain de Charles Darwin ; ce dernier, qui avait mis en évidence le rôle du milieu dans la sélection naturelle et avait postulé l’existence d’importantes variations individuelles au sein d’une même espèce ainsi que leur rôle clé dans le processus évolutif, considérait comme une grande énigme la nature de ces variations ainsi que leur mécanisme d’apparition. Le naturaliste britannique, qui avait eu connaissance des travaux de Mendel, ne leur accorda pourtant aucune attention à l’époque. C’est ainsi que les deux théories, celle de l’évolution et celle de la génétique, qui allaient par la suite se compléter et s’enrichir mutuellement, coexistèrent séparément durant plusieurs décennies, sans que personne n’ait l’idée de faire le lien entre elle »
-extrait de wikipedia
Encore une collaboration qui n’a pas eu lieu !!!
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages