L’effervescence médiatique liée au séquençage de la bactérie Escherichia coli responsable de l’intoxication d’une dizaine de personnes dans le Nord de la France (juin 2011), a engendré de multiples reportages et communiqués. Parmi eux, celui de l’AFP, et ce paragraphe comme point de départ à cet article :
« L’Institut est équipé depuis un mois d’un séquenceur « à haut débit » qui permet de déchiffrer un génome en un temps record à partir d’une « librairie » d’ADN stockée sur une sorte de puce électronique. Cette technologie est notamment utilisée dans la recherche sur le cancer et pourrait remplacer dans un futur proche l’amniosynthèse (sic). »
En France, deux méthodes « invasives » permettent de réaliser un diagnostic prénatal : L’amniocentèse ou prélèvement du liquide amniotique, et une biopsie de trophoblastes ou prélèvement des cellules du futur placenta.
Toutefois, cet acte cause chaque année environ 1% de fausses couches. Rapporté aux 90000 amniocentèses par an, on déplore jusqu’à 900 pertes de fœtus, qui paradoxalement sont sain dans la majorité des cas.
Ces conséquences iatrogènes sont terribles sur le plan humain et il apparait évident de mettre au point une méthode beaucoup moins dangereuse pour la mère et surtout pour le fœtus.
Depuis plusieurs années, il a été démontré la présence de cellules d’origine fœtales dans le sang maternel dès la huitième semaine de grossesse. Cette particularité a dernièrement fait quelques émules et ont décidé de l’exploiter pour la mise au point d’un diagnostic prénatal « non invasif ». Le point critique repose sur la très faible proportion de ces cellules provenant du fœtus ; de l’ordre d’une pour dix millions de globules blancs et cinq milliards de glo¬bules rouges par millilitre de sang maternelle.
L’une des méthodes les plus encourageantes, réalisée par l’équipe de Patrizia Paterlini-Bréchot, Unité Inserm U807, repose sur une méthode d’enrichissement par la taille des cellules trophoblastique dans le sang, dite « ISET » ( Isolation by Size of Epithelial Tumor / Trophoblastic cells ), leur caractérisation par la technique des empreintes génétiques, puis séquençage de l’ADN extrait. La société « Rarecells » a par ailleurs était créée sur cette base.
Cette méthode sensible et spécifique à 100%, a déjà été validée techniquement et cliniquement pour le diagnostic prénatal non invasif de l’amyotrophie spinale (Beroud C., Karliova M., Bonnefont J.P., et al. Prenatal diagnosis of spinal muscular atrophy by genetic analysis of circulating fetal cells Lancet 2003 ; 361 (9362) : 1013-1014) et de la mucoviscidose ( « Recherche de la mutation du gène CFTR » Saker A., Benachi A., Bonnefont J.P., et al. Genetic characterisation of circulating fetal cells allows non-invasive prenatal diagnosis of cystic fibrosis Prenat Diagn 2006 ; 26 (10) : 906-916) , et techniquement seulement pour la trisomie 21.
Une seconde approche, réalisée par des chercheurs de l’Université de Stanford, est basée sur l’étude de l’ADN fœtal libre extrait du sang maternel puis analysé grâce aux séquenceurs à haut débit.
Dans le cas de la trisomie 21 par exemple, l’assignement de plusieurs dizaines de milliers de séquences présenterait un déséquilibre statistique même si la proportion d’ADN fœtal par rapport à l’ADN maternel est extrêmement faible (Noninvasive diagnosis of fetal aneuploidy by shotgun sequencing DNA from maternal blood. Proceedings of the National Science Academy of the USA, 2008, 105, 16266–71). Si cette approche semble très attractive, plusieurs interrogations se posent suite à la lecture du travail scientifique, à la fois sur la méthodologie, l’analyse statistique et l’évaluation des résultats. (Commentaires sur les études de Fan et al.)
Dans l’éventualité d’un diagnostic basé sur le séquençage complet d’un génome, ou davantage ciblé tel l’ « amplicon sequencing » des gènes d’intérêts, la méthode non invasive s’appuyant sur une analyse globale du sang maternel nécessiterait une profondeur de séquençage conséquente. Ceci aurait pour incidence une orientation technique tournée vers des appareils de type « Solid 5500 » ou encore « Illumina HiSeq 2000 », cracheurs de « reads ».
Dans le cadre d’une application diagnostique, l’investissement financier est l’un des aspects considéré et en ce sens la méthode « ISET », plus contraignante dans la préparation du matériel biologique, permets néanmoins d’accéder, dans la perspective d’un séquençage ciblé, aux séquenceurs de paillasse, cinq à dix fois moins cher à l’achat et à des coûts de « runs » inégalés à ce jour. Record actuellement détenu par le PGM, Ion torrent dont la capacité des puces actuelles de 100Mb et d’1Gb à venir d’ici la fin d’année 2011, permettrait un multiplexage soit d’individus pour une mutation donnée, ou encore, imaginer une liste de séquences d’intérêts à diagnostiquer par individu.
Il y a fort à parier que le séquençage à haut débit finisse par devenir un outil incontournable dans le monde du diagnostic prénatal d’autant plus que les coûts d’équipement et de fonctionnement ne cessent de diminuer. Dans cette perspective, l’évolution vers les séquenceurs haut-débit de 3ème génération (à partir de cellule unique) pourrait faire la part belle à l’utilisation des cellules trophoblastiques.
Au travers de ces avancées et de l’ère du séquençage à haut débit, les perspectives d’un diagnostic prénatal non invasif semblent se dessiner et laisse entrevoir un énorme soulagement. Aussi, la possibilité de mettre en place des plates-formes permettant de cribler l’essentiel des pathologies génétiques est envisagée.
Aussitôt, les détracteurs soulèvent la notion d’eugénisme, dénonçant un diagnostic prénatal intégral en vue d’une « traque » des individus « non conformes ».
Il apparait évident que ces avancées technologiques risquent très prochainement de soulever de réels débats de société…
Depuis 2005, le séquençage haut débit, comme son nom l’indique, a permis d’accroître la quantité de séquences produites par unité de temps, d’individu et de machine. Si intrinsèquement le terme de révolution est associé à ce type de technologies, il semble qu’il serait plus indiqué de l’associer aux nouvelles approches qui en découlent.
En effet, le séquençage nouvelle génération permet d’aborder des études sous de nouveaux angles d’approches. Bien souvent ces approches existaient pour la plupart avant l’avènement de ces nouvelles machines mais leur mise en œuvre étaient bien souvent laborieuses, coûteuses. Beaucoup de techniques nécessitaient des a priori techniques ou scientifiques (des a priori dus à la sélection et aux designs de sondes déposées ou synthétisées sur un support solide dans le cas des puces à ADN permettant les études transcriptomiques). Les nouvelles méthodes de séquençage, quant à elles, permettent de lever certaines anticipations expérimentales. Ainsi une étude du niveau de modulation des transcrits peut grâce à l’emploi de ces technologies en découvrir de nouveaux, ce que ne permet pas ce même type d’études sur puces à ADN. En outre tout a priori constitue un biais expérimental potentiel.
Pour résumer, un peu simplement, le séquençage haut débit dépasse l’outil analytique pour devenir une méthode exploratoire à part entière.
L’objectif de cet article est de proposer un bref aperçu du spectre d’applications et des champs d’expertises que ces nouvelles approches révolutionnent (nous reviendrons plus tard plus en détail sur certaines).
Concernant les applications ayant pour finalité les études génomiques, sont à distinguer :
– Séquençage de novo
Cette application découle de la quantité même de séquences que ces nouvelles générations de machines sont capables de générer. Aujourd’hui il est admis qu’un séquençage de novo nécessite une profondeur de 25 X, c’est-à-dire qu’il est possible de séquencer l’ADN d’un organisme procaryote en un run de séquençage sur la plupart des configurations de séquenceurs. Ce type d’applications a pleinement bénéficié des outils bio-informatiques au niveau des logiciels, machines et compétences humaines de plus en plus disponibles pour tenter de banaliser cette application. Ainsi, le centre de Shenzen avec le BGI (Beijing Genomics Institute, Chine) propose deux projets (pompeusement intitulés library of digital life) le premier consiste au séquençage (et reséquençage) de 1000 génomes de plantes et d’animaux et de 10 000 génomes microbiens.
– Découvertes de SNPs (Single Nucleotide Polymorphisms)
Cette application a très vite trouvé une application directe, elle a contribué au développement de puces à ADN de génotypage haut débit. Ainsi Illumina a pu produire en quelques mois des puces à ADN permettant le génotypage en parallèle d’environ 2,5 millions de SNPs par échantillon (un format 5 millions de SNPs est en préparation) en s’appuyant sur les résultats du consortium 1000 génomes qui a enrichi les bases de données en variations génétiques de la séquence consensus humaine. Ces outils permettant de réaliser des études d’association en réalisant des profils génétiques de plus en plus résolutifs.
Pour ce type d’application, deux modalités sont à distinguer :
– le reséquençage ciblé de zones d’intérêt, étape faisant suite, par exemple, à une étude d’association et permettant après reséquençage d’un locus génétique associé à un caractère particulier, de déterminer la causalité du phénotype différentiellement observé en terme de séquence.
– le réséquençage exhaustif d’ADN génomique. Cette modalité quant à elle, permet la mise en œuvre d’études de comparaisons génétiques de souches (telle que le permet la CGH (Comparative Genomic Hybridization) en s’affranchissant de toute hybridation grâce au séquençage direct)
Sous ce champ expérimental des études génomiques, peuvent être classées toutes les études de métagénomiques où un milieu cherche à être caractérisé le plus exhaustivement et finement possible par la diversité et le degré de contribution de chaque micro-organisme vivant (ou mort…) qui le compose. Le séquençage haut débit permet de rendre accessibles ce type d’approches.
Etudes transcriptomiques
Des méthodologies employées sur puces à ADN telles que les études transcriptomiques ont évolué et été adaptées sur les plateformes de séquençage haut-débit. En outre, ces nouvelles approches permettent de mesurer plus finement des niveaux de modulation tout en tenant compte des isoformes des transcrits. Encore une fois, le fait de séquencer permet de limiter les biais en comparaison de l’emploi de puce à ADN. La lecture plus directe s’affranchit des éléments de design de sondes, des phénomènes d’hybridation etc. A terme, les séquenceurs haut débit supplanteraient les puces à ADN pour ce qui concerne les applications d’études transcriptomiques.
– Réalisation de profils d’expression globale où l’intégralité du transcriptome cherche à être finement caractérisé pour une condition donnée.
– Caractérisation d’ARN non codant
Lors d’études de profils d’expression, les méthodes de séquençage haut débit permettent d’envisager la détermination et caractérisation des ARNs non codants (ici sont particulièrement visés les miRNA et smallRNA).
Etudes épigénétiques
– Etudes de la méthylation de l’ADN (méthyl-seq)
Les études de la méthylation de l’ADN génomique cherche à cartographier les loci fortement métyhylés dans une circonstance donnée. Pour rappel, une faible méthylation favorise la transcription mais une forte méthylation, au contraire, l’inhibe. Lorsque le promoteur d’un gène est méthylé, le gène en aval est réprimé et est donc plus difficilement ou pas du tout transcrit en ARNm.
– Etudes d’association protéines-ADN
Le ChIP-séquençage, également connu sous l’appellation de ChIP-Seq, est utilisé pour analyser des interactions protéines/ADN. Le ChIP-Seq combine immunoprécipitation des zones génétiques sur lesquelles se trouvent fixées des protéines (ChIP) avec le séquençage haut débit de l’ADN afin d’identifier des motifs consensus. Il peut être utilisé pour une cartographie précise de sites de liaison pour une protéine d’intérêt.
Ces deux dernières applications ont dans un premier temps été développées sur la base des tiling-arrays. Le séquençage haut débit permet de diminuer les coûts d’investigation tout en gagnant en sensibilité.
Des technologies émergentes permettent souvent d’envisager de nouvelles applications diagnostiques. Ainsi quelques études depuis 2008, (Fan et al., Noninvasive diagnosis of fetal aneuploidy by shotgun sequencing DNA from maternal blood. Proceedings of the National Science Academy of the USA, 2008, 105, 16266–71), semblent ouvrir la porte à un diagnostic prénatal non invasif.
La recherche médicale avec son débouché clinique de la médecine personnalisée entrevoit des applications au séquençage haut débit. Ainsi, une équipe, montre dans des résultats publiés dans Nature Genetics, pour la troisième fois au monde, la faisabilité d’une étude menée sur la base du séquençage de l’exome, aboutissant à la découverte d’une causalité génétique (simple, puisque monogénique). Cette mutation du gène NOTCH2 causalité d’une maladie rare, le syndrome de Hadju Cheney, une ostéoporose sévère, a été identifiée et caractérisée efficacement par l’application des techniques de séquençage haut débit. Il y a peu ce type d’identifications n’auraient pas été financées puisque trop longue à mener, trop coûteuses pour des retombées certainement perçues comme limitées. Pour beaucoup, ces études menées à grandes vitesses trouveront des applications concrètes dans le champ de la médecine personnalisée… mais de cela nous reparlerons.
Notre laboratoire possède un PGM IonTorrent de Life Technologies. Cet outil haut-débit est un séquenceur de paillasse permettant de réaliser des runs à prix relativement accessible, inférieur à 800€/run (cf. notre historique du séquençage proposé dans un post précédent).
Nous avons pu réaliser, en avant-première, un séquençage complet d’une souche d’Escherichia coli sur une puce 316 ,qui permet d’obtenir 100Mb / run, soit l’équivalent d’un peu plus d’une couverture de 20X d’une Escherichia coli.
Pour plus d’informations sur ce séquençage, vous pouvez lire:
– le communiqué de presse associé : CP_Escherichia_coli
-L’article de Futura Sciences sur le sujet : http://www.futura-sciences.com/fr/news/t/genetique-1/d/sequencage-haut-debit-de-coli-vers-une-medecine-personnalisee_31316/
– Des articles plus généralistes sortis dans différents journaux :
Ou regarder ces reportages (datés du 06/07/2011) :
Reportage-LCI-séquençage-coli-06072011
Reportage-BFMTV-séquençage-coli-06072011
Le procédé de séquençage se déroule sur 3 jours :
– préparation de la librairie : 1 jour (fragmentation de l’ADN génomique…)
– préparation de la matrice : 1 jour (PCR en émulsion + enrichissement)
-séquençage de la matrice enrichie : 1/2 jour
Et on obtient le report suivant : Report Run IonTorrent
(*) Sequencing data were generated using system software and protocols bothnon released and non supported by Ion TorrentTM (part of Life TechnologiesTM) and may not reflect actual Ion Torrent PGMTM performance in term of throughput and/or accuracy.
 L’œuvre d’art contemporain fait débat et pour le mener à bien il semble toujours possible d’exhumer Duchamp ou bien encore de profiter des écrits d’un Yves Michaud. Humblement, cet article est une évocation de ce qui peut être qualifié d’art, simplement parce qu’il en porte le nom : le bio art.
L’œuvre d’art contemporain fait débat et pour le mener à bien il semble toujours possible d’exhumer Duchamp ou bien encore de profiter des écrits d’un Yves Michaud. Humblement, cet article est une évocation de ce qui peut être qualifié d’art, simplement parce qu’il en porte le nom : le bio art.
Commençons par ce qui n’est pas du bio-art… l’art qui imite la nature (ou l’inverse).
Il est parfois délicat de qualifier les courants et orientations de l’art contemporain, il ne faut pas se méprendre : un arbuste taillé (même par un Edouard aux mains d’argent lui-même créature bio artistique) en forme de dinosaure par exemple ne bénéficierait pas de l’appellation de bio art, sinon que dire de la période art nouveau où la végétation des boiseries et ferronneries domestiquées par Victore Horta s’invitait en douces volutes dans les escaliers des maisons des années 1900. Ces derniers exemples nous placent toujours sous la sentence d’Aristote : l’art imite la nature.
Des artistes exploitent le vivant comme médium artistique mais plutôt dans un but contemplatif et du laisser faire, tel Michel Blazy et ses fameuses sculptures pourrissantes qui ont ravi ou dégoûté le public du palais de Tokyo en 2007.
Une des modalités du bio art consiste à exploiter les moyens analytiques scientifiques pour les ériger en œuvre d’art à part entière, en œuvres porteuses de sens, une forme de rencontre entre le numérique et l’organique. Jean Claude Ameisen avec une mise en parallèle d’une image tridimensionnelle d’un kyste de Pneumocystis carinii et du portrait tourmenté de Francis Bacon, nous propose une lecture ironique et lyrique en inversant les termes du dogme aristotélicien que l’art contemporain cherche à dépasser (ici la nature imite l’art).
Qu’est ce que le bio art…? Un art qui joue des oxymores en inventant une nature synthétique …
Le bio art (ou art biotech) est une forme de l’art contemporain qui modifie les processus de vie. Cette forme d’art utilise trois modalités possibles :
Le bio art cherche en grande partie à en créer une nouvelle (nature), telle un Monsanto de salon dirait ses détracteurs. L’art réalise la nature par l’intervention non divine de la science.
1) amener de la biomatière à des formes inertes ou à des comportements spécifiques. Beaucoup d’artistes travaillent sur la frontière entre naturel et artificiel, comme le mobile perpétuel à base photosynthétique d’Amy Franceschini et Michael Swaine.
2) détourner des outils et des processus biotechnologiques pour en réaliser une œuvre en soi (ou à soi…), ce point sera plus particulièrement abordé puisqu’il a trouvé une application commerciale.
3) inventer ou transformer des organismes vivants sans réels objectifs scientifiques ou industriels. Ainsi, Joe Davis, un artiste américain notamment affilié au MIT a encodé des textes en prenant les quatre bases A, T, G, C de l’ADN pour réimplanter ceci dans des organismes vivants. Il a encodé par exemple un fragment génétique codant une phrase d’Héraclite – » Le dieu dont l’oracle est à Delphes ne révèle pas, ne cache pas mais il signifie » dans le gène d’une drosophile (gène responsable de la vue chez cette mouche, cette modification n’altère pas le phénotype de l’animal).
Cette dernière approche de transformation du vivant est la plus radicale et a pour objectif déclaré de susciter un débat que la bio-éthique mène depuis les années 60.
Une œuvre marquante, troublante et génétiquement gênante a été réalisée par Eduardo Kac avec son projet de lapine transgénique, vert fluorescente appelée « Alba« , ce projet a pu être mené à bien par des techniques transgéniques utilisées dans l’art. Ce lagomorphe chimérique rendu fluorescent grâce à un gène de méduse devait provoquer un débat sur le statut des animaux transgéniques (de la transgression à la transgénèse, en somme…)
Quelques groupes ont émergé pour pousser l’investigation bio-artistique. Parmi quelques groupes de recherche institutionnels, deux d’entre eux symbiotica et bioteknica, réciproquement rattachés à l’université de Western Australia et à l’université de Québec à Montréal, proposent un grand nombre d’expositions et d’ouvrages plutôt bien accueillis par la communauté scientifique. Outre des biologistes et des plasticiens ce type de structures accueille des philosophes, sociologues et anthropologues afin de permettre l’analyse des interactions entre les biotechnologies et la société, entre l’art et la biologie, l’Homme et son environnement…
L’innovation et la recherche du spectaculaire qui a aussi cours dans le monde des sciences et notamment dans le monde de biologie cellulaire et moléculaire (dont rappelons le, grand nombre de techniques cherchent à montrer de manière indirecte ce qui demeure invisible) devient un médium que le bio art exploite pour des fins tant esthétiques qu’égotistiques. En effet, au carrefour entre le bio art, la biologie moléculaire et Ikea, ont fleuri des marchants d’œuvre d’art relativement bon marché proposant votre propre emprunte ADN (détournant ainsi les techniques de « fingerprinting » moléculaire) imprimée qui deviendra ornement d’intérieur, un vernis commercial transformant le bio art en « ego art ».
Ainsi se sont développées sur internet des sociétés (telle que la société DNA 11, par exemple) proposant de mettre en œuvre ce type de techniques qui désertent les laboratoires pour se banaliser dans un champ d’application inattendu.
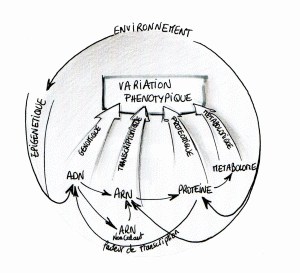
L’une des ambitions de la biologie intégrative est d’étudier le plus finement et le plus efficacement possible les interactions menant à une variation phénotypique. Ce réseau peut être d’une vertigineuse complexité, il demande l’intégration et l’analyse de données hétérogènes avant d’envisager toute interprétation.
Ces nouvelles approches exigent l’interdisciplinarité : la biologie moléculaire (biologie humide) doit être supportée par la bioinformatique (biologie sèche) afin de rendre réalisables des études de plus en plus ambitieuses, coûteuses et complexes.
Le schéma proposé ici permet d’entrevoir les interactions possibles entre génome, transcriptome et protéome… chaque interaction donne lieu à des méthodes, des techniques d’études particulières que nous tâcherons de développer au travers de ce blog. En guise de préambule nous vous proposons d’écouter une émission très didactique et plutôt exhaustive datant du 5 février 2011, « Sur les épaules de Darwin » de Jean Claude Ameisen.
Sur les épaules de Darwin – Jean Claude Ameisen
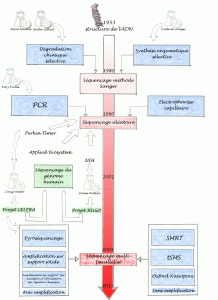
Un bref historique des évolutions du séquençage de l’ADN permet de comprendre ce que certains nomment révolution technologique… certainement concernant l’univers de la biologie moléculaire une innovation comparable à celle la PCR.
Allan Maxam , Walter Gilbert (USA) et Frederick Sanger (Royaume-Uni), les pionniers de la conquête de la séquence, ont mis au point deux méthodes très différentes permettant d’accéder à la lecture de la séquence. Maxam, Gilbert ont exploité des stratégies de dégradation chimique sélective quand Sanger choisit une stratégie de synthèse enzymatique sélective. Pour ces découvertes, Gilbert et Sanger ont été récompensés par le prix Nobel de chimie en 1980. L’histoire ne retient effectivement que Sanger, à juste titre, puisque cette stratégie bénéficiant de l’invention de laPCR (invention de Kary Mullis publiée en 1986, lui aussi nobélisé par le prix de chimie en 1993) et du développement de l’électrophorèse capillaire, permettant de simplifier la partie séparative et analytique. C’est ainsi que des sociétés comme Perkin Elmer, Beckman Coulter et Applied Biosystem ont investi le marché sur le principe même du séquençage Sanger sans le révolutionner mais en permettant son automatisation et l’augmentation du nombre de réactions de séquences analysables par jour. Une fois amorti le coût des séquenceurs, le coût de revient d’une séquence n’a cessé de baisser permettant de banaliser l’accès à la séquence.
Bien que se montrant de plus en plus performant, des applications comme le séquençage complet de génomes eucaryotes supérieurs, les approches métagénomiques (nécessité de cloner), les études de modulation de transcrits (la méthode SAGE les permettait sur la base du séquençage Sanger, cette méthode est lourde et nécessite toujours une phase de clonage) ont connu des limites quasi infranchissables (méthodes nécessitant trop de temps et de capitaux). Prenons à titre d’exemple les projets de séquençage du génome humain. Ces projets auront nécessité plus de 10 ans de travail et de 300 millions de USD pour le projet privé de Celera et un peu moins de 3 milliards de USD pour le projet HUGO (HUman Genome Organisation), le match entre Craig Venter (fondateur de Celera avec le soutien de Perkin Elmer) et James Watson (premier directeur du NIH coordinateur du consortium international) se solde par un match nul entaché de polémiques avec les publications en 2001 des premières séquences du génome humain.
L’année 2005 a connu à grands bruits, l’arrivée de nouvelles méthodes de séquençage à haut débit. Ces méthodes de séquençage massif font appel aux techniques de clonage et d’amplification moléculaire, leur spécificité relevait de leur stratégie de lecture.
En effet, la société 454 (rachetée par Roche en 2007), utilise des méthodes de pyroséquençage (luminescence par libération de pyrophosphate) de fragments d’ADN isolés dans des micro-gouttes comme micro-réacteur de PCR isolés au sein d’une émulsion, la société Solexa (rachetée par Illumina en 2007), utilise des méthodes d’amplification sur support solide permettant l’incorporation de bases terminateurs de chaîne réversibles marqués par des fluorochromes. La société Agencourt (rachetée par Applied Biosystem en 2006), a quant à elle basé son système de détection sur le principe de l’amplification par émulsion et hybridation-ligation chimique.
Depuis 2007, une certaine frénésie s’empare du monde de la biologie moléculaire, un très grand nombre de machines que l’on qualifie tour à tour de NGS (Next Generation Sequencing), HTS (High Throughput Sequencing), ou encore de manière plus appropriée sequençage multi-parallélisé se trouvent disponibles et évoluent vers le plus de profondeur, ou vers le moindre coût, rendant obsolètes les versions précédentes (la société Illumina à titre d’exemple depuis le rachat de Solexa a lancé un modèle de séquenceur tous les ans).
Cette révolution technologique est sortie des laboratoires pour attirer l’attention d’investisseurs et autres banquiers conscients qu’une ruée vers l’or accompagnant la ruée vers la séquence pouvait s’engager. Dans ce sens nous mettons à disposition un document datant de 2007 réalisé pour le compte de la Deutsche Bank (ce document très complet a été co-réalisé par nombre de consultants en biotechnologies, il permet de décortiquer assez finement les 3 technologies présentes sur le marché à partir de 2005 et de montrer les attentes des mondes scientifique et financier)
Afin de terminer ce bref tour d’horizon il semble nécessaire de hiérarchiser ces nouvelles technologies. Il est possible de distinguer tout d’abord deux grands groupes : les technologies sans amplification (permettant de séquencer une seule molécule d’adn matriciel, citons exemple de la technologie SMRT de Pacific Biosciences, la technologie tSMS de Helicos Biosciences ou encore celle toujours en développement mais très attendue développée par Oxford Nanopore) et avec amplification (toutes les technologies amplifiant une matrice clonale d’adn issu d’une librairie)
Parmi cette seconde classe, il est possible de distinguer trois grandes classes de matériel :
– les usines à produire de la séquence (type Hiseq 2000 de Illumina permettant de séquencer jusqu’à 200 Gb par run de 8 jours
– les séquenceur de paillasse (type PGM de IonTorrent de taille et prix beaucoup plus modeste permettant de réaliser des run autour de 100 Mb avec une perspective de 1 Gb pour début 2012)
– les séquenceurs haut débit en voie d’obsolescence (454 de Roche, les versions des séquenceurs Illumina antérieures au HiSeq 1000 etc.)
Cet historique est bien entendu un prétexte pour planter le décor et présenter les forces en présence. Après l’âge des pionniers (Maxam et Sanger), celui de la douce exploitation (séquenceur capillaire) vient celui de l’accélération que certains nomment révolution… ces technologies marquent quoi qu’il en soit un tournant pour la génomique et pour les applications actuelles et en devenir qui y sont liées. Enfin la diffusion de ces technologies va de paire avec un changement de physionomie des laboratoires : la biotechnologie est une science composite où bio-informatique, biologie cellulaire et moléculaire mais aussi physique et statistique deviennent de plus en plus interdépendantes.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages