Ce billet très court, pour faire l’annonce (un peu tardive) du début d’un MOOC. Ce dernier s’annonce excellent, traitant du séquençage de génomes complets bactériens.

Ce cours estival est proposé par l’excellente Université Technique du Danemark (DTU)
En voici le plan :
WEEK 1
Module 1
Welcome and introduction to typing of bacteria and use of Whole genome sequencing applied to surveillance of bacterial pathogens and antimicrobial resistance
3 vidéos
WEEK 2
Module 2
Introduction to Next Generation sequencing
3 vidéos
WEEK 3
Module 3
Whole genome sequencing tools- demonstration of analysis tools for species identification, MLST typing and finding resistance genes
3 vidéos
WEEK 4
Module 4
Whole genome sequencing tools- demonstration of analysis tools for Serotyping of Salmonella and Escherichia coli strains , and finding plasmid replicons
3 vidéos
WEEK 5
Module 5
Whole genome sequencing tools- demonstration of analysis tools for multiple analyzes, phylogenetic tree building and finding genetic markers from self-made databases and Summative Tutorial exercise
5 vidéos, 1 lecture
Noté: Tutorial final Quiz
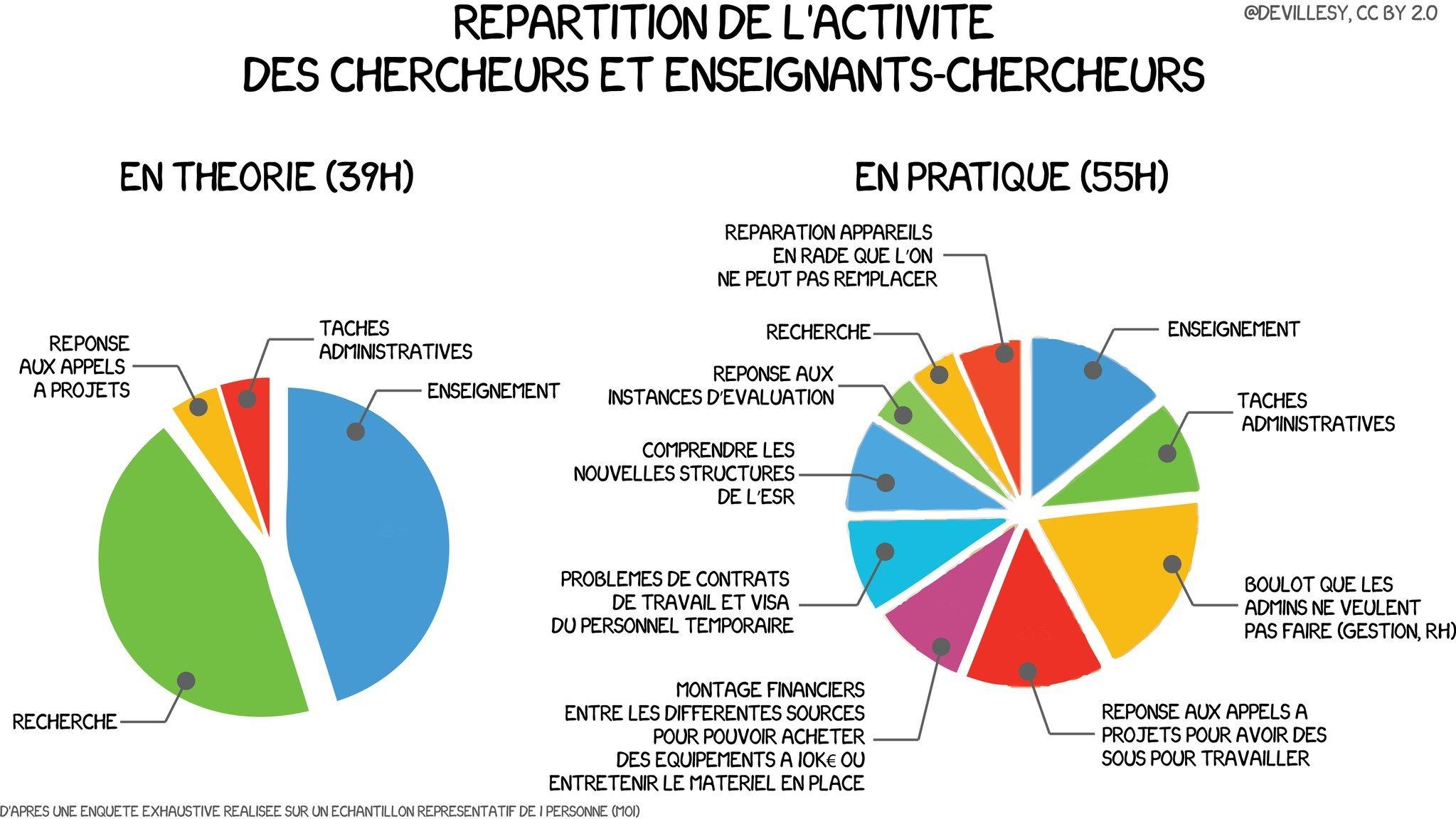
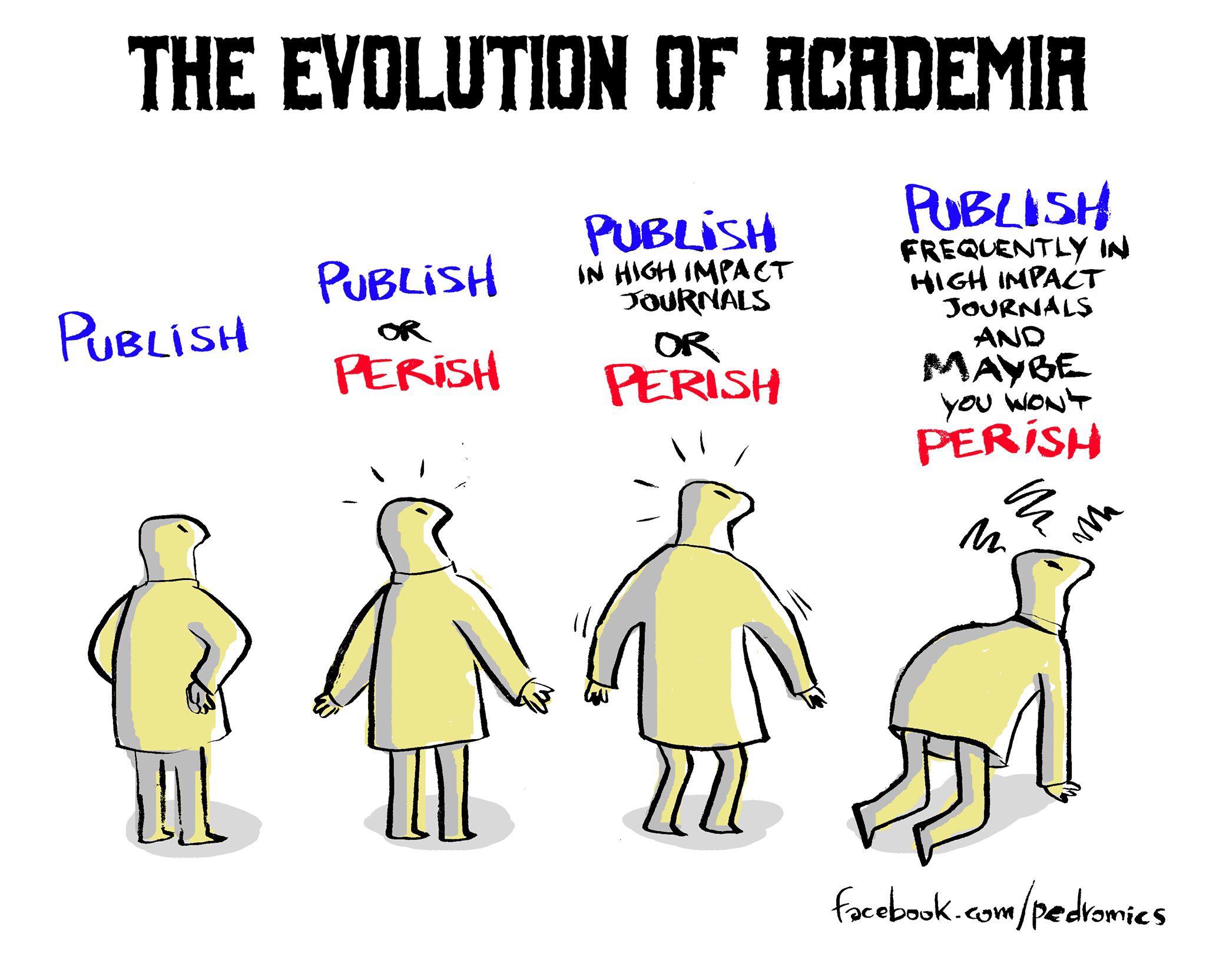
Avec humour et sans complètement tomber dans la caricature extrême, cette infographie met en lumière le travail protéiforme des chercheurs & enseignants-chercheurs, ces hybrides qui naviguent entre laboratoires et amphithéâtres. Plombés par des tâches administratives… il faut dire qu’ils sont bien aidés par du personnel administratif parfois un peu mono-tâche (à café) soucieux de faire coller son volume horaire pratique à la théorie, les chercheurs et enseignants-chercheurs sont les rares personnes à devoir chercher leur propre budget pour répondre à l’injonction d’accroître le corpus de connaissances, en étant tour à tour : évalués, évaluateur, éditeur, vulgarisateur, secrétaire, encadrant de stagiaire de thésards, professeur, RH…
Tout ceci m’a remis en mémoire, ce dessin de Charb issu des cahiers pédagogiques…
 Pour avoir connu moult galères dans la quête d’un jeu de données (en l’occurrence issu de Ion Torrent) afin de tester des versions de pipelines analytiques ou pour optimiser les paramètres de ceux-ci, nous avons décidé de publier un jeu de données, les résultats en sortie de pipeline et les conclusions statistiques puis biologiques qui en découlent. Avant, dans et après la moulinette bio-informatique.
Pour avoir connu moult galères dans la quête d’un jeu de données (en l’occurrence issu de Ion Torrent) afin de tester des versions de pipelines analytiques ou pour optimiser les paramètres de ceux-ci, nous avons décidé de publier un jeu de données, les résultats en sortie de pipeline et les conclusions statistiques puis biologiques qui en découlent. Avant, dans et après la moulinette bio-informatique.
En espérant, que ceci puisse éviter des galères à d’autres… :
Targeted metagenomic sequencing data of human gut microbiota associated with Blastocystis colonization
Concernant l’exploitation « biologique » de ce jeu de données, l’article publié dans Scientific Data est rattaché à celui-ci :
Colonization with the enteric protozoa Blastocystis is associated with increased diversity of human gut bacterial microbiota
Voici une illustration, sous forme d’une caricature, de l’évolution des injonctions ressenties par les chercheurs académiques… la course à l’excellence et la quête du facteur d’impact comme ligne d’horizon.
 Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?
Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?

En juillet 2017, Stockholm accueillera la 3ème conférence annuelle « Metagenomics and Metadesign of Subways and Urban Biomes (MetaSUB)« , qui rassemblera des chercheurs dédiés à la cartographie du métagénome urbain de plus de 67 villes à travers le monde (dont Paris et Marseille). Ce projet ambitieux a débuté il y a deux ans, alors que le professeur Christopher Mason et son équipe ont réalisé la première étude sur la microflore de surface, dressant le microbiome de la ville de New York. Avec ces données moléculaires essentiellement basées sur le séquençage haut-débit 16S, le projet a étudié la façon dont ce «microbiome urbain» change avec des variables telles que la météo, la propreté, les matériaux de construction et même les niveaux socio-économiques du quartier. L’équipe a cherché à établir des profils de microbiotes dans le métro, à identifier les bio-menaces potentielles et à fournir des données complémentaires qui peut être utilisé par la ville pour créer une «ville intelligente», c’est-à-dire qui agrège des données hétérogènes pour améliorer son urbanisme.
Près de la moitié (48%) de l’ADN séquencé ne correspondait pas aux organismes connus, soulignant qu’un microbiota incognita entoure les usagers du métro. Le projet du métro de New York n’était que le début, ce qui a conduit à la création d’un consortium international de laboratoires pour établir une « cartographie » mondiale de microbiomes dans les systèmes de transport en commun notamment. En cliquant sur la capture d’écran ci-dessous, on pourra se faire une idée de la communication « Metasubienne » qui n’a rien à envier à celle de Tara Oceans ou de MetaHit (qui sont toutes trois, d’excellentes communications autour d’un projet et d’un consortium).
Voici en quelques mots les promesses annoncées dans l’article Geospatial Resolution of Human and Bacterial Diversity with City-Scale Metagenomics (Cell Syst, 2016) :
« La région métropolitaine de New York City (NYC) est un endroit idéal pour entreprendre une étude métagénomique à grande échelle car c’est la plus grande et la plus dense des États-Unis; 8,2 millions de personnes vivent sur une masse continentale de seulement 755 km2. En outre, le métro de New York est le plus grand système de transport en commun dans le monde (par le nombre de stations), qui s’étend sur plus de 406 km et utilisé par 1,7 milliard de personnes par an. Ce vaste écosystème urbain est une ressource précieuse qui nécessite un suivi pour le maintenir et le sécuriser contre les actes de bioterrorisme, les perturbations environnementales ou les épidémies. Ainsi, nous avons cherché à caractériser le métagénome de NYC en examinant le matériel génétique des microorganismes et d’autres ADN présents dans, autour et au-dessous de New York, en mettant l’accent sur les métros et les zones publiques très empruntés. Nous envisageons cela comme une première étape vers l’identification des menaces biologiques potentielles, la protection de la santé des New-Yorkais et la mise à disposition de données moléculaires qui pourront être utilisée par la ville pour créer une «ville intelligente», c’est-à-dire celle qui utilise des données de grande dimension pour améliorer l’urbanisme, la gestion de l’environnement bâti, des transports en commun et de la santé humaine. »
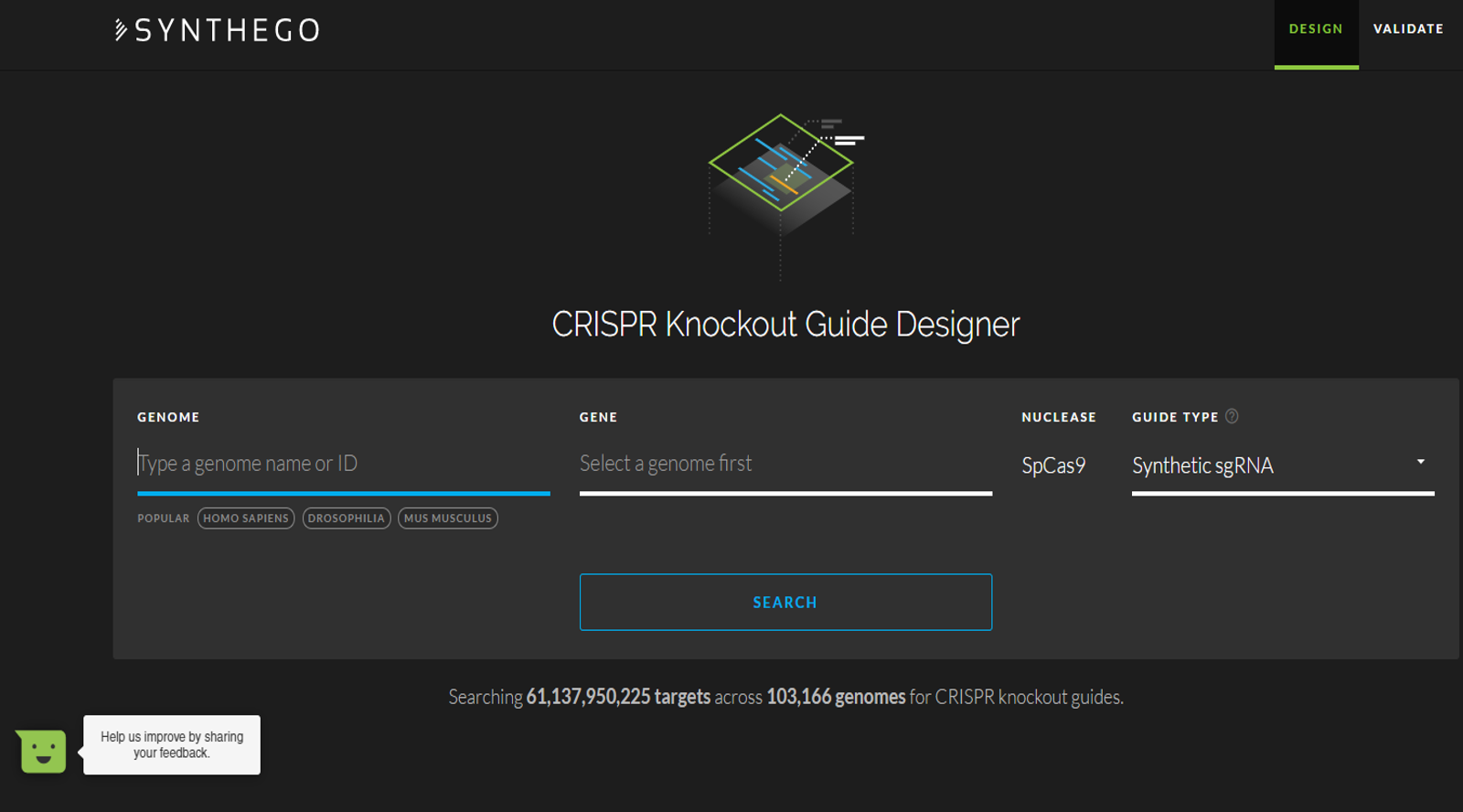
Voici un outil simplissime, rapide, en ligne, permettant de designer et de qualifier informatiquement des sgRNA, dans un contexte de KO réalisé par CRISPR/Cas9.
Vraiment pas mal !
Cliquez sur la capture d’écran ci-dessous et tentez votre design.
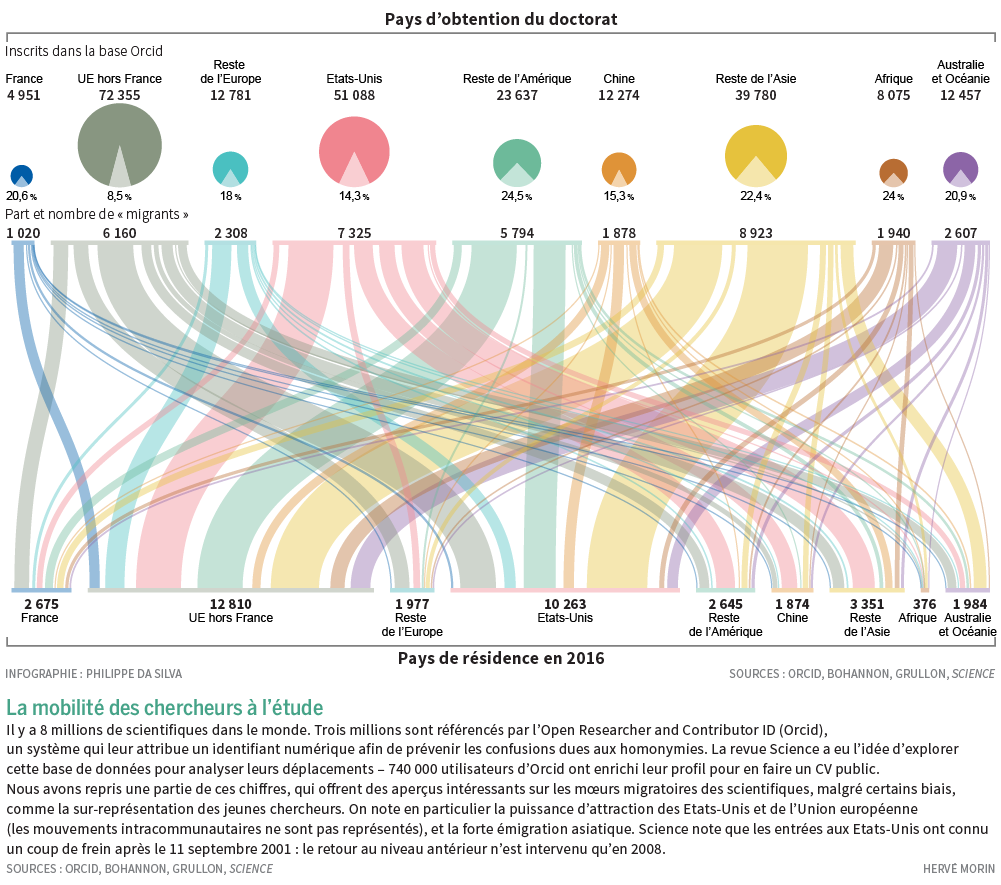
 Voici une infographie tirée du Monde Science et Techno du 23/05/2017. Celle-ci a été permise grâce au numéro ORCID qui permet d’identifier, de façon non ambiguë tout chercheur contributeur d’une publication scientifique. Selon l’Open Researcher and Contributor ID, « ORCID est une organisation à but non lucratif qui a pour objectif d’aider à créer un monde dans lequel tous les intervenants dans les domaines de la recherche, de l’université et de l’innovation sont identifiés de manière unique et sont reliés à leurs contributions et à leurs affiliations, au-delà des limites des disciplines, des frontières et des époques. » Concernant les migrations de chercheurs, l’Union Européenne et les Etat-Unis restent attractifs. En France, un cinquième des chercheurs ayant obtenu leur diplôme sur le territoire, émigre, ce qui est plutôt beaucoup… une question peut se poser : est ce que la proportion de chercheurs quittant leur pays d’obtention de doctorat ne serait pas inversement proportionnelle à l’intérêt de ce pays pour sa propre recherche scientifique…?
Voici une infographie tirée du Monde Science et Techno du 23/05/2017. Celle-ci a été permise grâce au numéro ORCID qui permet d’identifier, de façon non ambiguë tout chercheur contributeur d’une publication scientifique. Selon l’Open Researcher and Contributor ID, « ORCID est une organisation à but non lucratif qui a pour objectif d’aider à créer un monde dans lequel tous les intervenants dans les domaines de la recherche, de l’université et de l’innovation sont identifiés de manière unique et sont reliés à leurs contributions et à leurs affiliations, au-delà des limites des disciplines, des frontières et des époques. » Concernant les migrations de chercheurs, l’Union Européenne et les Etat-Unis restent attractifs. En France, un cinquième des chercheurs ayant obtenu leur diplôme sur le territoire, émigre, ce qui est plutôt beaucoup… une question peut se poser : est ce que la proportion de chercheurs quittant leur pays d’obtention de doctorat ne serait pas inversement proportionnelle à l’intérêt de ce pays pour sa propre recherche scientifique…?
Ce « marqueur » – le taux d’émigration des chercheurs diplômés du pays qu’ils quittent- peut, en même temps, être lié à une bonne santé d’un système éducatif et de formation.
En effet, plus les étudiants diplômés seront perçus comme bien formés plus ils auront de facilités à le quitter, attirés par des pays dont la recherche est plus dynamique (entendons par là des pays rémunérant mieux, offrant de meilleurs capacités d’accueil) que celui dans lequel ils ont obtenu leur diplôme. En définitive, cette proportion, ce marqueur peuvent en quelque sorte être liés à une distorsion entre deux capacités pour un pays : celui de former et celui de réaliser une recherche de haut niveau permettant de garder les chercheurs que ce même pays a formé.
 Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
ONT a fourni, en ce début de mois de mai 2017, informations concernant les développements technologiques et les perspectives de commercialisation. L’une d’elles, concerne une nouvelle « flow cell » pour MinIon, appelée Flongle (Flow Cell Dongle) pour les applications de diagnostics cliniques. Le développement du Flongle aidera également le travail de l’entreprise sur SmidgION, un séquenceur miniature avec de petites flow cells alimentées par un téléphone mobile (voir la photo ci-dessous).
Clive Brown, responsable technologique d’ONT, a donc présenté diverses mises à jour lors du « London Calling 2017 », le grand barnum des utilisateurs de la technologie, en ce début de mois de mai. En ce qui concerne le séquenceur MinION, Brown a déclaré que la technologie est d’ores et déjà capable de délivrer plus de 20 Gb de données par run de 48 h. (Actuellement, les utilisateurs sont plus autour des 15 Gb, ce rendement plus limité serait partiellement lié à la préparation de la bibliothèque, en particulier la quantification appropriée de la taille et de la quantité d’ADN de départ, selon Brown).

En mars, la société avait parlé d’une nouvelle méthode de séquençage, appelée séquençage 1D2, où les deux brins d’une librairie bicaténaire sont poussés par nanopore séquentiellement sans être physiquement connectés. La méthode contourne un brevet détenu par Pacific Biosciences et aboutit à des lectures plus précises que la seule lecture 1D, où seul un brin de la librairie constituée est séquencé. ONT prévoit de publier le kit de séquençage 1D2 courant mois de mai 2017, ainsi qu’une nouvelle chimie appelée R9.5. Les kits 2D ne sont plus disponibles et l’entreprise vient juste d’interrompre les anciennes cellules R9.4.
Dans l’ensemble, la précision de lecture brute est maintenant supérieure à 90 % pour la chimie R9.4 et supérieure à 95 % pour la R9.5 (en mode 1D2), ces deux chimies tournant à 450 bases / min.
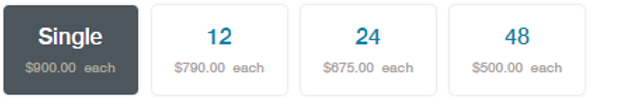
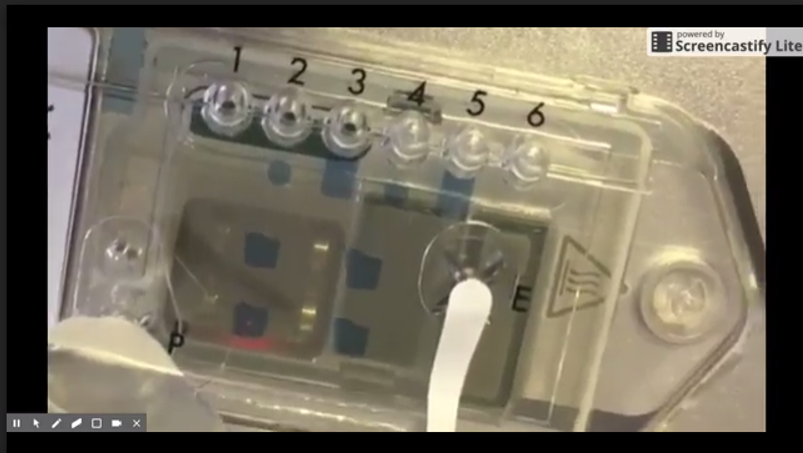
Ci-dessus, les prix actuels de la chimie 9.5. Là où l’on constate que le prix décroit drastiquement avec la quantité commandée… je pense qu’une mutualisation ou la création d’une centrale d’achats s’imposent ! Avec cette nouvelle chimie,ONT a diffusé une nouvelle version, la 1.6, de son logiciel MinKnow ainsi que de son basecaller Albacore (maintenant en 1.1). En outre, ONT a récemment commercialisé des kits de séquençage direct d’ARN et diffusé un pipeline pour réaliser le profil de résistance aux antibiotiques appelé ARMA (an analysis workflow for identification of antibiotic-resistant microorganisms in real time). À partir d’août, la société prévoit d’envoyer des flow cells à la température ambiante, des développements sont en cours pour allonger leur date limite d’exploitation (aujourd’hui une flow cell reçue doit être utilisée sous 8 semaines). Toujours visant le marché de l’utra-portabilité, en visant la décentralisation de l’acte de séquençage, ONT développe également un petit module de calcul pour le basecalling qui ferait environ la moitié de la taille du séquenceur MinION et pouvant y être directement connecté. Sur ce volet de la portabilité, le dispositif de préparation d’échantillons, VolTrax (voir vidéo ci-dessous), d’Oxford Nanopore est maintenant entre les mains de plus de 50 utilisateurs qui ont récemment reçu leurs premiers kits. La société développe actuellement un kit à base de transposase rapide et un kit d’indexation 4-plex rapide.
Les chercheurs de l’entreprise travaillent déjà sur une nouvelle version, VolTrax V2, qui devrait être disponible à la fin de 2017. Cette version permettra la PCR, la quantification des échantillons et le contrôle de la qualité des échantillons, tout en utilisant les mêmes aimants et appareils de chauffage que la version actuelle. Il sera également capable de gérer plus d’échantillons et d’exécuter des protocoles de préparation d’échantillons plus complexes. Il sera livré avec un chargeur de réactif avec des réactifs lyophilisés (lyophilisés, c’est mieux, parce que le séquenceur qui tient dans la main c’est bien, mais s’il vous faut un congélateur pour réaliser, sur le terrain, la moindre réaction…)
En mars, Oxford Nanopore a annoncé le lancement du GridIon X5, un séquenceur nanopore de bureau (et non plus de poche comme le MinION) pouvant permettre de travailler jusqu’à 5 flow cells à la fois et disposera d’un débit de 100 Gb par run de 48 heures. La plate-forme est livrée avec un cluster local de calcul haute performance qui permet le basecalling en temps réel ainsi que l’analyse des données. La première unité a été expédiée en début de semaine (en atteste la photo ci-dessous)

La société a récemment utilisé le GridIon pour séquencer un génome humain à 20 X, en utilisant 5 flow cells et la chimie R9.4 (en mode 1D).
Le GridION X5 permet d’exécuter simultanément ou individuellement jusqu’à cinq expériences; Les utilisateurs peuvent choisir d’utiliser tout ou partie de cette ressource à tout moment. La version actuelle de la chimie et du logiciel permet de générer jusqu’à 100 Gb de données pendant une exécution GridION X5 et le module de calcul peut analyser ces données en temps réel.
En utilisant la même technologie de base que le MinION et PromethION, le GridION X5 offre la possibilité de séquencer ADN et ARN en temps réel (dans la vidéo ci-dessous Clive Brown se transforme en Pipetman pour la promotion du GridION) :
Oxford Nanopore devait envoyer il y a peu les premiers consommables pour PromethION permettant de délivrer 50 Gb et pourraient en générer jusqu’à 120 Gb /jour dans un futur proche. En vitesse de croisière cette configuration sera capable de générer plus de données que le NovaSeq d’Illumina. Du lourd, du gros et du très petit : de quoi satisfaire les plateformes de séquençages et bientôt certainement des utilisateurs non touchés actuellement par la technologie aujourd’hui, puisque la version ci-dessous permet de préparer une librairie et de la séquencer avec quelques breloques qui tiennent dans le creux d’une main !

 Dans sa lettre d’opinion du 8 mai 2017, Declan Butler, journaliste à Nature, relaie le soulagement des scientifiques français qui se félicitent d’avoir échappé à une vague populiste synonyme de renfermement du pays. Rappelons qu’un grand nombre de scientifiques avaient appelé à voter contre Marine Le Pen, engagement plutôt rare, devoir de réserve oblige.
Dans sa lettre d’opinion du 8 mai 2017, Declan Butler, journaliste à Nature, relaie le soulagement des scientifiques français qui se félicitent d’avoir échappé à une vague populiste synonyme de renfermement du pays. Rappelons qu’un grand nombre de scientifiques avaient appelé à voter contre Marine Le Pen, engagement plutôt rare, devoir de réserve oblige.
Il faut dire que notre élection présidentielle arrivait dans un contexte très particulier, après le Brexit, après l’élection de Donald Trump, le tout sur une montée internationale des populistes et d’un euroscepticisme confinant souvent à l’europhobie caractérisée.
Après l’arrivée de Donald Trump, dans le bureau ovale, les scientifiques américains ont observé, avec désolation les coupes de 5,8 milliards $ du NIH (18 % de baisse par rapport au budget de 2017) pendant que le budget de la défense gonflait plus encore. Même si le Congrès aura son mot à dire et pondérera certainement les fougues anti-sciences du locataire de la maison blanche, le signal était fort d’un Trump vs Science, Trump n’aime pas la science et cette dernière le lui rend bien.
L’élection du 45ème président américain a engendré un mouvement March For Science que nous avons traduit en France par la « Marche pour la Science… » Il s’agissait d’aller vite, puisque beaucoup de scientifiques ont été touchés par la distorsion du réel et de la vérité, conséquence de tweets intempestifs d’un président nouvellement élu. Robert N. Proctor, historien des sciences à l’Université de Stanford, a déclaré que le March for Science était « sans précédent en termes d’échelle et d’ampleur au niveau de la communauté scientifique impliquée » [cette mobilisation s’est implantée dans] « une perception plus large d’une attaque massive contre les notions de vérité qui sont sacrées pour la communauté scientifique ».
Cette marche-là n’aura suscité que peu d’intérêt et de relais médiatique dans une France en voie d’élire son président. Les deux éléments à la racine de la mobilisation mondiale en faveur de la recherche scientifique ont eu comme épicentre:
- Le dénigrement de Donald qui traite de « fake news » tout fait scientifique allant contre des intérêts privés, économiques. Ainsi, il annonce la relance de l’industrie carbonée en traitant de mensonger tout argumentaire scientifique démontrant que la Terre se réchauffe du fait de l’activité humaine.
- La concrétisation de son projet de déshabiller la recherche (avec elle la santé et l’éducation) pour habiller l’armée… une caricature qui subit très bien l’épreuve des faits.
Le pouvoir de la vérité, contre la vérité du pouvoir…
Ce contexte permet de comprendre cette lettre de Nature expliquant que les scientifiques français se sont sentis soulagés, même s’ils n’ont pas eu réellement peur d’une arrivée à la tête du pouvoir exécutif d’un Trump français. Ils anticipent plus sur le résultat des élections législatives. En effet, derrière le soulagement, il y a cette peur que les valeurs de progrès, d’humanisme puissent être fragiles, en démocratie, face à la montée du populisme.
Vous pourrez accéder à la lettre, du 08 Mai 2017, en usant de votre souris ci-dessous :
Nous sommes soulagés, certains un peu plus arguant, certainement à juste titre, qu’Emmanuel Macron est un fervent promoteur de l’innovation. Au-delà de ce soulagement, la lettre de Nature conclut par une touche d’espoir et de fierté : « la victoire de Macron fournit de l’espoir, disent les scientifiques. « Avec tout ce que nous avons vu dans le monde récemment, depuis l’élection de Trump, jusqu’au Brexit et la montée de l’extrémisme en Europe, pour la première fois, nous avons un fort mouvement opposé à cela », explique Édouard Brézin, physicien émérite à la École Normale Supérieure à Paris et ancien président du CNRS. « Je suis heureux que ce mouvement vienne de la France… »«

Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages






Bioproduction: la France pas si mal que ça !
La carte obtenue par le LEEM – le syndicat du milieu pharmaceutique qui s’est substitué en 2002 au Syndicat national de l’industrie pharmaceutique (SNIP)- fait apparaître un regroupement de l’activité autour de quatre bassins principaux :
– la région Rhône-Alpes
– la vallée de la Seine
– l’Alsace
– le département du Nord
L’état des lieux de 2014 du LEEM est plutôt évocateur :
– La demande en biomédicaments est passée de 5 à 15 % entre 2000 et 2012.
– La France dispose notamment de capacités de production significatives dans le domaine des vaccins, des produits dérivés du sang, des hormones et des produits pour le diagnostic.
– 13 000 personnes sont employées dans la bioproduction
– Les formations françaises en biotechnologies sont reconnues au niveau international. Il en va de même pour le processus de reconversion des industries chimiques vers le vivant. Cette synergie de compétences est un atout.
Outre ces éléments visant à saluer une filière qui a su prendre un bon élan, d’un point de vue plus pratique, ce post est aussi l’occasion de fournir une liste de ces entreprises qui recherchent des profils plutôt jeunes et plutôt bien formés. Cette liste est évidemment non exhaustive et tirée de l’excellent recensement de l’Industrie Pharma Magazine.