 Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le principe assez simple du séquençage SBS (Sequencing By Synthesis) est en train de se décliner au sein de machines haut-débit plus accessibles en terme de coût… il s’agit là de l’arrivée sur le marché d’une nouvelle concurrence : des séquenceurs génériques tels que ceux que proposent le distributeur Azco Biotec, Inc. Ces séquenceurs viennent directement concurrencer ceux d’Illumina accompagnés de leurs réactifs « génériques » tels que ceux distribués par la société Proteigene…
Ces séquenceurs « génériques » ne révolutionnent rien, leur part de marché est actuellement marginale… les laboratoires académiques préfèrent souvent l’original à la copie, malgré tout la concrétisation de la « promesse » –qui l’a formulée au juste ?– d’un génome humain pour 100 $ d’ici 2015 passe par la large diffusion de machines dont la technologie a fait ses preuves… Le retard pris par les séquenceurs de 3ème génération laisse le champ libre à la 2ème. Le marché risque de se saturer en séquenceurs de paillasse (séquenceur de 2ème génération tels que le MiSeq d’Illumina et les PGM et Ion Proton de Life Technologies).
En pleine guerre froide des compagnies de biotechnologies, développeuses et vendeuses de matériel de séquençage… une fuite de documents est organisée. Le tout pour fournir aux personnes prescriptrices ou décideuses d’un futur achat de séquenceur, les arguments pour orienter ou justifier leur choix… parfois même leur mauvaise foi.

En cliquant sur la volute nucléique (depuis le 30/08/2012 – ce lien est censuré par Illumina- cf. commentaires de ce même article) ci-dessus vous aurez accès à un vrai-faux document confidentiel fourni par Illumina. Ce document assez bien réalisé a devancé celui de Life Technologies présenté dans notre article précédent. Il met l’accent sur le bénéfice, au niveau de la qualité des données générées, que procure un MiSeq (Illumina) par rapport à un PGM (Ion Torrent, Life Technologies)
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
Voici pour continuer sur les développements qui ont lieu sur la plateforme de séquençage de Ion Torrent, le PGM, une nouvelle note d’application qui ouvre de nouvelles perspectives dans le champ du séquençage haut-débit : le fait de pouvoir très bientôt séquencer des fragments de 400 bases dans les deux sens. Ainsi, le PGM de Ion Torrent sera la seule technologie capable de fournir sur des reads plutôt longs un séquençage paired end (sachant que le kit et protocole associé « mate paire » est d’ores et déjà disponible). Evidemment, ce développement vient contrecarrer une critique qui peut être portée sur cette plateforme de séquençage : sa « justesse » de séquençage sur séquences brutes générées à Q20 (une erreur générée pour 100 bases séquencées, qui dépasse en général Q50 sur séquence consensus, soit 1 erreur pour 100 000 bases), comparable cependant au taux d’erreur d’un 454 de chez Roche.
La note d’application, plutôt enthousiasmante, est très didactique (notamment pour évoquer les subtilités des différences entre séquençage piared end et mate pair) laisse entrevoir de grandes perspectives… seul bémol, le kit paired end ne serait disponible que dans le premier trimestre de 2012.
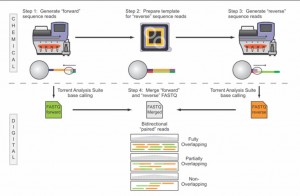
Le bien beau document que voilà ! Se repérer dans toutes les options de préparation des échantillons à séquencer, options qui sont fonctions de l’origine et de l’application que l’on souhaite mettre en oeuvre par l’intermédiaire du séquençage, peut être un réel casse-tête que ce document souhaite simplifier ici.
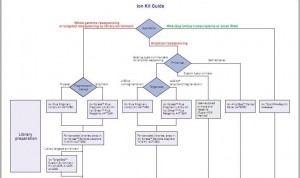 Cet arbre de décision s’étoffera très rapidement : d’autres branches pousseront avec les protocoles « mate pair » & « paired end », les nouvelles indexations… et surtout l’arrivée du kit permettant de séquencer 400 b… Peut être que les branches portant les options 100 b seront, quant à elles, pour l’occasion élaguées.
Cet arbre de décision s’étoffera très rapidement : d’autres branches pousseront avec les protocoles « mate pair » & « paired end », les nouvelles indexations… et surtout l’arrivée du kit permettant de séquencer 400 b… Peut être que les branches portant les options 100 b seront, quant à elles, pour l’occasion élaguées.
Ce document, et beaucoup d’autres, est disponible sur le site de la communauté Ion Torrent.
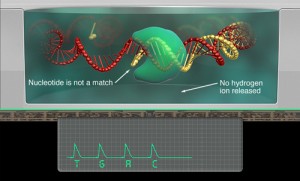
Pour donner suite aux demandes assez nombreuses de détails concernant le principe de fonctionnement du séquençage multiparallélisé PGM (Ion Torrent / Life Technologies).
Sur le lien suivant vous trouverez une vidéo de principe de la technologie Ion Torrent (Life). En supplément, la publication de juillet 2011 de Rothberg et al. dans Nature (pour quelques informations supplémentaires). Il est à noter pour les connaisseurs que Jonathan Rothberg est l’un des fondateurs de la la société 454 Life Sciences (ayant donné naissance au séquenceur haut-débit le plus utilisé dans un passé proche, utilisant le principe du pyroséquençage), technologie vendue à Roche et le créateur de la société Ion Torrent avant que celle-ci ne soit rachetée 350 millions de dollars par Life Technologies.
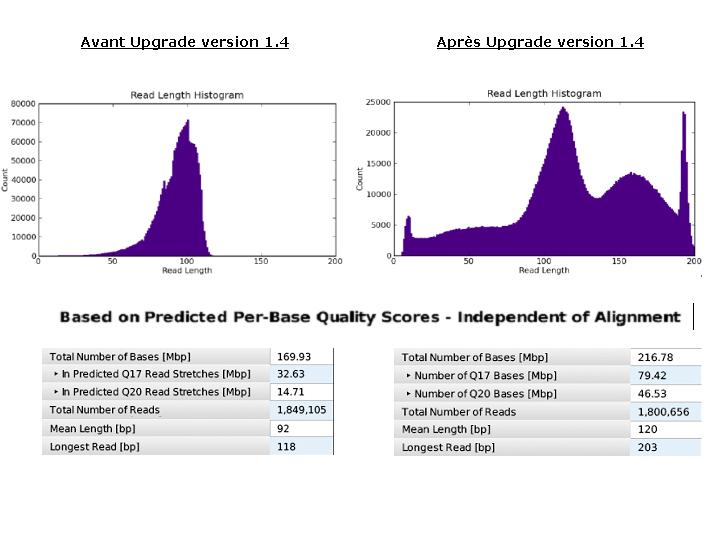
L ’évolution de la technologie Ion torrent permet depuis ces derniers jours, d’accéder au format de puce « 316 » succédant à la « 314 ». Ces puces multiplient par 10 la quantité de données générées à l’issue d’un « run » de séquençage aboutissant ainsi à 100 Mb.
De manière à bénéficier de la capacité totale de ces puces, une mise à jour du serveur, ainsi qu’une configuration du PGM en version 1.4 sont nécessaires et ont été réalisées ce jour. Ces modifications se traduisent par une amélioration de l’algorithme d’analyse (basecalling, alignement, …) optimisant le nombre de reads considérées et traitées. Le développement apporté au niveau des « runs » permet désormais d’intégrer des « reads » de plus grande taille (jusqu’à 200b environ) directement lié à une amélioration de la précision de séquençage.
Techniquement, quelques modifications au niveau du PGM ont conduit au remplacement de certaines tubulures (diamètre supérieur aux précédentes) : Cet épisode fut l’occasion de découvrir les entrailles de la machine…
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages