90 % de notre nourriture repose sur 23 espèces seulement. La diversité de ce que l’on cultive ou élève impacte le contenu de nos assiettes. Inversement, nos pratiques alimentaires et culturales impactent la biodiversité. Du champ à l’assiette, de l’assiette au champ, la biodiversité s’est imposée comme indicateur de la santé de notre terre nourricière, thermomètre d’un système défaillant, se corrigeant ou ignorant parfois son potentiel d’autodestruction. Après la prise de conscience, quelles solutions ?
Source : Biodiversité : prendre soin de son assiette pour prendre soin de la planète
 Si vous vous intéressez au séquençage haut-débit, que vous souhaitez avoir un panorama des diverses technologies à disposition et que vous êtes friands de schémas de principe: la publication de Sara Goodwin, John D. McPherson & W. Richard McCombie. Cet article publié dans la revue Nature de mai 2016 promet de faire un retour en arrière sur 10 ans d’évolution du séquençage haut-débit. Elle parvient à tenir ses promesses et livre effectivement des schémas (avec la charte graphique « Nature ») très bien faits, très pédagogiques ! En outre, le tableau (Table 1 | Summary of NGS platforms) permet à tous les pourvoyeurs de projets nécessitant le recours à du séquençage, d’avoir un pense bête sous la main pour associer la bonne technologie à la question biologique qui leur incombe… Et comme vous êtes pressés, vous pourrez retrouver l’intégralité de cet article en vous promenant et cliquant sur l’image ci-dessous.
Si vous vous intéressez au séquençage haut-débit, que vous souhaitez avoir un panorama des diverses technologies à disposition et que vous êtes friands de schémas de principe: la publication de Sara Goodwin, John D. McPherson & W. Richard McCombie. Cet article publié dans la revue Nature de mai 2016 promet de faire un retour en arrière sur 10 ans d’évolution du séquençage haut-débit. Elle parvient à tenir ses promesses et livre effectivement des schémas (avec la charte graphique « Nature ») très bien faits, très pédagogiques ! En outre, le tableau (Table 1 | Summary of NGS platforms) permet à tous les pourvoyeurs de projets nécessitant le recours à du séquençage, d’avoir un pense bête sous la main pour associer la bonne technologie à la question biologique qui leur incombe… Et comme vous êtes pressés, vous pourrez retrouver l’intégralité de cet article en vous promenant et cliquant sur l’image ci-dessous.
.jpg) Une femme américaine devient « biologiquement » plus jeune après le suivi d’une thérapie génique que sa propre société a développé. Où quand Dorian Gray fait du commerce sans nuance (attention ! jeu de mots) en mode transhumanisme 2.0.
Une femme américaine devient « biologiquement » plus jeune après le suivi d’une thérapie génique que sa propre société a développé. Où quand Dorian Gray fait du commerce sans nuance (attention ! jeu de mots) en mode transhumanisme 2.0.
Elizabeth Parrish, directrice générale de Bioviva USA Inc., est devenue, à grand renfort de communication, le premier être humain « à rajeunir » grâce à la thérapie génique développée par cette même entreprise. Cette dernière, sur la page d’accueil de son site internet, promet de démocratiser, dans un avenir proche, la thérapie génique et cellulaire. Dans un environnement convivial et confortable, les médecins de la société sont là pour corriger, avec précision, votre patrimoine corporel en voie de décrépitude.
L’un des premiers développements de la société, trouve une application dans la lutte contre le vieillissement, ce qui constitue toujours un excellent business plan compte tenu d’une corrélation établie entre âge et solvabilité. Ce philtre de jeunesse cible les télomères dont la taille serait proportionnelle à notre délai de péremption. De par leur structure particulière, les télomères requièrent d’être maintenus par une transcriptase inverse cellulaire spécifique appelée télomérase. En absence ou dans le cas de défaillance de télomérase, les télomères raccourcissent progressivement, jusqu’à atteindre une taille critique qui entraîne un arrêt des divisions cellulaires caractérisant la sénescence réplicative. Ainsi, les télomères forment une structure essentielle dans le contrôle de la viabilité cellulaire. Ceux-ci permettent de maintenir l’équilibre entre le vieillissement cellulaire et le risque de prolifération cellulaire incontrôlée.
Le score des télomères est calculé en fonction de la longueur des télomères des lymphocytes T. Ce résultat est basé sur la moyenne des longueurs des télomères des lymphocytes T par rapport à celle de la population américaine de même classe d’âge. Plus le score est élevé plus les cellules concernées seront considérées comme « jeunes » et par extension, plus le patient sera lui-même considéré comme biologiquement jeune. En septembre 2015, Elizabeth Parrish âgée de 44 ans (un peu jeune notre candidate au rajeunissement) a reçu deux des thérapies géniques expérimentales de sa propre entreprise:
- l’une pour la protéger contre la perte de la masse musculaire liée à l’âge
- l’autre combattant cette diminution quantitative de cellules souches associée au vieillissement et à ses conséquences
Le test évoqué ici a été initialement conçu pour démontrer l’innocuité de la dernière génération des thérapies géniques et cellulaires. Si les premières données s’avèrent exactes et incontestables, il s’avère que ce test constituera une première mondiale: l’allongement des télomères dont le rognage était perçu comme inéluctable, irréversible. Dans le viseur de Bioviva, le vieillissement réversible est donc en ligne de mire ou du moins les gérascophobes que le test aura rassuré voire convaincu.
Auparavant, la thérapie génique a été utilisée pour allonger les télomères des cellules murines cultivées, mais jamais sur un patient humain. En septembre 2015, les scores des télomères des globules blancs d’Elizabeth Parrish ont été collectés, par une clinique spécialisée au niveau des tests de laboratoire (SpectraCell à Houston), immédiatement avant que les traitements ne lui furent administrés. Ces scores ont révélé que les télomères d’Elizabeth Parrish étaient inhabituellement courts pour son âge, la laissant précocement vulnérable aux maladies liées à l’âge. En mars 2016, les mêmes tests ont été effectués par SpectraCell. Ces derniers ont révélé que ses télomères avaient « allongé » d’environ 20 ans, passant de 6,71kb à 7,33kb: les globules blancs d’Elizabeth Parrish sont devenus biologiquement plus jeunes. Ces résultats ont été contrôlés de façon indépendante par les fondations: Bruxelles Heales (Healthy Company Life Extension), et la fondation britannique Biogerontology Research Foundation.

Elizabeth Parrish mais qui aimerait bien le devenir
Elizabeth Parrish argumente: « actuellement, peu de thérapies offrent de réels avantages pour les personnes souffrant de maladies du vieillissement. La modification du mode de vie a un impact que limité pour le traitement de ces maladies. Les progrès de la biotechnologie apparaît être une meilleure solution, [avec ce test] nous avons fait l’histoire! ». Plusieurs signaux alimentent le scepticisme des membres de la communauté scientifique. Ainsi le fait qu’il n’y ait pas aujourd’hui de corrélation établie entre la longueur des télomères et la santé d’une personne. « C’est comme pour les cheveux gris, ce n’est pas parce qu’on se les teint qu’on vivra plus longtemps » commente Dana Glei, chercheuse à l’Université de Georgetown. Bioviva continuera de contrôler le sang de Parrish pendant les années à venir. Il reste à évaluer si le succès observé sur des leucocytes peut être étendu à d’autres tissus ou organes, est ce que ce teindre les cheveux ou rallonger ses télomères nous permet de perdre (ou gagner, cela dépend du point de vue) quelques années. Toutes ces interrogations pourraient avoir leurs réponses dans les cellules d’Elizabeth Parrish, le «patient zéro» de la thérapie génique réparatrice. Depuis ses premières injections de thérapie géniques, Bioviva a suscité un intérêt mondial, le scepticisme de communauté scientifique, l’engouement des investisseurs et à fourni un cas d’école pour tout bon bioéthicien.
Malgré, le tirage d’oreilles de la FDA (la Food and Drug Administration), les consommateurs de génomique récréative ne se sont pas réellement détournés de 23andMe, société dans laquelle Google a injecté 4,0 millions d’USD. La société de Anne Wojcicki l’ex-épouse du cofondateur de Google, Sergey Brin, n’a pas vu une perte significative de clientèle et, au contraire, a continué de recruter de nouveaux clients… pour franchir le cap du 1.000.000 durant l’année 2016.

L’implication de Google dans la société s’essoufflant (un divorce prononcé entre les fondateurs respectifs des deux sociétés, en 2015), 23andMe a cherché à transformer sa base de données de profils génomiques en profits financiers directs. Ainsi, plus d’une dizaine de sociétés ont négocié un accès à ces informations (Genentech, Pfizer, etc.). Ces informations semblent aujourd’hui constituer le vrai « business model » de 23andMe.
Si cette monétisation était complètement prévisible, il faut avouer que l’impact ou plutôt le non impact de la prise de position de la FDA atténue grandement ce que l’on avait pu annoncer au sein de l’article : 23andMe au point mort.

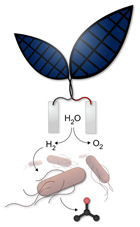
Les cellules photovoltaïques ont un potentiel considérable pour satisfaire les besoins futurs en énergie renouvelable, cependant des méthodes efficaces et évolutives de stockage de l’électricité intermittente qu’elles produisent, sont aujourd’hui attendues pour la mise en œuvre, à grande échelle, de l’énergie solaire. Un stockage de cette énergie solaire pourrait passer par la case carburant. Le travail, présenté dans PNAS de février 2015 (Efficient solar-to-fuels production from a hybrid microbial–water-splitting catalyst system), rapporte le développement d’un système bioélectrochimique évolutif, intégré dans lequel la bactérie Ralstonia eutropha est utilisée pour convertir efficacement le CO2, avec l’hydrogène et l’oxygène produits à partir de dissociation de l’eau, en biomasse et alcools (cf. schéma ci-contre). Les systèmes photosynthétiques artificiels peuvent stocker l’énergie solaire et réduire chimiquement le CO2. Le système de fractionnement-biosynthétique hybride est basé sur un système de catalyseur inorganique, relativement abondant sur Terre (nécessairement sinon écologiquement ce ne serait pas terrible, avouons le…), biocompatible pour séparer l’eau en hydrogène et oxygène à des tensions basses. Lorsqu’elle est cultivée en contact avec ces catalyseurs, Ralstonia eutropha consomme le H2 produit pour synthétiser de la biomasse et des carburants voire d’autres produits chimiques, à partir de faible concentration de CO2 – sous entendu à des concentrations voisines de celles présentes dans l’air. Ce système évolutif a une efficacité énergétique de réduction de CO2 d’ ~ 50% lors de la production de la biomasse bactérienne et d’alcools. Ce dispositif hybride couplé à des systèmes photovoltaïques existants donnerait une efficacité énergétique de réduction des émissions de CO2 d’environ 10%, supérieure à celle des systèmes photosynthétiques naturels ! Nous en conviendrons la bactérie utilisée ici est génétiquement modifiée afin d’orienter son métabolisme vers une voie anabolique d’intérêt.

Ces travaux ouvrent la voie vers la « photosynthèse de synthèse ». Dans cette configuration intégrée, les rendements du solaire à la biomasse vont jusqu’à 3,2% du maximum thermodynamique pour dépasser celle de la plupart des plantes terrestres. En outre, l’ingénierie de R. eutropha a permis la production d’isopropanol jusqu’à 216 mg/L, le plus haut rendement jamais rapporté (> 300 %). Ce travail démontre que les catalyseurs d’origine biotique et abiotique peuvent être interfacés, intégrés… pour permettre à partir de l’énergie solaire, du CO2 et de quelques bactéries de développer des systèmes efficaces stockant une énergie intermittente sous forme de molécules organiques (le biomimétisme est la vraie tendance du moment).
Dans cette première version (celle du PNAS) l’électrode utilisée en nickel-molybdène-zinc s’est avérée toxique pour les bactéries qui voyaient leur ADN attaqué… dans cette version améliorée publiée dans Science (3 juin 2016) , l’électrode toxique a été changée par une autre composée de cobalt-phosphore… Selon Daniel Nocera, le promoteur de l’étude : « Cela nous a permis d’abaisser la tension conduisant à une augmentation spectaculaire de l’efficacité. »
, l’électrode toxique a été changée par une autre composée de cobalt-phosphore… Selon Daniel Nocera, le promoteur de l’étude : « Cela nous a permis d’abaisser la tension conduisant à une augmentation spectaculaire de l’efficacité. »
Nocera et ses collègues ont également été en mesure d’élargir la gamme de produits q’un tel système est capable de synthétiser pour y inclure l’isobutanol (un solvant) et l’isopentane (utilisé dans des boucle fermée pour actionner des turbines), ainsi que le PHB (un précurseur de bioplastiques). La conception chimique du nouveau catalyseur permet également une certaine « auto-régénération, » ce qui signifie que l’électrode ne sera pas lessivée au fur et à mesure de son activité.
 A moitié scientifique et à moitié homme d’affaire, Craig Venter qui n’a pas très bon goût concernant les couvertures de ses livres essaie de mettre la main sur les données de plusieurs centaines de milliers à plusieurs millions de génomes (séquences totales ou profils génétiques). Mais que l’on se rassure c’est pour le bien de l’humanité ou au moins de la transhumanité !
A moitié scientifique et à moitié homme d’affaire, Craig Venter qui n’a pas très bon goût concernant les couvertures de ses livres essaie de mettre la main sur les données de plusieurs centaines de milliers à plusieurs millions de génomes (séquences totales ou profils génétiques). Mais que l’on se rassure c’est pour le bien de l’humanité ou au moins de la transhumanité !
Depuis 2005, les technologies de séquençage n’ont cessé d’être plus rapides et moins chères. En 2014, plus de 225.000 génomes humains étaient déjà séquencés grâce à plusieurs initiatives dont le fameux « 100 000 Genomes Project » britannique lancé en 2013. Début 2014 Illumina lançait une campagne de publicité mettant en scène le HiSeqX Ten, le premier séquenceur permettant d’atteindre la promesse d’un coût de séquençage humain à 1000 $. Cette année AstraZeneca annonçait sa collaboration avec le Human Longevity Institute de Craig Venter permettant à ce dernier un accès aux génomes ou profils génomiques de 2 000 000 de personnes d’ici 2020. En utilisant la seule séquence d’ADN, Venter dit que son entreprise peut maintenant prédire la taille, le poids, la couleur des yeux et la couleur des cheveux d’une personne, et produire une image approximative de son visage. Une grande partie de ces « détails » est dissimulé dans les variations rares, dit Venter, dont le propre génome a été mis à disposition dans les bases de données publiques depuis plus d’une décennie. Soit dit en passant, même ce promoteur d’un certain transhumanisme regrette son geste : « Si je devais conseiller un jeune Craig Venter », je dirais, réfléchissez bien avant que vous veniez déverser votre génome sur Internet« …
Quelques questions centrales demeurent et l’une d’elle consiste à envisager que le génome d’une personne n’est pas du ressort de sa seule propriété… en effet, rendre disponible son génome revient à rendre disponible une partie des informations de ces enfants et des enfants de ceux-ci etc. Effectivement, les promoteurs de la génomique à large échelle envisagent de dépasser les problématique de l’héritabilité cachée (à ce sujet, lire l’excellent article de Bertrand Jordan dans M/S : Le déclin de l’empire des GWAS). Voici un extrait très pertinent qui explicite ce problème : « Les identifications réalisées dans le cadre des études GWAS sont certes scientifiquement valables et utiles pour la compréhension du mécanisme pathogène (donc porteuses d’espoirs thérapeutiques), mais, rendant compte de moins d’un dixième des héritabilités constatées, elles passent visiblement à côté d’un phénomène important… Comment résoudre ce paradoxe ? Il faut pour cela revenir sur ce qu’examinent réellement les GWAS. Elles se limitent aux Snip, faisant (pour le moment du moins) l’impasse sur les copy number variations (CNV), ces délétions, duplications ou inversions dont on a découvert récemment plusieurs centaines de milliers dans notre génome. Et même pour les Snip, elles ne donnent pas une image complète des variations génétiques entre individus. Par la force des choses, les 500 000 Snip représentés sur les puces d’Affymetrix ou d’Illumina (et qui ont préalablement été étudiés par le consortium HapMap) correspondent à des poymorphismes assez facilement repérables dans un échantillon de population : la règle adoptée a été de ne retenir que les Snip pour lesquels la fréquence de l’allèle mineur est au moins égale à 5 %. Cet usage était nécessaire pour limiter les difficultés dans le positionnement des Snip lors de l’établissement des cartes d’haplotypes ; mais il a pour conséquences que les GWAS n’examinent que les variants fréquents… Selon une hypothèse largement répandue, les maladies multigéniques fréquentes (diabète, hypertension, schizophrénie…) seraient dues à la conjonction de plusieurs allèles eux aussi fréquents : c’est la règle « common disease, common variant » souvent évoquée depuis une dizaine d’années. Les résultats de la centaine d’études d’association pangénomiques pratiquées à ce jour indiquent que cette hypothèse est très probablement fausse : les variants communs ne rendant compte que d’une faible partie de l’héritabilité, le reste est vraisemblablement dû à des variants rares (ponctuels ou non) dont ces études ne tiennent pas compte puisque les puces utilisées ne les voient pas. »
Ainsi, pour franchir ce cap, une solution simple est envisagée : le changement de résolution avec pour credo le passage de profils génomiques (quelques millions de SNPs) à l’intégralité du génome… et après l’épigénome et en même temps le métagénome. Si ces sciences bâties sur une technologie en pleine révolution permettent l’accès à un patrimoine humain universel (l’information génomique quasi exhaustive), si ces sciences renouvellent sans cesse leurs promesses -il faut des fonds et donc convaincre les pouvoirs publics pour acheter la technologie américaine qui permet d’accomplir ces sciences- hypothéquer le patrimoine humain ou pire le privatiser pourrait être une erreur dramatique dont on a du mal à mesurer l’étendue des conséquences.
 Une jeune start-up est née en mai 2012 : cette société issue du CNRS est porteuse d’une innovation dans le secteur du séquençage haut-débit. Enfin, une alternative française aux anglosaxons qui sont présents sur le marché depuis une dizaine d’année ! Maintenant… espérons que ce nouveau né n’arrive pas trop tard sur un marché animé par des fournisseurs de séquenceurs de 2ème génération (un marché mature) et d’autres fournissant des solutions de 3ème génération, riches de promesses.
Une jeune start-up est née en mai 2012 : cette société issue du CNRS est porteuse d’une innovation dans le secteur du séquençage haut-débit. Enfin, une alternative française aux anglosaxons qui sont présents sur le marché depuis une dizaine d’année ! Maintenant… espérons que ce nouveau né n’arrive pas trop tard sur un marché animé par des fournisseurs de séquenceurs de 2ème génération (un marché mature) et d’autres fournissant des solutions de 3ème génération, riches de promesses.
PicoSeq derrière ce nom emprunt d’humilité se cache une technologie de séquençage des plus ingénieuses : en effet, SIMDEQ™ (SIngle-molecule Magnetic DEtection and Quantification) la technologie de PicoSeq utilise une approche biophysique pour extraire des informations à partir de la séquence d’ADN ou d’ARN.
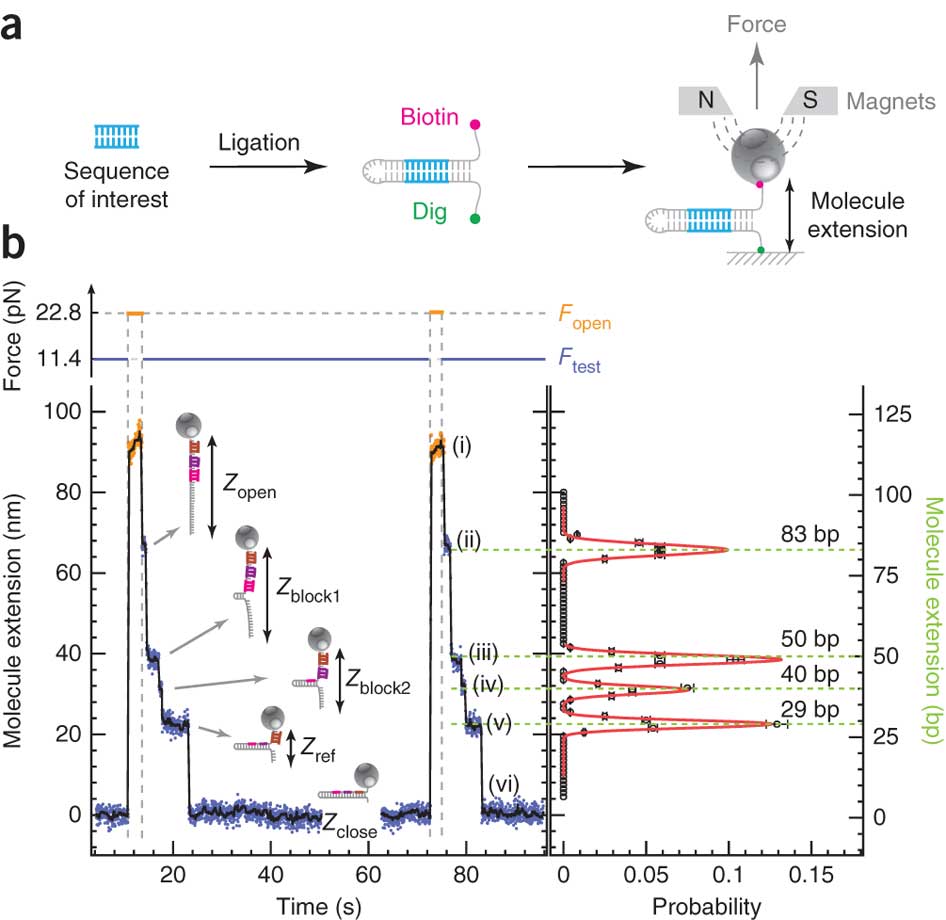
En s’appuyant sur cette représentation schématique tirée de Ding et al. (Nature Methods, 2012), on y voit un peu plus clair. Des fragments d’ADN ou d’ARN que l’on souhaite analyser servent de matrice pour la réalisation d’une librairie en «épingle à cheveux». Pour chaque épingle à cheveux, un côté d’un brin d’acide nucléique est attaché sur une surface solide plane et l’autre à une bille magnétique. En plaçant les billes dans un champ magnétique, modulant celui-ci de manière cyclique, les épingles à cheveux peuvent être auto-hybridées ou non (zip ou unzip). Ce processus peut être effectué des milliers de fois sans endommager les molécules constitutives de la librairie. La position de chaque bille est suivie à très haute précision permettant de voir ce processus d’ouverture et de fermeture en temps réel: nous avons donc là un signal brut permettant, en fonction de la force appliquée pour ouvrir totalement l’épingle à cheveu et des séquences d’oligonucléotides séquentiellement introduites dans le système de modifier la distance bille-support et de jouer sur le temps nécessaire où la force s’applique pour ouvrir l’épingle à cheveu…
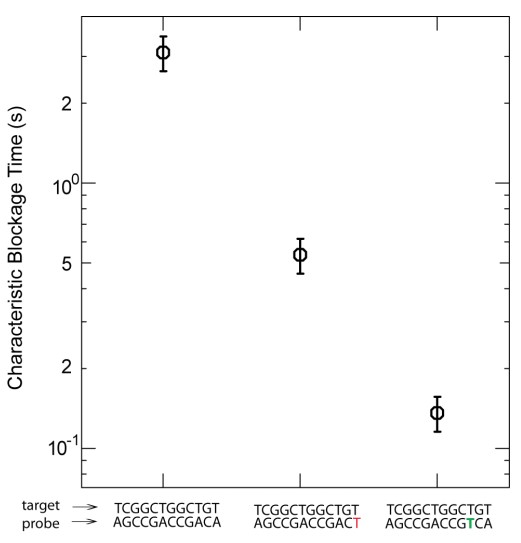
La figure ci-dessus (présente dans les données supplémentaires de l’article sus-cité) permet d’appréhender le potentiel de discrimination de la méthode… où le temps de blocage est fonction du nombre de mésappariements et de la position de ces mésappariements…
Finalement si l’aspect technique est intéressant puisqu’en rupture avec les méthodes proposées par PacBio certainement un peu moins avec le système proposé par Oxford Nanopore Technologies, si la perspective annoncée par PicoSeq est réellement séduisante: l’accès modifications épigénétiques de l’ADN, la question centrale est de savoir si le pas de la commercialisation (dans des conditions propices au succès) d’un tel outil, sera franchi.
Un article d’ Atlantico de septembre 2015, titré : les trois raisons pour lesquelles la France est incapable de rivaliser avec les géants américains de l’analyse ADN, est assez éclairant pour imaginer comment la concrétisation d’une preuve de concept peut être un chemin ubuescokafkaïen. Pour illustrer cela les propos de Gordon Hamilton, le directeur de la startup PicoSeq qui s’inquiète sur les entraves « typically french » peuvent faire office de témoignage. Ce dernier s’inquiète : « La qualité de la recherche scientifique (en France) est incroyable, l’une des meilleures » « le seul souci, c’est que l’on a beaucoup de difficultés ici à transformer ces recherches en vrai business » pour finir par citer en exemple les lenteurs administratives spécificités latines : « nous avons mis presque deux ans pour négocier les licences nécessaires aux brevets de Picoseq. Le même processus en Californie prend entre deux semaines et deux mois. Sur un marché aussi rapide que celui-ci, deux ans c’est très long. Tout change vite, c’est donc impossible pour nous d’être de sérieux concurrents de ces sociétés américaines qui ont toujours un temps d’avance ».
 Voici le fruit d’un bien joli projet : aborder, à portée d’enfants (à partir de 8 ans), l’essentiel de la microbiologie sous forme de propos scientifiques recueillis et « montés » par Florence Pinaud accompagné des illustrations de Stéphane Kiehl. En 88 pages -la moitié des pages étant dévolue aux illustrations-, ce livre tout carré, la guerre secrète des microbes, permet d’initier nos chères têtes plus ou moins blondes (parfois leurs chers parents), aux secrets qui nous habitent tous: de quoi les microbes sont ils le nom ? Les curieux trouveront des éléments relatifs à la vaccination, aux stratégies de défense mises en place par notre système immunitaire ou encore la raison pour laquelle une fourmi folle est irrépressiblement attirée par le fait d’aller grimper en haut d’un brin d’herbe afin qu’elle puisse plus aisément être ingérée par une vache saine d’esprit…
Voici le fruit d’un bien joli projet : aborder, à portée d’enfants (à partir de 8 ans), l’essentiel de la microbiologie sous forme de propos scientifiques recueillis et « montés » par Florence Pinaud accompagné des illustrations de Stéphane Kiehl. En 88 pages -la moitié des pages étant dévolue aux illustrations-, ce livre tout carré, la guerre secrète des microbes, permet d’initier nos chères têtes plus ou moins blondes (parfois leurs chers parents), aux secrets qui nous habitent tous: de quoi les microbes sont ils le nom ? Les curieux trouveront des éléments relatifs à la vaccination, aux stratégies de défense mises en place par notre système immunitaire ou encore la raison pour laquelle une fourmi folle est irrépressiblement attirée par le fait d’aller grimper en haut d’un brin d’herbe afin qu’elle puisse plus aisément être ingérée par une vache saine d’esprit…
Assez brièvement, à l’aide d’un champ lexical martial, les sujets tiennent en haleine les chérubins avides de savoir. Les illustrations, quant à elles, ne sont pas à proprement parler pédagogiques mais artistiques frôlant parfois l’abstraction. Elles ne font pas simplement écho à ce qui est abordé textuellement mais elles livrent une belle poésie picturale et interprètent à elles seules le propos scientifique -le transcendent, oserait on dire ! Facultatives donc essentielles ! Très franchement, il est rare de trouver un livre aussi bien produit conciliant un propos adapté (certains appellent cela de la vulgarisation…) tout en étant exigeant, et cerise sur le gâteau, accompagné d’illustrations n’ayant pas pour objectif d’aider à la compréhension mais plutôt à nourrir un imaginaire motivant pour la compréhension de la phénoménologie d’un monde invisible peuplé de microbes, virus et autres parasites.

 Journaliste dans les secteurs économiques et sociaux, Florence Pinaud a concilié les sujets très sérieux et quelques joyeuses incursions dans la presse jeunesse. Journaliste à La Tribune, elle travaille également pour Espace social européen et anime un blog sur les animaux. Auteur d’ouvrages documentaires et pratiques (Manager la génération Y, 2011, éditions Dunod), elle a publié en 2012 chez Actes Sud Junior Respecter les animaux à petits pas, La mode sous toutes les coutures, C’est mathématique !, A l’école des espions et La guerre secrète des microbes.
Journaliste dans les secteurs économiques et sociaux, Florence Pinaud a concilié les sujets très sérieux et quelques joyeuses incursions dans la presse jeunesse. Journaliste à La Tribune, elle travaille également pour Espace social européen et anime un blog sur les animaux. Auteur d’ouvrages documentaires et pratiques (Manager la génération Y, 2011, éditions Dunod), elle a publié en 2012 chez Actes Sud Junior Respecter les animaux à petits pas, La mode sous toutes les coutures, C’est mathématique !, A l’école des espions et La guerre secrète des microbes.
 Oxford Nanopore Technologies (ONT) crée un nouveau marché pour le séquençage haut-débit: le séquençage (haut-débit ?) à la portée de tous et en mode tout-terrain… mais pourquoi faire ? Le principe de cette technologie a été abordé dans plusieurs de nos articles : les données produites sont constituées de longs reads (quelques milliers de bases frôlant les 10 kB de moyenne), des reads assez bruités au-delà de 10 % d’erreurs; suffisamment longs pour permettre une identification quasi-certaine mais encore trop bruité (et trop peu profond dans le format portable MinION et maintenant SmidgION) pour pratiquer un bel assemblage de novo.
Oxford Nanopore Technologies (ONT) crée un nouveau marché pour le séquençage haut-débit: le séquençage (haut-débit ?) à la portée de tous et en mode tout-terrain… mais pourquoi faire ? Le principe de cette technologie a été abordé dans plusieurs de nos articles : les données produites sont constituées de longs reads (quelques milliers de bases frôlant les 10 kB de moyenne), des reads assez bruités au-delà de 10 % d’erreurs; suffisamment longs pour permettre une identification quasi-certaine mais encore trop bruité (et trop peu profond dans le format portable MinION et maintenant SmidgION) pour pratiquer un bel assemblage de novo.
Donc imaginez vous, perdu au fin fond de l’Amazonie à la recherche de cette plante évoquée par le « sorcier » de la tribu Mashco-Piro que vous venez de quitter, plante potentiellement inconnue de notre médecine occidentale… Qu’à cela ne tienne! vous marchez en quête de la dite plante, vous, votre panneau solaire, votre smartphone, vos appareils ONT (Voltrax + SmidgION). Quasi certain de vérifier in situ votre trouvaille à l’aide d’un séquençage de 3ème génération. Enfin, ceci serait parfait si l’on oublie que Metrichor (le BaseCaller d’ONT) fonctionne en ligne… (cf schéma ci-dessous)… malgré les rêves les plus fous de Google et autres Facebook le fin fond de l’Amazonie n’est pas couvert par la 4G ! On peut imaginer qu’une application pour nos téléphones intelligents devra accompagner la mise sur le marché de cette suite d’appareils pour permettre une analyse en mode stand alone.
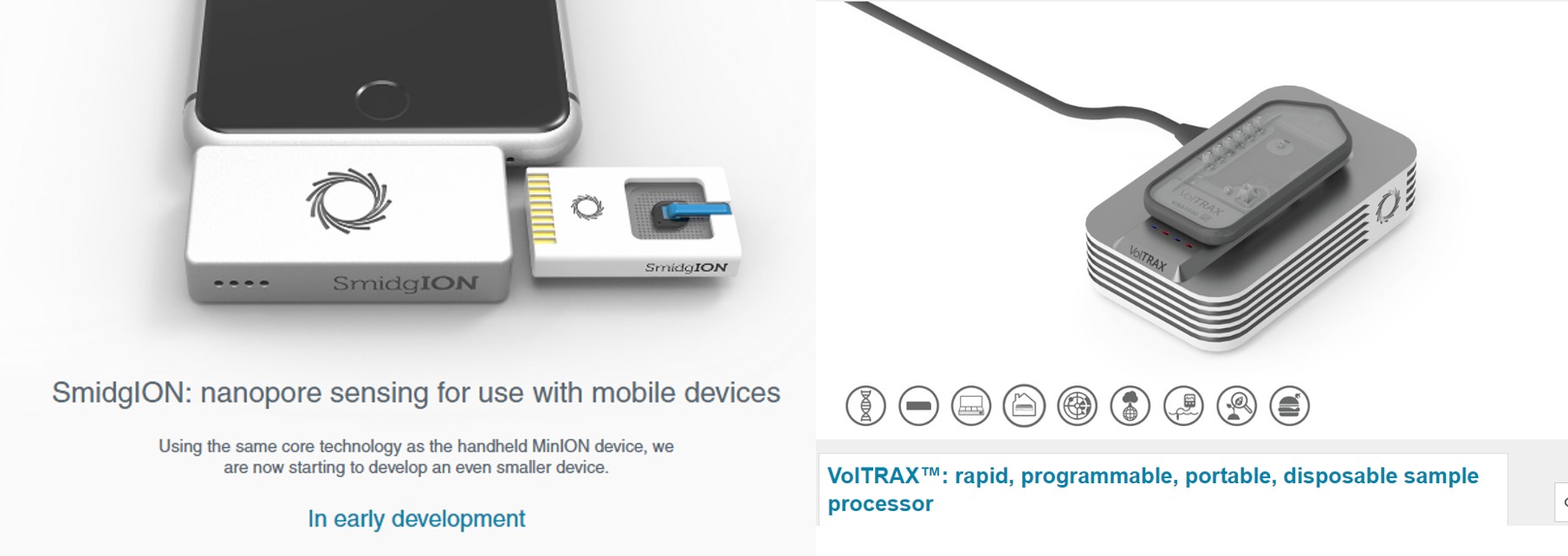
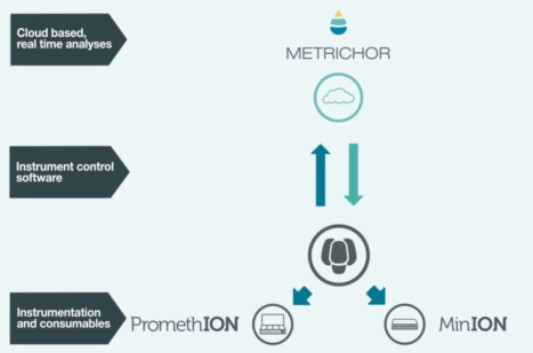
VolTRAX, l’une des promesses d’ONT, permet d’envisager la préparation de librairies à séquencer, ceci même perdu en pleine brousse. Par exemple, les nouveaux kits développés permettent une préparation d’une librairie en une dizaine de minutes. Que vous deviez séquencer un isolat du virus Zika ou Ebola sur le terrain (la logistique et le temps sont comptés) ou que vous deviez séquencer dans votre laboratoire favori, ce type d’automates permettant de simplifier les opérations relatives à l’élaboration de librairies de séquençage est souvent bien accueillis par les techniciens qui pourront s’adonner à des activités plus pertinentes.
 La plate-forme Bilille organise le 23 juin 2016 une journée scientifique pluridisciplinaire sur le thème de l’analyse bioinformatique des données de métagénomique.
La plate-forme Bilille organise le 23 juin 2016 une journée scientifique pluridisciplinaire sur le thème de l’analyse bioinformatique des données de métagénomique.
Cette journée aura lieu à l’amphi Buttiaux de l’Institut Pasteur de Lille.
Le programme préliminaire et le formulaire d’inscription sont disponibles sur le site de la plateforme:
https://wikis.univ-lille1.fr/bilille/metagenomique2016
Date limite d’inscription: 17 juin (l’inscription à cette journée est gratuite, mais obligatoire).
Ces journées s’adressent aux chercheurs, ingénieurs et doctorants en biologie, bioinformatique et biostatistiques. Les exposés sont conçus pour être accessibles, ouverts et favoriser la discussion.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages


