Le système alimentaire est confronté à la défiance des consommateurs tiraillés entre hantise et factualité. Les crises alimentaires, appréciées des médias, modifient notre perception d’un risque associé au besoin vital de se nourrir. À quels dangers réels le consommateur est-il confronté ? À quels défis notre système alimentaire devra-t-il faire face pour garantir notre sécurité alimentaire ?
Source : Crises alimentaires : entre risques authentiques et peurs médiagéniques
Voici OMICtools, une initiative française visant à aider les chercheurs dans la recherche d’outils appropriés pour leurs besoins en analyses de données ‘omiques’
https://omictools.com/
Le constat est simple : les avancées dans les domaines du séquençage, des puces à ADN, de la spectrométrie de masses ont révolutionné la recherche biologique et biomédicale. Cette explosion de données générées engendre de nouvelles problématiques et entraîne une demande toujours plus forte en analyse. Par voie de conséquence, la communauté bioinformatique/biostatistique est extrêmement dynamique en ce qui concerne la création de nouveaux logiciels/méthodes pour l’analyse de données « omiques ». L’émergence de standard dans ce contexte est plutôt complexe au vue du nombre de solutions existantes pour répondre à des problématiques précises.
OMICtools propose une base de données « curée » en accès libre de plus de 4000 solutions d’analyses. Dès le départ, depuis notre problématique d’analyse et grâce à une arborescence à trois niveaux, nous sommes guidés pour arriver à une liste robuste d’outils répondant aux besoins.
OMICtools ne se contente pas de décrire précisément le rôle de chaque solution analytique, il liste également les bases de données associés ainsi que des liens très intéressants vers la littérature tel que des protocoles analytiques ou des comparaisons d’outils. Le fait de lier outils, bases de données et littérature est une des forces de l’outil.
Pour illustrer la navigation, si nous nous plaçons dans le cadre d’une analyse RNA-seq si nous nous posons la question simpliste (mais néanmoins essentielle) : comment dois-je analyser mes données? De (très) nombreuses questions sous-jacentes surgissent en même temps que des solutions : quel outil pour aligner ou assembler? deNovo ou sur référence? S’intéresse t-on à l’épissage alternatif? à la détection de variants? à la quantification des mRNA? quelle méthode de quantification? quelle méthode de normalisation? quelle mesure d’expression différentielle? quelles analyses fonctionnelles en aval?
Pour chaque question, il existe des solutions logicielles, plus ou moins efficaces, plus ou moins fonctionnelles, mais également des comparatifs sous forme de publications qui peuvent aider aux choix. OMICtools propose donc de référencer ces solutions par problématiques en essayant de séparer le bon grain de l’ivraie (OMICtools est vérifié et mis à jour par ces auteurs).
OMICtools permet à un novice dans un domaine de rapidement visualiser les workflows d’analyse et les enjeux analytiques grâce à des schéma associés en illustration de chaque problématique (ex : schéma d’analyse metagénomiques…).
Le projet Plume (Promouvoir les Logiciels Utiles Maîtrisés et Économiques dans la communauté de l’Enseignement Supérieur et de la Recherche – www.projet-plume.org) est une initiative assez similaire initiée en 2007 et référençant plus de 1200 solutions logicielles. Le projet Plume est plus généraliste, les fiches sont bien plus détaillées et sont en français (ce qui peut-être un inconvénient pour une portée plus internationale). Le projet Plume fonctionne toutefois en mode dégradé faute de moyen…
La publication associée à OMICtools est disponible ici : http://database.oxfordjournals.org/content/2014/bau069.long
Après quelques mois d’absence suite à un problème de mise à jour, Biorigami est de retour. Nous nous excusons pour cette longue période d’inactivité.
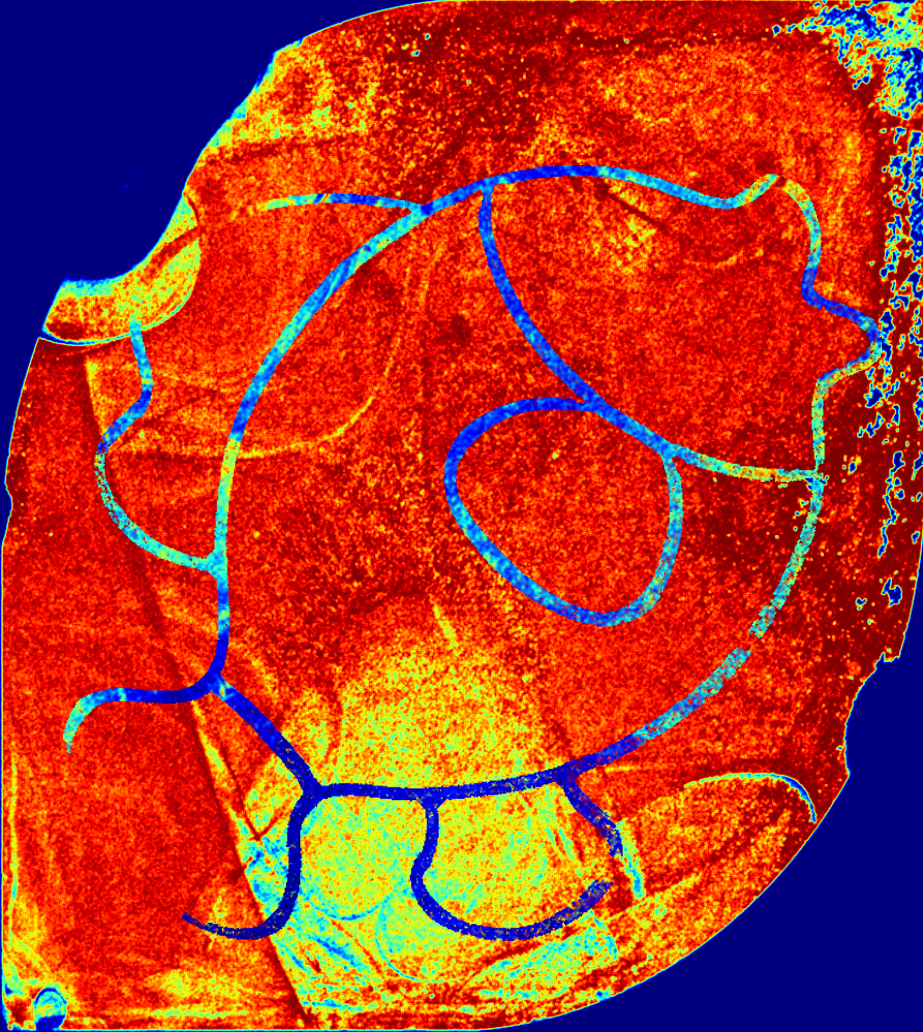
Une heatmap (carte de densités) suite à un séquençage Ion Torrent évalue la densité de billes vivantes (porteuses d’une, ou malheureusement plusieurs, dans le cas de billes polyclonales, séquences matrice de séquençage).
Plus la surface de la puce de séquençage tire vers le rouge sombre plus important devrait être le nombre de reads séquencés. A l’inverse, le bleu sera indicateur d’une zone non pourvue de billes vivantes ou de billes tout court.
Ce premier indicateur de « qualité » de run est très précoce puisqu’il n’est pas nécessaire d’attendre la fin du run pour l’obtenir… ainsi une surface de puce en dégradés de bleus ne vous laissera aucune chance quant à l’exploitation des données (volume trop faible de reads). Il est à noter que la seule lecture de la séquence clé TCAG (portée par la matrice de séquençage) permet à la suite logiciel du PGM Ion Torrent de définir une bille vivante. Ainsi le rouge intégral est loin d’être un indicateur suffisant d’un séquençage satisfaisant.
Aujourd’hui à titre d’exemple nous avons obtenu lors de notre run cette heatmap (photo_Gaël Even) :
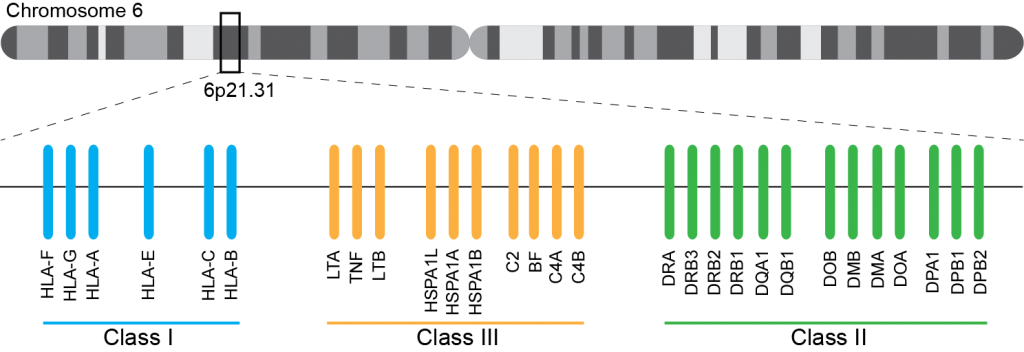
En cancérologie, l’allogreffe de moelle osseuse s’inscrit dans un parcours thérapeutique notamment comme traitement de consolidation après une chimiothérapie. Aussitôt, les notions de rejet ou d’acceptation du greffon apparaissent et il devient indispensable que les systèmes HLA (Human Leucocyt Antigens, découvert en 1950) du donneur et du receveur soient les plus proches possibles.
Ce système immunogène, situé sur le bras court du chromosome 6 chez l’homme, est caractérisé par son polygénisme et son polymorphisme qui sont à l’origine d’une grande variabilité interindividuelle et en fait le déterminant principal du résultat de greffe. L’ensemble des gènes HLA sont subdivisés en trois régions du chromosome 6 qui contiennent chacune de nombreux gènes avec ou sans fonction immunologique. On distingue ainsi la région CMH de classe I, de classe II, et de classe III.
A ce jour, un rendu de typage est ciblé sur une portion génomique restreinte codant pour le HLA. Il s’agit de l’exon 2 et 3 des loci HLA-A, HLA-B et HLA-C (région I), l’exon 2 et 3 des loci HLA-DQ (DQ-A et DQ-B) et l’exon 2 pour HLA-DR (DRA et DRB1), où repose prés de 70% du polymorphisme. La région III ne renfermant pas de gènes intervenant dans la présentation antigénique.
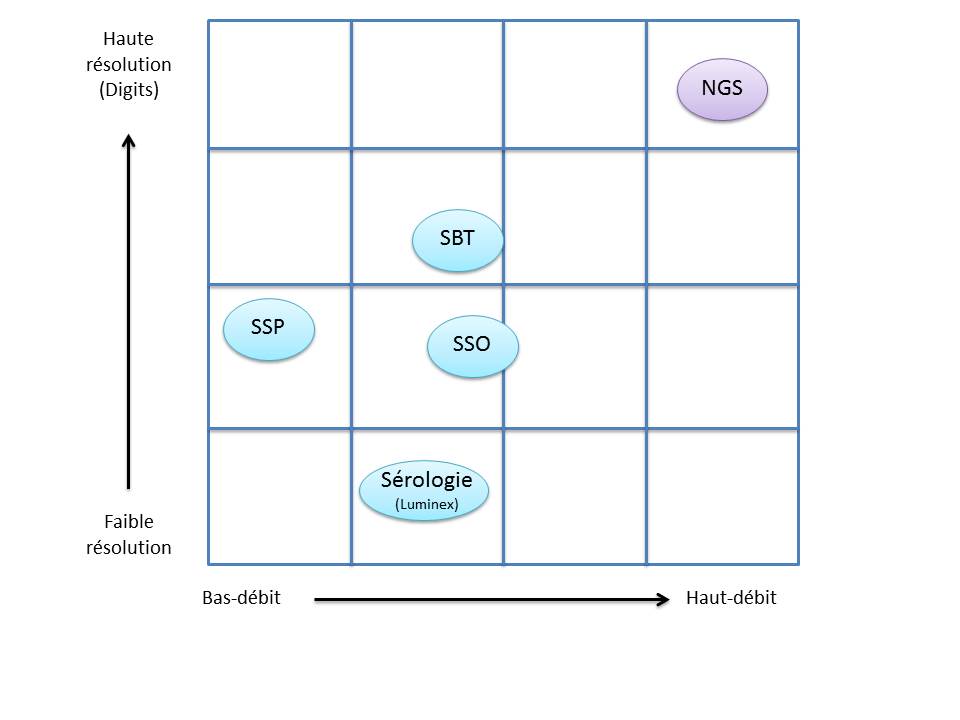
C’est ainsi que différentes approches de typages ont été développées et l’avènement de la technique de PCR au milieu des années 1980 a pallié aux limites de résolution de la sérologie employée jusqu’alors. D’un typage rendu au niveau générique (2 Digits), la « PCR-SSO » (Sequence Specific Oligonucleotide) et la « PCR-SSP » (Sequence Specific Primer) développées dans les années 1990 ont permis d’accéder à un résultat allélique (4 Digits). Cette avancée technologique s’est poursuivie la décennie suivante avec la « PCR-SBT » (Sequence Based Typing) ou séquençage « Sanger » puis plus récemment avec la PCR en temps réel (ex: linkage). Toutes ces techniques de biologie moléculaire ont permis de mettre en avant le polymorphisme et la grande diversité génétique du HLA. Chaque année, de nombreux allèles sont découverts, alimentant continuellement la banque de données de référence IMGT.
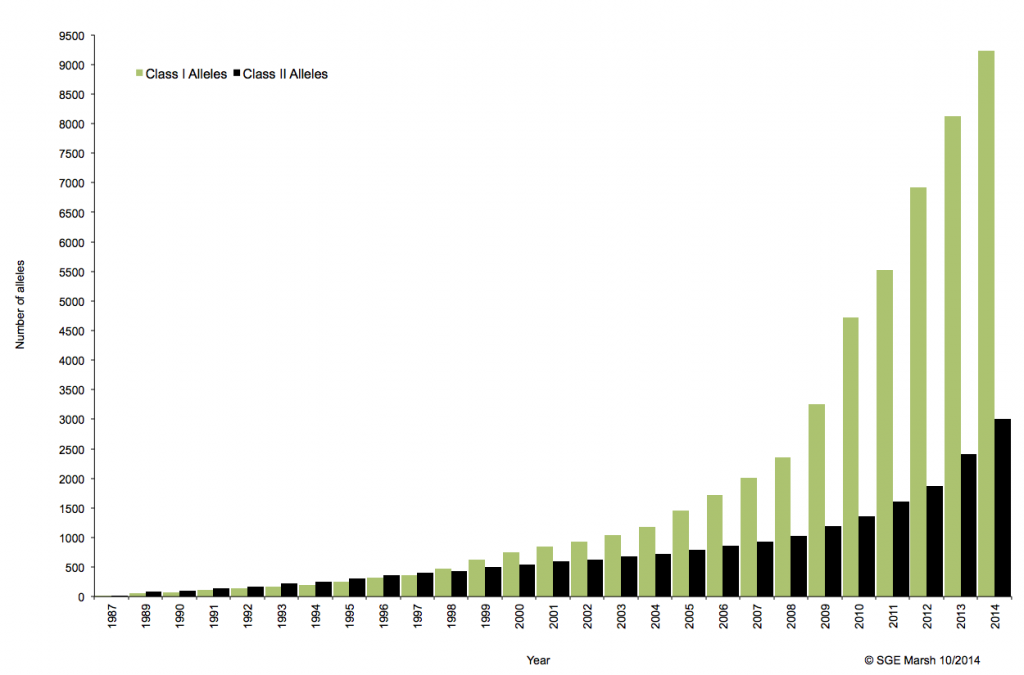
Associé à cette augmentation constante du nombre d’allèles typés, le nombre d’ambigüités croît et met progressivement en difficulté les technologies conventionnelles qui atteignent leurs limites. De plus, le pourcentage de réussite des allogreffes n’atteint environ que 50%. L’exploitation du reste de l’information génomique permettrait potentiellement d’améliorer cette performance par un typage plus affiné.
Le recours au séquençage nouvelle génération apparait donc inévitable. En plus de gérer les ambiguïtés par un séquençage allélique, le NGS permet de traiter simultanément de grandes quantités d’échantillons réduisant ainsi le coût unitaire, d’accéder à un niveau de résolution supérieur (4,6 ou 8 digits) tout en ayant la capacité de cibler des loci entier (Long Range PCR).
Ainsi, plusieurs stratégies existent avec leur solution technique adaptée à la préparation de la matrice d’ADN à séquencer:
– L’amplification ciblée des régions d’intérêt par PCR de fusion. Cette approche permets un gain de temps et une réduction des coûts en s’affranchissant des étapes de fragmentation, ligation, et autres purifications… .Par ailleurs elle n’est pas la mieux adapté dans le cas d’une couverture de séquençage de l’ensemble de la région génomique HLA.
– L’amplification par « Long Range PCR » permet une couverture complète des différents loci étudiés. Les fragments de plusieurs Kb subissent alors une fragmentation, une ligation des adaptateurs et indexation. Cette approche permet d’accéder à davantage d’informations (régions exoniques et introniques).
– La capture de séquences par hybridation. Même si cette solution est bien caractérisée elle n’est pas si efficace en terme de capture avec une disparité selon la taille des fragments.
– Le séquençage de génome entier ou d’exome. Cette approche est la moins biaisée et couvre tous les gènes du système HLA. Paradoxalement, l’analyse nécessite beaucoup trop de ressources pour une utilisation en routine et ne permets pas de traiter autant d’échantillons simultanément.
Plusieurs sociétés commerciales proposent des solutions clé en main depuis la préparation des échantillons (avec l’option « Long Range PCR » qui semble la plus plébiscité) jusqu’à l’analyse de résultats via leur logiciel dédié. Certaines sont en cours de validation de méthode pendant que d’autres tentent d’inonder le marché. Parmi elles, Gendx, Omixon, Illumina, One lambda, Life technologies, Immucor, etc…
L’exploitation des capacités et caractéristiques des solutions de séquençage à haut-débit permettrait d’affiner considérablement le typage HLA. Ainsi le décryptage de l’ensemble des régions codantes et non-codantes du génome d’intérêt représente un enjeu important dans la réussite des greffes. Par ailleurs, cette approche nécessitera une mise à jour considérable des banques de données (IMGT) avec une validation de nombreux nouveaux allèles.
Au cours des dix dernières années, la génomique connait une avancée technologique indéniable au travers des différents procédés de séquençage à haut-débit de deuxième génération. Néanmoins, certaines limites techniques subsistent, notamment par rapport à la quantité d’ADN requit, impliquant donc son extraction à partir de plusieurs millions de cellules. Cette contrainte implique une dilution de l’information pour des cellules aux fonctions biologiques bien souvent hétérogènes jusqu’au sein d’un même tissu.
En attendant la démocratisation du séquençage de troisième génération (Séquençage ADN sans amplification clonale) aux caractéristiques techniques qui permettraient un virage vers la génomique à l’échelle de la cellule unique , des méthodes alternatives appliquées à la seconde génération se développent afin d’accéder à cette hétérogénéité cellulaire. Ces solutions s’accompagnent donc, en amont du séquençage, d’une inévitable amplification de l’ADN de la cellule, préalablement isolée soit microdissection laser, système de microfluidique (ex: C1 Fluidigm), cytométrie en flux, ou encore micropipettage.
Ce poste est donc l’occasion de présenter la méthode d’amplification MALBAC, pour Multiple Annealing and Looping Based Amplification Cycles (Science, Zong et al.).
La méthode MALBAC repose sur l’utilisation de primers spécifiques (séquence de 8 nucléotides variables s’hybridant aléatoirement sur l’échantillon, couplée à une séquence connue de 26 nucléotides) générant des amplicons aux extrémités complémentaires. Cette particularité favorise la formation d’une boucle, évitant ainsi aux brins néo-synthétisés de servir à nouveau de matrice à la PCR et d’engendrer un biais d’ amplification (contrairement à la MDA). Ces étapes d’amplifications quasi-linéaires sont répétées cinq fois, puis les amplicons sont amplifiés par PCR exponentielle classique, en amont du séquençage.

MALBAC favorise une meilleure couverture de séquençage ainsi qu’une amplification plus uniforme (cf représentation ci-dessous) . De par ses performances, elle surclasse les méthodes conventionnelles tel que PEP-PCR, DOP-PCR ou encore MDA, pour Multiple Displacement Amplification, méthode la plus répandue depuis dix ans. Jusqu’à 83% de couverture à 10X de profondeur contre 45% pour la MDA. Parmi les autres atouts, la quantité d’ADN initiale requise n’est que de 0.5pg (contre 1000pg pour la MDA) et la polymérase Bst associée connait un taux d’erreur de 1/10000 bases.
Les performances de cette méthode permettent ainsi de reconsidérer les études génomiques ciblant un matériel biologique rare. Parmi elles, l’analyse de cellules tumorales circulantes, de tissus microdisséqués, de cellules embryonnaires, de micro-organismes, de cellules foetales circulantes, , etc…
Vous qui cherchez une méthode permettant de mettre en évidence des variants rares au sein d’une population hétérogène de produits PCR, l’ice-COLD PCR est peut être faite pour vous.
En effet, identifier des variants rares noyés dans de l’ADN « sauvage » a été techniquement une demande forte de divers champs d’expertises médicales tels que la cancérologie, l’infectiologie et le diagnostic prénatal. Nous avons déjà parlé sur Biorigami, d’une approche assez largement répandue qui consiste à séquencer très profondément (à haut-débit donc) tout ou partie de génome en vue d’identifier ces variants rares potentiellement associés à des phénomènes de résistance à des antibiotiques (à titre d’exemple vous pouvez consulter le travail d’Eurekagenomics présenté sous forme de poster)
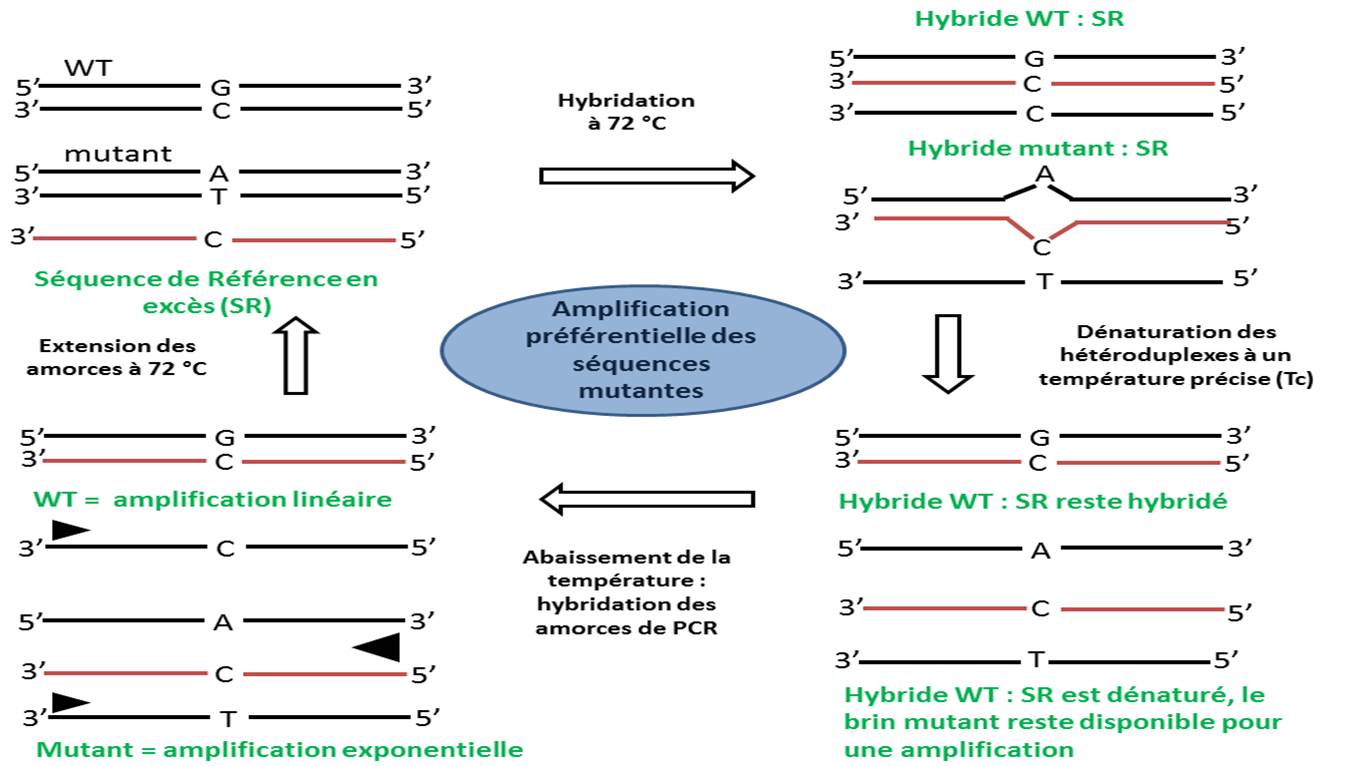
Derrière l’acronyme tautologique, ice COLD, se cache la terminologie : Improved & Complete Enrichment CO-amplification at Lower Denaturation temperature PCR, ce qui pourrait se traduire en français approximatif par : une amélioration de la COLD PCR qui est elle même : une co-amplification à sub-température de dénaturation-PCR. Qu’est ce qui se cache derrière cet énième acronyme ? Pour y voir plus plus clair, voici pour commencer, ce petit schéma:
En observant le schéma ci-dessus, commençons en haut à gauche : nous avons donc des séquences double brin sauvage en large quantité et potentiellement de l’ADN mutant. En premier lieu, il convient de designer des amorces permettant une amplification du locus d’intérêt (pour un amplicon de taille généralement autour de 100 pb). Il vous faudra ensuite une séquence (notée SR sur le schéma) 3′ phosphate pour prévenir des amplifications lors des phases de PCR. Cet oligonucléotide (employé autour d’une concentration de 25 nM finale) viendra s’hybrider parfaitement sur la cible ADN wt et constituera un hétéroduplexe avec toute cible contenant un polymorphisme. A l’aide de la connaissance du Tm (melting temperature ou température de fusion) de l’homoduplexe ADNwt//SR -(note : pour ce faire, il sera nécessaire de designer un autre couple d’amorces afin que la taille du brin SR amplifiée soit égale au brin SR qui lui même est plus court que l’amplicon généré par les premières amorces designées) acquise par la réalisation de courbe de fusion par PCR en temps réel, les hétéroduplexes seront dénaturés en chauffant à une température critique (Tc), généralement de 1 °C inférieure au Tm constaté de l’homoduplexe. Nous sommes, à l’issue de cette étape, en bas à droite de notre schéma, ensuite il restera à réaliser la PCR qui ciblera largement préférentiellement les ADNs disponibles, dénaturés… donc ceux portant des mutations.
Les avantages de cette technique sont multiples :
Cette technique peut être appliquée sur diverses types d’échantillons : ADN fœtal circulant (sérum, plasma), cellules tumorales circulantes, divers fluides corporels, FNA, FFPE, TMA. Elle nécessite de petites quantités de tissu ou d’ADN et peut permettre détecter toutes les mutations présentes au sein du locus amplifié.
En aval, cette méthode accepte une foultitude de techniques de détection qui permettent de réaliser le diagnostic à proprement parlé des mutations :
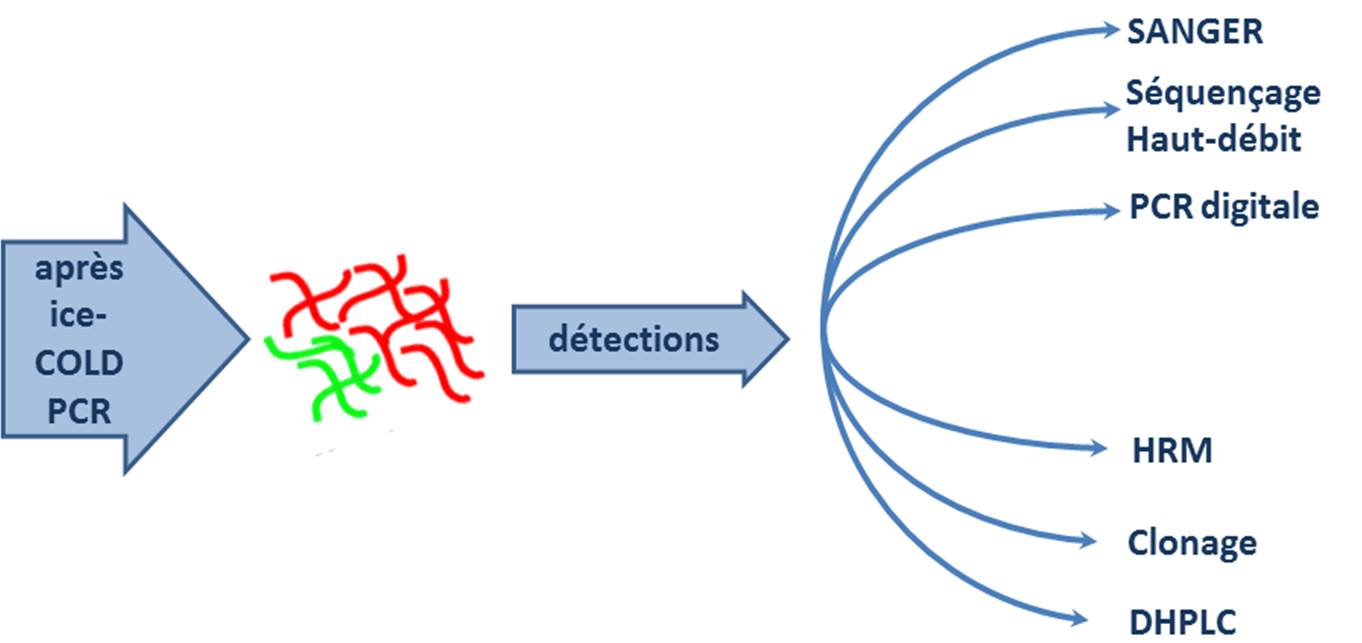
L’ice-COLD PCR est une technique qui est peu exigeante, elle nécessite de disposer d’un thermocycleur assez correct et d’un fournisseur d’oligonucléotides performant (rapide et pas cher) et évidemment de s’y connaitre quelque peu en PCR…
Si, par hasard, vous souhaiteriez plus de précisions, voici une vidéo des plus instructives, l’occasion pour nous de présenter l’excellent site LabTube.com dont une capture d’écran vous est proposée ci-dessus. Ce site internet héberge moult vidéos de conférences, diapositives illustrant une technique (dont l’ice COLD-PCR), bref une source d’informations techniques dont il ne faut pas se priver. Pour preuve au sujet de la recherche de mutations rares voici une présentation de Jorg Tost, directeur du laboratoire épigénétique et environnement (LEE), du Centre national de génotypage, Institut de Génomique/CEA.
Le développement des technologies à haut-débit dédiés aux petits ARNs non codant, récemment identifiés (fin des années 90), voit régulièrement déferler des solutions commerciales et libres pour l’analyse gene ontology.
La puissance de cet outils réside dans:
2) l’incorporation de deux algorithmes pour la caractérisation des fonctions biologiques et pathways sur la base de la prédiction des gènes cibles et faisant appel à cinq bases de données (KEGG, Biocarta, PID, Reactome et Gene Ontology – cf fig. ci-dessous, rectangle orange).
Citation
PLoS One. 2012;7(8):e42390. doi: 10.1371/journal.pone.0042390. Epub 2012 Aug 1.
miRSystem: an integrated system for characterizing enriched functions and pathways of microRNA targets.
Lu TP1, Lee CY, Tsai MH, Chiu YC, Hsiao CK, Lai LC, Chuang EY.
 Vous pouvez tout aussi bien lire cet article en sur le site du Monde : http://www.lemonde.fr/sciences/. Il s’agit du point de vue proposé par Guillaume Miquelard-Garnier, cofondateur du think-tank l’Alambic et maître de conférences au CNAM.
Vous pouvez tout aussi bien lire cet article en sur le site du Monde : http://www.lemonde.fr/sciences/. Il s’agit du point de vue proposé par Guillaume Miquelard-Garnier, cofondateur du think-tank l’Alambic et maître de conférences au CNAM.
Les grandes questions médiatiques du moment concernant l’enseignement supérieur et la recherche, qu’il s’agisse par exemple du campus Paris-Saclay, des partenariats public-privé à l’université, ou des MOOCs [Massive Open Online Courses, cours en ligne ouverts et massifs], laissent souvent de côté une problématique pourtant essentielle : quelle politique de recrutement à court ou moyen terme envisage-t-on pour la recherche académique française, et subséquemment, qui pour faire la recherche en France ?
Historiquement, le système français était plutôt fondé sur des crédits dits récurrents (attribués directement aux laboratoires et répartis ensuite entre chercheurs). Le fonctionnement typique d’un laboratoire était un assemblage de petites équipes « pyramidales » de permanents (un chercheur senior, deux ou trois chercheurs junior) avec un recrutement plutôt jeune, et donc relativement peu d’étudiants ou de chercheurs « précaires » (attaché temporaire d’enseignement et de recherche, post-doctorants…).
Si l’on préfère, le fonctionnement se faisait avec un ratio permanents/non-permanents élevé. Dans ce système, le chercheur junior est celui qui est en charge de faire la recherche au quotidien, d’obtenir les résultats et d’encadrer de près, avant de, plus expérimenté, migrer vers des activités de mentorat scientifique de l’équipe (rôle du chercheur senior).
Ce fonctionnement était à l’opposé de celui, par exemple, des Etats-Unis. La recherche y est financée exclusivement ou très majoritairement sur projet, c’est-à-dire par « appels d’offres » ou « appels à projets ». Les chercheurs, principalement à titre individuel, décrivent leurs idées sous forme de projets à des agences gouvernementales ou des industriels, une sélection étant ensuite effectuée par des panels d’experts et l’argent réparti en fonction de ces choix.
Dans ce système, il y a peu de permanents, beaucoup de non-permanents recrutés sur les budgets issus des appels à projets pour la durée de ceux-ci, et un fonctionnement « individuel » (chaque chercheur, junior ou senior, gère son propre groupe de doctorants et post-doctorants, en fonction de son budget). Le chercheur est, dès son recrutement (aux USA, la tenure track), un chef de groupe-chef de projets, dont le rôle est de définir les grandes orientations intellectuelles, de trouverles financements, de les répartir et de recruter. La recherche proprement dite est alors très majoritairement effectuée par les non-permanents.
SITUATION ALARMANTE
Il n’est pas, ici, question de débattre des avantages et inconvénients des deux systèmes. Le premier favorise le mandarinat et un système de « rente scientifique » quand le second amène une tendance à l’effet Matthieu (6 % des chercheurs américains monopolisent 28 % des financements) et mise beaucoup pour le recrutement des précaires sur un fort attrait des pays asiatiques qui n’est peut-être pas éternel.
Toutefois, il faut constater que, depuis une quinzaine d’années au moins, et encore plus depuis la mise en place de l’Agence nationale de la recherche (ANR) en 2005 et les réformes engagées par Valérie Pécresse et poursuivies jusqu’à aujourd’hui, le système français est en mutation.
Les recrutements sont devenus plus tardifs, autour de 33 ans en moyenne pour un maître de conférences ou un chargé de recherches, soit typiquement cinq ou six années après l’obtention de la thèse (pour les sciences dures). Les financements récurrents ont été largement diminués au profit des financements par projet. Les appels à projets, principalement par le biais de l’ANR ou européens, ont développé les recrutements de post-doctorants. Les initiatives pourpromouvoir l’excellence individuelle des chercheurs se sont multipliées (la prime d’excellence scientifique pérennisée même si rebaptisée, les bourses jeunes chercheurs nationales ou européennes sur un modèle de tenure track ou servant à financer un groupe de recherche indépendant).
Or, plus récemment, la révision générale des politiques publiques (RGPP) conduit, malgré l’autonomie des universités, à un effondrement alarmant des recrutements de chercheurs et enseignants-chercheurs permanents à l’université et dans les établissements publics à caractère scientifique et technologique (EPST). En trois ans, le CNRS est passé de 400 chercheurs recrutés par an à 300 (soit d’environ 350 à 280 jeunes chercheurs). Cette année, on compte au total 1 430 postes de maîtres de conférences ouverts au concours alors qu’il y en avait encore 1 700 il y a deux ans et 2 000 il y a cinq ans. Cette situation a conduit le conseil scientifique du CNRS à s’alarmer récemment, et ne devrait pas s’améliorer à la suite des énormes problèmes financiers de bon nombre d’universités, dont le symbole est Versailles-Saint-Quentin.
En parallèle, le budget de l’ANR alloué aux projets est passé de plus de 600 millions d’euros en 2010 à moins de 500 actuellement, le nombre de projets financés de 1 300 en 2010 à très certainement moins de 1 000 en 2014.
A cela s’ajoutent les effets liés à la loi Sauvadet de 2012. Cette loi favorisant la titularisation des personnels contractuels après six ans passés dans la fonction publique a jeté un froid dans certains laboratoires et certaines disciplines scientifiques (notamment celles qui recrutaient plutôt à 35 ans qu’à 31…). Les budgets ne permettant que rarement ces « cdisations » non planifiées, les ressources humaines des organismes sont aujourd’hui très craintives et rendent difficile l’embauche d’un post-doctorant dès la quatrième voire la troisième année.
La baisse du budget de l’ANR, censée s’accompagner d’une revalorisation des financements récurrents, a d’ailleurs probablement et principalement servi àfinancer ces titularisations non anticipées dans la mesure où l’argent n’est en tout cas pas arrivé jusqu’aux laboratoires. En lien avec cette loi Sauvadet et cette résorption de la « précarité » dans l’enseignement supérieur et la recherche, on peut également souligner que les règles de l’ANR concernant l’embauche de contractuels ont été rendues plus drastiques : il faut aujourd’hui trois permanents à temps plein sur un projet pour recruter un non-permanent à temps plein pour la durée de celui-ci.
AU MILIEU DU GUÉ
Nous nous trouvons donc aujourd’hui au milieu du gué, et l’on se demande si nous y sommes arrivés de façon réfléchie ou simplement par suite de tiraillements successifs et aléatoires vers les directions opposées prises par ces deux systèmes.
Des financements récurrents qui ont quasiment disparu mais également des financements sur projets qui s’effondrent. Des permanents qui ont de moins en moins de temps à consacrer à la pratique de la recherche pour en passer de plus en plus à la gestion (de projets, de groupes) pour les plus talentueux ou chanceux, et à la lutte pour l’obtention des crédits nécessaires à leur activité pour les autres. Et de moins en moins de docteurs non permanents dans les laboratoires et de plus en plus de difficultés à les financer ou plus simplement à les attirer (puisqu’on ne peut honnêtement plus rien leur promettre et que les salaires proposés ne sont toujours pas compétitifs).
La question se pose alors simplement : qui, en dehors des quelques doctorants passionnés qui pourront encore être financés (et alors que les difficultés d’insertion des docteurs sont toujours récurrentes en France, ce qui est tout sauf une incitation à envisager le doctorat comme un choix de carrière judicieux), fera demain de la recherche dans les laboratoires publics en France ?
Point de vue : LE MONDE SCIENCE ET TECHNO | 05.05.2014 à 17h00 • Mis à jour le 07.05.2014 à 13h57

A l’occasion de son stage d’observation, la jeune Marieke nous fait partager ses remarques suite à son passage sur la plateforme de génomique Pegase-biosciences. Ce point de vue peut permettre à d’autres jeunes lycéens ayant une inclination pour le monde de la recherche en sciences de la vie de confirmer ou d’infirmer leur motivation pour débuter un parcours scientifique. Comme quoi, parfois, ces stages d’observation peuvent être un peu plus qu’une figure imposée.
Laissons la parole à Marieke !
Dans le cadre de mon année de 1ère S, j’ai effectué un stage d’observation d’une semaine, à l’Institut Pasteur de Lille. Ayant un attrait pour les sciences, je voulais découvrir le milieu de la recherche. J’avais quelques a priori que je voulais confirmer ou non, savoir si ce domaine me convient. Pendant ce stage, j’ai échangé avec 18 scientifiques (10 femmes, 8 hommes) travaillant à l’Institut Pasteur de Lille en recherche biologique. Je me suis intéressée au groupe BDPEE (axé parasitologie), et au groupe Pégase (axé génomique). En amont du stage, je me posais plusieurs questions : quels sont les métiers relatifs à la recherche, pour quel profil particulier, quelles formations pour y accéder ?
Commençons par la conclusion ! De tous ces entretiens, il en est ressorti un constat global : quelle que soit la profession exercée, le secteur de la recherche regroupe des personnes passionnées et curieuses, pour qui la science semble être une vocation. Le domaine de la recherche demande également de la rigueur, de l’organisation et un esprit de synthèse. Ces qualités sont fondamentales pour la quasi-totalité des chercheurs.
Age moyen : 30 ans
Répartition : 10 femmes (55%), 8 hommes (45%)
Nombre moyen d’années post bac : environ 5
Emploi actuel est le premier : 80%
En réalisant les interviews, j’ai constaté qu’il existait des professions différentes au sein de la recherche. Pour faciliter leur description, je les ai rassemblées par domaine.
A- Biologie « in vitro [1] »
(i) Enseignement – chercheur : 50 % recherche, 50 % enseignement
(maître de conférences, professeur)
Les 2 fonctions sont très complémentaires et se renforcent mutuellement. Les maîtres de conférences actualisent leur cours grâce aux publications et leurs travaux de recherche permettent de garder un pied dans la communauté scientifique. La transmission, le partage du savoir apportent une vraie satisfaction, complétée par le projet de recherche. Avec la HDR (Habilitation à Diriger les Recherches), les maîtres de conférences et professeurs encadrent les doctorants. Enfin, malgré peu de postes disponibles, le statut de fonctionnaire est synonyme de sécurité d’emploi.
La volonté de vouloir poursuivre des recherches, la préparation des cours, la constitution de la bibliographie (c’est-à-dire la mise à jour en continu des connaissances liées à la thématique d’étude), la recherche de moyens pour financer le projet de recherche, nécessite beaucoup de temps. Cependant cet investissement parait secondaire si l’avidité de connaissances et la curiosité sont au rendez-vous.
(ii) Gestion du laboratoire et des projets de recherche : 50 % recherche, 50 % gestionnaire
(chargé de recherche, chargé d’études)
La gestion du laboratoire implique la mise en place et l’encadrement de projets de recherche. Le chargé de recherche développe et teste des idées par la conceptualisation de protocoles, ce qui demande une capacité de concentration importante. Il encadre l’équipe technique en gérant les relations avec le reste de l’entreprise ou de l’équipe de recherche.
(iii) Equipe Technique et Ingénieur : 50 % recherche, 50 % production
(technicien, ingénieur d’étude, ingénieur de recherche)
Ces personnes collaborent aux découvertes : autant sur la mise en place des protocoles que sur les manipulations. Elles prennent part aux discussions pour enrichir et interpréter les hypothèses de travail. Le renouvellement des technologies et des techniques enrichit les connaissances, et ne laisse pas de place à la monotonie. L’autonomie et les responsabilités sont également un aspect plaisant de cette fonction. Cette profession permet une évolution de carrière.
B- Biologie « in silico [2] », Bio-informatique
(bio-informaticien, ingénieur de recherche, analyste en génétique quantitative)
Les travaux en génomique [3] génèrent un nombre très important de données qu’il faut traiter et analyser afin que les biologistes « in vitro » puissent dégager du sens, l’équipe informatique interprète les données générées, ou crée des algorithmes permettant leur interprétation. Par l’informatique, elle répond à des questions d’ordre biologique, ce qui confère un aspect pluridisciplinaire à ce domaine. L’analyste met en évidence l’essence des résultats à l’aide d’études statistiques. Le renouvellement des problématiques très diverses confère un aspect non répétitif très apprécié.
Cette science s’adresse à des personnes rigoureuses, entreprenantes, ayant un attrait pour les mathématiques (logique, analyse, statistiques).
C’est un métier de plus en plus porteur, car, grâce aux nouvelles technologies (génotypage ou séquençage à haut-débit), les données générées sont de plus en plus importantes et conséquentes.
Lorsque je me suis intéressée à la formation, je me suis rendu compte qu’elles étaient très variées. En effet, il n’y a pas une formation type, classique pour accéder au monde de la recherche. La plupart des interviewés ont un parcours atypique et on constate que les passerelles sont multiples.
Cependant, parmi les différentes orientations, certaines voies sont plus souvent empruntées :
BTS – formation concrète délivrant des bases solides notamment pour les manipulations.
Fac (Licence et Master) – enseignement général, pluridisciplinaire. Recommandé pour les étudiants qui hésitent sur leur orientation. Cependant, cette formation nécessite une motivation non négligeable.
Doctorat – 3 ans consacrés à la réalisation d’une thèse. Il peut éventuellement être « surdiplômant ».
Je trouve que c’est un milieu propice à la réflexion scientifique, passionnant, idéal si on aime s’investir dans des projets. Certains enseignants – chercheurs sont capables de passer 8 ans sur un seul parasite, le Cryptosporidium par exemple.
J’ai été particulièrement impressionnée par la quantité d’anglais. La maîtrise de l’anglais scientifique est une aptitude vitale en recherche pour communiquer lors des congrès, pour lire et rédiger des protocoles et des articles.
Je ne savais pas du tout que la publication avait une place si importante dans le quotidien des chercheurs. A la fois pour se tenir informé des avancées, pour communiquer ses découvertes, pour faciliter l’obtention de moyens financiers, pour critiquer les travaux de recherches, pour se faire reconnaître, les scientifiques publient des articles, souvent en anglais, dans la presse scientifique. La publication est nécessaire pour trouver des financements. Ainsi, une sorte de cercle vicieux s’installe progressivement : il faut des financements pour publier et pour obtenir ces financements, il faut publier. Or plus le journal à une audience importante, plus il a un facteur d’impact élevé et plus il est difficile de publier dans ce journal. Cette pratique, fondamentalement utile au sein de la communauté scientifique, est cependant contestée. Par exemple, au-delà des 192 heures de cours obligatoires, il est demandé aux maîtres de conférences de produire un article par an. Cela peut éventuellement déboucher sur une pratique de la publication déraisonnée et excessive.
[1] In vitro : locution latine signifiant au laboratoire, par extension ici cela désigne les expériences menées au laboratoire
[2] In silico : locution latine signifiant par ordinateur
[3] La génomique est une discipline de la biologie moderne. Elle étudie le fonctionnement d’un organisme, d’un organe, d’un cancer, etc. à l’échelle du génome, et non plus limitée à celle d’un seul gène
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages