L’un des pionniers des séquences nucléique et protéique est décédé, il y a peu, le 19 novembre 2013, à l’âge de 95 ans. Il avait commencé sa thèse en octobre 1940 avec pour projet de rechercher une protéine consommable purifiée d’herbe (relativement abondante dans le royaume de Georges VI). C’est, au final, sous la direction d’Albert Neuberger (qui avait fuit l’Allemagne avec l’arrivée d’Hitler au pouvoir) que Frederick Sanger devient docteur en 1943.
Les deux hommes se respectaient, à propos de « son maître » Albert, Frédérick Sanger avait déclaré : « c’est Neuberger, qui le premier, m’a appris à faire de la recherche, à la fois techniquement et comme un mode de vie, et je lui dois beaucoup. »
L’homme qui a donné son nom à une technique de séquençage des nucléotides ouvrant la voie à un pan entier d’une science, à un institut de recherche (le Wellcome Trust Sanger Institute) employant plus de 900 personnes est une fierté (jamais anoblie) de l’empire britannique : une des quatre personnes à avoir reçu deux prix Nobel (prix Nobel de chimie). Son premier prix Nobel de chimie pour le séquençage de l’insuline (1958) sanctionne un travail de 10 ans. Rejoignant Crick (le penseur), au laboratoire de biologie moléculaire du Conseil médical de la Recherche à Cambridge, Sanger (le fonceur) se met à développer une technique de séquençage de l’ADN. Ce travail lui apportera son second prix Nobel en 1980.
« Quand j’étais jeune, mon père me disait que les deux activités les plus intéressantes dans la vie sont la recherche de la vérité et de la beauté, et je crois qu’Alfred Nobel a dû ressentir la même chose quand il a donné ces prix pour la littérature et les sciences.«

Un document reprenant le nécessaire à savoir sur l’oeuvre de Frederick Sanger (réalisé par Richard Daniellou, Enseignant-chercheur, ENSCR) est disponible au format pdf.
L’image en tête de cet article est issue d’un portrait de Frederick Sanger réalisé par Paula A. MacArthur (huile sur toile : 1991, collection: National Portrait Gallery de Londres).
Bertrand Jordan a eu son doctorat en physique nucléaire à 1965 et a très bien fait de se convertir à la biologie moléculaire. Entre autres choses, il a réalisé en 1982 l’isolement du premier gène d’histocompatibilité humain, et en 2000, il a fondé la Génopole de Marseille. Bertrand Jordan est membre de l’Organisation européenne de biologie moléculaire (EMBO) ainsi que de l’organisation internationale HUGO (Human Genome Organisation).
Mais surtout, et c’est ce qui nous intéresse ici, l’auteur de plus de 150 publications scientifiques fait œuvre de vulgarisation et offre depuis plus de dix ans, des chroniques génomiques publiées au sein de la revue Médecine / Sciences. Ces chroniques sont accessibles gratuitement (vous pouvez y accéder directement sur le site de www.medecinesciences.org ou en cliquant sur l’image ci dessus en haut à gauche).
On peut vous conseiller de lire le point de vue de Bertrand Jordan sur les séquenceurs haut-débit dans sa chronique de mars 2010 intitulée : le boom des séquenceurs nouvelle génération, sans oublier le point de vue partagé (parfois longtemps après lui) par un nombre croissant de chercheurs sur les GWAS, dans sa chronique de mai 2009 : le déclin de l’empire des GWAS. C’est là tout l’intérêt de ces chroniques qui, bénéficiant d’un propos clair, simple, construit, argumenté et rare, permettent de prendre un peu de recul sur les applications d’une science qui n’ont pas toujours tenu leurs promesses. Ces chroniques peuvent être l’occasion de faire tomber le masque, de dégonfler l’air de ces vessies qui se prennent parfois pour des lanternes.
Bertrand Jordan, chroniqueur mais aussi auteur de quelques livres aux titres évocateurs, parfois mémorablement provocateurs :
Voyage autour du Génome : le tour du monde en 80 labos. Editions Inserm/John Libbey, Paris, 1993
Voyage au pays des gènes. Editions. Les Belles Lettres/ Inserm, Paris, 1995
Génétique et génome, la fin de l’innocence. Editions Flammarion, Paris, 1996
Les imposteurs de la génétique. Editions du Seuil, Paris, 2000 (Prix Roberval Grand Public 2000)
Le chant d’amour des concombres de mer. Editions du Seuil, Paris, 2002
Les marchands de clones. Editions du Seuil, Paris, 2003
Chroniques d’une séquence annoncée. Editions EDK, Paris, 2003
Le clonage, fantasmes et réalité. Essentiel Milan, Editions Milan, 2004
Thérapie génique : espoir ou illusion ? Editions Odile Jacob, Paris, 2007
L’humanité au pluriel : la génétique et les questions de race. Le Seuil, collection Sciences ouvertes, Paris, 2008
Autisme, le gène introuvable : Le Seuil, Paris, 2012
 Tout commence ici par un film de piètre qualité (je ne crois pas qu’il en existe une version française), Who the #$&% Is Jackson Pollock ? Ce film retrace l’histoire d’une femme, Terri Horton, de 73 ans, ancienne conductrice de camion long courrier qui a acheté une toile dans un magasin d’aubaines pour 5 USD. Il s’avère que cette toile a été attribuée à Jackson Pollock et en demande 50 millions de USD. Le film dépeint les tentatives de Horton pour authentifier et vendre la peinture comme une œuvre originale de Pollock. Son authenticité est douteuse, car la peinture a été achetée dans un magasin d’aubaines, n’est pas signé, et est sans provenance. Le principal problème avec la peinture, c’est qu’elle « n’a pas l’âme d’un Pollock », selon les collectionneurs. En outre, Pollock avait de nombreux imitateurs de son vivant. C’est là qu’intervient un spécialiste en techniques légales d’authentification, iconoclaste et donc décrié, Peter Paul Biro. Un document pdf (disponible ici) reprend toute la palette mise à disposition de ce dernier pour permettre une authentification légale. Tout part d’une observation « microscopique » dont l’objectif est de rechercher des traces éventuelles de l’artiste (poils, cheveux, empreintes digitales etc.). Les pages 13 et 14 abordent la technique mise en œuvre.
Tout commence ici par un film de piètre qualité (je ne crois pas qu’il en existe une version française), Who the #$&% Is Jackson Pollock ? Ce film retrace l’histoire d’une femme, Terri Horton, de 73 ans, ancienne conductrice de camion long courrier qui a acheté une toile dans un magasin d’aubaines pour 5 USD. Il s’avère que cette toile a été attribuée à Jackson Pollock et en demande 50 millions de USD. Le film dépeint les tentatives de Horton pour authentifier et vendre la peinture comme une œuvre originale de Pollock. Son authenticité est douteuse, car la peinture a été achetée dans un magasin d’aubaines, n’est pas signé, et est sans provenance. Le principal problème avec la peinture, c’est qu’elle « n’a pas l’âme d’un Pollock », selon les collectionneurs. En outre, Pollock avait de nombreux imitateurs de son vivant. C’est là qu’intervient un spécialiste en techniques légales d’authentification, iconoclaste et donc décrié, Peter Paul Biro. Un document pdf (disponible ici) reprend toute la palette mise à disposition de ce dernier pour permettre une authentification légale. Tout part d’une observation « microscopique » dont l’objectif est de rechercher des traces éventuelles de l’artiste (poils, cheveux, empreintes digitales etc.). Les pages 13 et 14 abordent la technique mise en œuvre.
Même si l’attribution d’un tableau à son auteur par l’intermédiaire de la caractérisation singulière de l’ADN de ce dernier restera anecdotique, le besoin d’identification et d’authentification peuvent être un marché potentiel pour des sociétés de biotechnologie.
Ce sujet avait été abordé dans notre article « l’ADN, nouveau support de l’information numérique » qui montrait l’utilisation faite de cette molécule en guise de code barre pour traquer les pétroliers dégazant le reliquat de leur cargaison (je serais curieux de connaître le nombre de capitaines mazouteurs confondus par cette méthode).

Ainsi, l’une de ces sociétés de biotechnologie, la société Applied DNA Sciences, Inc, propose des produits d’aide à l’identification et l’authentification. L’ADN devient un code barre censé être inviolable, un élément visant à protéger de la contrefaçon des processeurs, des vêtements, des spiritueux. Une autre application vise à sécuriser les transferts de fonds (un nouveau moyen pour marquer et tenter de suivre les billets marqués).
Cette société surfant sur le besoin de sécurité et de dissuasion développe un spray fluorescent chargé en molécules d’ADN. Mise en situation… Un voleur braque un bijoutier à l’aide d’une arme de poing (cf. schéma ci-dessous).
Le bijoutier a deux choix :
. user de violence et de mesures coercitives pouvant flirter avec le délit
. oublier qu’un revolver est pointé sur lui, appuyer sur un bouton rouge qui asperge l’agresseur d’un spray fluorescent génétiquement marqué… prier pour que cela suffise à mettre en fuite le braqueur.
On l’aura compris, il ne s’agit pas là de l’idée du siècle. L’ADN est mis à toutes les sauces, de l’authentification à l’arme absolue de dissuasion pour tout braqueur effrayé par l’association d' »idées » : ADN + experts police + haute technologie = preuve = prison.

L’ADN comme moyen d’identifier l’auteur d’un tableau -comme quelque chose dans la continuité des empreintes digitales de Vinci laissées lors de son brillant sfumato– au molecular fingerprinting des peintres contemporains en passant par l’absurde spray anti-braqueur qui laisse esquisser un sourire moqueur, la molécule support de l’information génétique est d’ores et déjà la base d’un business associé au besoin d’authentification sans cesse plus important…
Et voici le programme en anglais:
9H00 Registration / coffee
9H15 Introduction of the day
9H30 KEYNOTE LECTURE 1: Joris Veltman (N): EXOME SEQUENCING IDENTIFIES DISEASE CAUSING DE NOVO MUTATIONS, Department of Human Genetics – Radboud University Nijmegen Medical Centre – Nijmegen, The Netherlands
10H15 Jean Muller (F): MULTI-GENE RESEQUENCING FOR MOLECULAR DIAGNOSIS OF OBESITY, Integrative Genomics and Bioinformatic Laboratory (IGBMC) – Genetic Diagnostic Laboratory – Strasbourg, France
10H45 Coffee break
11H15 Amélie Bonnefond (F): NEW TECHNOLOGIES IN GENE RESEQUENCING AND IN METHYLOME ANALYSIS, Department of Genomics and Metabolic Diseases – CNRS UMR8199 & Institut de Biologie de Lille – Institut Pasteur – Lille, France
11H45 Didier Goidin (F): LAST DEVELOPMENT OF AGILENT NGS SOLUTIONS, Agilent R & D – Les Ulis, France
12H15 Sean Humphray (UK): SEQUENCING FOR GENOMIC MEDICINE, Illumina R & D – Chesterford, United Kingdom
12H45 Lunch
14H00 Ludovic Dupont (F): FRENCH FOUNDATION FOR RARE DISEASES: STRUCTURE, MISSIONS, ACHIEVEMENTS AFTER ONE YEAR OF EXISTENCE French Foundation for rare diseases – Paris, France
14H15 KEYNOTE LECTURE 2: Nicolas Lévy (F): THE GENOMICS AND POST GENOMICS OF RARE DISEASES: THE PROGERIA CASE, Department of Medical and Development Genetics Inserm U910 Faculté de médecine de la Timone & Department of Medical Genetics – Hôpital d’enfants de la La Timone – Marseille, France
15H00 Wim Van Criekinge (B): NEXT GENERATION EPIGENETIC PROFILING, Center in Pharmaco(epi)genomics NXTGNT (www.nxtgnt.com) Department of Mathematical Modelling, statistics and Bioinformatics – Ghent University – Gent, Belgium
15H30 Alexandra Lespagnol (F): TARGETED HIGH-THROUGHPUT SEQUENCING FOR SOMATIC ANALYSIS ON FORMALIN-FIXED, PARAFFIN- EMBEDDED (FFPE) TUMOR SAMPLES, Plateforme Hospitalière de Génétique Moléculaire des Cancers, CHU Pontchaillou – Rennes, France
16H00 Coffee break
16H30 KEYNOTE LECTURE 3: Jacques Beckmann (S): COPY NUMBER VARIANTS AND THEIR ROLE IN DISEASES
17H15 Conclusive remarks

 Un bon nombre de thésards abordent fébrilement la période de la fin d’été. Un été passé à bronzer à l’ombre des néons clignotants des laboratoires et arpentant des bureaux devenus déserts, on imagine le thésard comme une ombre fantomatique à la recherche d’une machine à café… une vision, un peu cliché ? Quoi qu’il en soit, un site internet vous permettra de travailler légèrement votre anglais et de faire preuve d’un peu d’autodérision : il s’agit du très connu et parfois même brillant : PhD comics (www.phdcomics.com). Il s’agit de brasser les névroses des aspirants chercheurs pour porter leur point de vue sur ce futur métier pollué par tant d’images d’Epinal.
Un bon nombre de thésards abordent fébrilement la période de la fin d’été. Un été passé à bronzer à l’ombre des néons clignotants des laboratoires et arpentant des bureaux devenus déserts, on imagine le thésard comme une ombre fantomatique à la recherche d’une machine à café… une vision, un peu cliché ? Quoi qu’il en soit, un site internet vous permettra de travailler légèrement votre anglais et de faire preuve d’un peu d’autodérision : il s’agit du très connu et parfois même brillant : PhD comics (www.phdcomics.com). Il s’agit de brasser les névroses des aspirants chercheurs pour porter leur point de vue sur ce futur métier pollué par tant d’images d’Epinal.




Ainsi, le PhD -dans le contexte de PhD Comics l’abréviation est mise pour Piled Higher and Deeper en lieu et place de Philosophiæ doctor (l’intitulé du diplôme de docteur dans le système universitaire anglo-saxon) – est un parcours semé d’embûches entre désillusions, travail de força et stress d’un avenir pour lequel le diplôme de docteur ne semble plus un passeport pour un travail universitaire intellectuellement enrichissant et financièrement roboratif.

Jorge Cham, l’auteur des textes et dessins, connait le sujet ! Il a, en effet, débuté cette bande dessinée alors qu’il était étudiant à Standford où il visait un doctorat en génie mécanique. Comme le monsieur est dessinateur de comics, universitaire et américain, il s’est engagé dans un vaste tour des universités (plus d’un centaine) pour présenter son « the Power of Procrastination » (le pouvoir de la procrastination). Avec cette thèse (à prendre ici dans les deux sens), Jorge Cham, contrecarre les discours tenus par les coachs certainement plus cupides que psychologues, adeptes du développement personnel à l’anglo-saxonne. En effet, lors de sa présentation, l’auteur de PhD Comics démontre que le fait de remettre à plus tard est en général quelque chose de bénéfique pour l’élaboration d’un concept (ce en quoi il mérite l’appellation primitive de PhD !).
Ceci étant, PhD Comics met le doigt sur une réalité tangible de notre côté de l’Atlantique : une espèce de « paupérisation » du diplôme et avant cela du statut des étudiants se destinant à l’accomplissement d’une thèse… des éléments faisant tellement partie du panorama qu’on ne les voit plus (sur le sujet lire : Thésard, une vie de loser – article de Libération du 20 mars 2007)
Encore que pour ce qui nous intéresse ici, les thésards des Sciences du vivant peuvent être enviés par ceux des Sciences humaines qui sont entrés avant eux dans cette spirale de la lose. Donc la caricature proposée par PhD Comics nous amène à nous poser des questions quant au statut et au devenir d’étudiants parfois désabusés, mais aussi à la perception de la science comme objet d’accomplissement professionnel (plus que comme discipline intellectuelle). Ainsi, il en va du morceau choisi ci-dessous.

A la croisée de la biologie, de la biochimie, de l’électronique, de la mécanique des fluidiques et de la thermodynamique, de nouveaux outils se préparent. Ceux-ci viendront peut être trouver des applications dans les laboratoires de génomique mais au-delà de cette perspective, ils pourraient trouver des applications au niveau des professionnels de santé eux-mêmes avant d’être accessibles aux clients-patients-citoyens directement.
Les Lab-on-Chips (ou Laboratoire sur puce) sont un concept qui a émergé dans les années 90 et qui au sein des laboratoires de biologie moléculaire a trouvé une application largement répandue sous la forme des puces Agilent consommables pour BioAnalyzer. La technologie d’Agilent, basée sur une électrophorèse ou une cytométrie de flux microfluidique, offre un outil de contrôle de la quantité, qualité (distribution de taille, intégrité des ARNs extraits etc.) des molécules considérées. Même si les puces Agilent sont affublées du qualificatif de « Lab-on-Chip« , elles nécessitent un appareillage dédié et bien entendu un PC relié. L’outil est encore loin du modèle rêvé, c’est-à-dire complètement autonome (en une version « stand alone »).
L’émergence de ce type de laboratoires a été, en partie, permise grâce à des budgets alloués à la lutte contre le bioterrorisme. En effet, des laboratoires qui tiennent dans une poche de treillis, qui peuvent être employés sur n’importe quel théâtre d’opération, peuvent venir parfaire le paquetage des experts en sécurité intérieure. Les laboratoires Sandia (Sandia National Laboratories) font partie des principaux laboratoires nationaux du département de l’Énergie des États-Unis et sont des pionniers de ces applications avec les développements réalisés par MicroChemLab qui est actif depuis 1988 sur ce type de développements.
Aujourd’hui les laboratoires unipersonnels de diagnostic embarqués sont devenus très populaires, pour des applications liées à des analyses biochimiques. L’un des exemples les plus fameux concerne le BG-Star (Sanofi Diabète) permettant de réaliser en quelques secondes, très aisément, des lectures de glycémie. L’outil est basé sur le principe de l’électrochimie dynamique où une bandelette jetable accueille une goutte de sang qui vient activer une glucose oxydase.
Les avantages de ce type de technologies :
– Une faible consommation de volume de fluide, en raison des volumes internes faibles des puces, ce qui minimise la quantité de déchets, baisse des coûts des réactifs onéreux et surtout minimise la quantité nécessaire d’échantillon de départ
– Une rapidité d’exécution où l’efficacité des réactions biochimiques est permise par la miniaturisation de l’outil, en effet la taille du dispositif ne nécessite qu’une faible quantité d’énergie pour fonctionner
– Un meilleur contrôle du processus en raison d’une réponse rapide du système (par exemple, le contrôle thermique pour des réactions chimiques exothermiques)
– Une parallélisation massive due à la compacité, qui permet l’analyse à haut-débit
– Une réduction des coûts de fabrication, permettant une utilisation de puces jetables, un processus qui deviendra rentable s’il est fabriqué en masse
– Une plateforme plus sûre pour les produits chimiques, les études radioactives ou biologiques en raison de la grande intégration du système (souvent en système clos), des faibles quantités de réactifs et d’échantillons nécessaires
Les vertus de la miniaturisation ne nécessitent pas plus amples apologies ! Depuis quelques années l’idée d’un laboratoire qui tient au creux de la main a réellement commencé à voir le jour.
Des exemples de ces laboratoires portatifs commencent à poindre dans des versions de plus en plus intégrées.
Ainsi la technologie Mondrian développée par NuGEN, technologie basée sur de la microfluidique digitale, permet de simplifier les étapes de constitution de librairie de séquences haut-débit.
Destinagenomics propose un système analytique ne faisant pas intervenir d’enzymes. Ce système fait intervenir des sondes interrogatives de mutations que la société souhaite embarquer dans des puces : la preuve du concept a été publiée par Pernagallo et al. (Sensors (Basel). 2012).
http://youtu.be/k-NnBS2xDLo
Panasonic, le groupe japonais, numéro 4 mondial de l’électronique, en partenariat avec la société belge, Imec, propose d’embarquer dans une puce d’ 1 cm2 : une pompe permettant aux fluides de circuler, une unité d’extraction d’ADN à partir de la goutte de sang prélevée, un thermocycleur, une unité de purification de produits de PCR, et un système de détection de SNPs.

Le concept est séduisant, mais pour être viable et concurrencer les coûts de génotypages basse densité qui sont relativement bas (quelques euros / marqueur), les concepteurs des Laboratoires sur Puces misent donc sur une large diffusion de leur produit. Ainsi la production de masse impacte drastiquement le coût de production qui lui-même est impacté sur le prix vente… un prix de vente attractif et une simplicité d’utilisation qui dope les ventes… et donc fait baisser les coûts de production…
Le pari réside dans le fait d’amorcer ce cercle vertueux, en quelque sorte chaque opérateur du marché – au niveau d’une application donnée- souhaite assommer la concurrence ! Une course au développement est donc lancée.
Revenons à la science ! Vous trouverez ici le document pdf. présentant la technologie de Panasonic/Imec,
Concernant l’application de séquençage, Oxford Nanopore proposera une « clé USB » capable de séquencer, le Minion.
Dans le futur il est envisageable, d’imaginer un outil combinant la technologie de séquençage de 3ème génération, d’un Oxford Nanopore, par exemple, avec en amont un dispositif intégrant (à l’instar du projet de Panasonic), une micro-pompe, un réseau microfluidique permettant d’alimenter en échantillon et réactifs les différents éléments permettant une extraction d’ADN/ARN, la constitution d’une librairie, une purification…
Parle-t’on ici de science fiction ? Pour un début de réponse, il faut comprendre que séparément toutes ces solutions technologiques existent déjà.
Pour les plus férus de laboratoires miniaturisés, un journal scientifique est là pour eux, explicitement nommé : Lab on a Chip.
 23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
Début 2013, le produit coûte : 99 USD auxquels il faut ajouter 79,95 USD pour les frais de port (ces frais limitent l’accès au vieux continent à 23andMe), la note se monte donc pour un Français à 137,70 € (mi 2011, le prix était de 200 €). Il est facile dorénavant de comprendre que, les volumes des puces Illumina grimpant, la part du coût allouée aux consommables a baissé, mais que la distribution web a ses limites… la poste.
Tests génomiques ?
Le test génétique que propose 23andMe permet :
– d’estimer les facteurs de risques d’être, un jour, atteint d’une maladie à composante majoritairement génétique
– d’estimer vos aptitudes physiques et intellectuelles (en présupposant que la variabilité génétique de ces traits contribue fortement auxdits traits estimés)

– de déterminer son origine géographique, ainsi que, pour le folklore, de quel personnage célèbre dont 23andMe possède le génotype, vous vous rapprochez le plus (sur le ce point lire notre article : A la recherche du chromosome perdu : aux sources des hominidés)
– enfin, 23andMe accumule des génotypages venant alimenter une base de données associée à une population de référence et ainsi accroître l’information génomique disponible pour d’éventuelles recherches (à mener par d’autres…), tout cela financé par les clients de 23andMe… il fallait y penser et oser, c’est chose faite !
Principe ?
Contrairement à un amalgame souvent rencontré, la société californienne n’utilise que très peu le séquençage pour son produit phare. Le principe réside dans l’extraction de l’ADN contenu dans les cellules de la salive, ADN qui fera l’objet d’un génotypage haut-débit permettant de cartographier le génome du client. 23andMe ne fait que compiler les données de GWAS (Genome Wide Association Studies), les études d’association publiées et agglomérées plus ou moins subtilement. Ce type d’études consiste en l’association de profils génétiques à des phénotypes caractérisés. En outre, 23andMe exploite la connaissance des marqueurs en Déséquilibre de Liaison avec une maladie ou une aptitude, le tout en concaténant l’information scientifique publique.
Des témoignages de clients pour en convaincre de nouveaux
Depuis le début 2013, plusieurs témoignages ont été utilisés par la presse pour décrire ce que proposait 23andMe, une publicité à moindre frais. Celui de Slate relate l’histoire de Jill Uchiyama, une personne qui a été adoptée à la naissance et qui à l’âge de 41 ans par l’intermédiaire des services proposés par 23ansMe a retrouvé quelques cousins plus ou moins proches (300 cousins) et en a rencontré un… un manière de recréer de simili-liens de sang. Un autre présent sur le site Quora.com témoigne du cas d’un père découvrant qu’il avait des risques de développer une intolérance au gluten alors que des praticiens ont perdu un temps infini à déterminer l’étiologie de la maladie cœliaque dont souffrait sa fille. Autant de témoignages de nature à convaincre de futurs clients !
Un accueil scientifique mitigé
Un premier article de Jane Kaye « The regulation of direct to consumer genetic tests » (Human Molecular Genetics, 2008), s’inquiète de la perte de contrôle des professionnels de santé, de l’évolution de la législation et de la confidentialité des données transitant par ces nouvelles sociétés de services. L’auteure appelle de ses vœux une régulation de cette « génomique récréative » où l’on peut partager sur les réseaux sociaux son potentiel génétique, ses risques de développer une maladie ou encore rechercher des « cousins » potentiels.
Deux ans après ce premier article, celui de Barbara Prainsack et Howard Wolinsky (Future Medicine, 2010) « Direct-to-consumer genome testing: opportunities for pharmacogenomics research ? » pondère le propos. Dans cet article, les auteurs ne s’inquiètent plus vraiment de l’existence d’un système divulguant des informations génomiques directement au consommateur sans passer par la case du prescripteur médical, mais s’interrogent sur les bénéfices que la recherche scientifique pourrait tirer de ces génotypages payés par les consommateurs eux-mêmes. Entrerait-on dans l’ère du libéralisme génomique ? L’article de Future Medicine développe l’aspect crowdsourcing (en français collaborat ou externalisation ouverte) en s’inquiétant légèrement tout de même d’un problème majeur : le respect de la vie privée…
Une inquiétude majeure
A une époque où des services étatiques développent des projets tels que PRISM, l’outil de surveillance du web utilisé par la NSA ou d’autres que la DGSE utiliserait, le consommateur de la génomique personnalisée pourrait et devrait s’inquiéter de la confidentialité des données génomiques le concernant. En effet, le web est le tuyau par lequel transite toutes les informations génomiques émanant des analyses de ces sociétés de service. La confidentialité des données semble être le nouveau frein au développement de ce type de marché.
Ce poste a vocation de passer en revue les différentes étapes de la technologie de séquençage à haut-débit du PGM Ion Torrent, depuis la préparation de la librairie jusqu’à l’obtention des données brutes. Ce survol technologique permet de rassembler un maximum d’explications techniques et de termes clefs associés à cette technologie. L’intérêt est de répondre essentiellement à une attente de néophytes ou futurs utilisateurs du PGM friands de retours d’expériences.
L’arbre de décision Ion Torrent s’étant considérablement développé, seuls les axes « Whole genome » et « Amplicon » serviront de support à l’ensemble de la présentation.
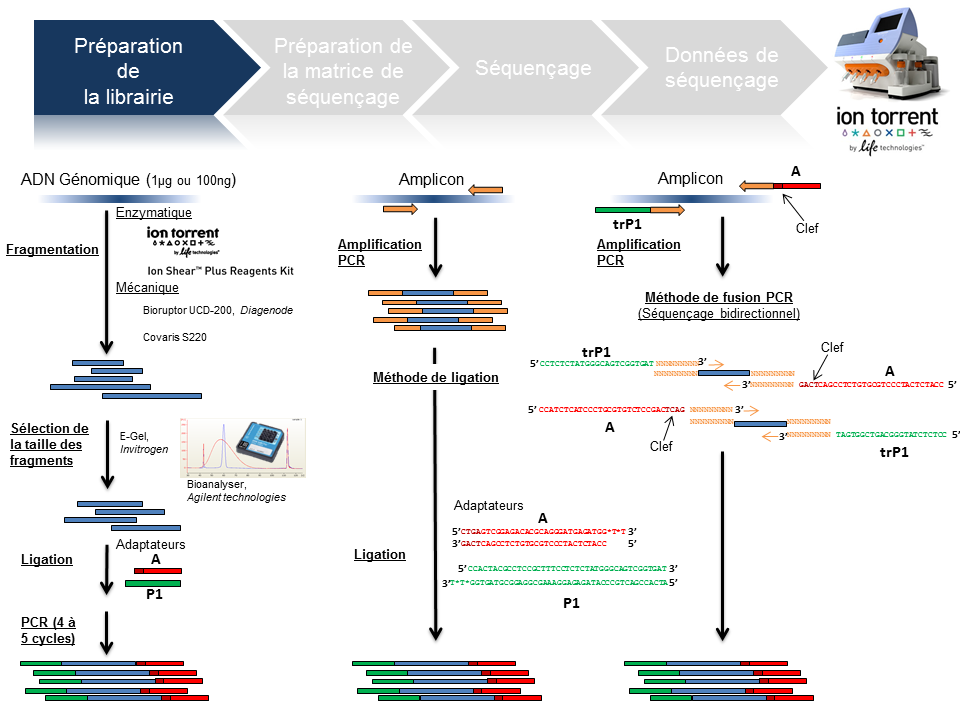
Préparation de la librairie
La finalité d’une préparation de librairie pour le PGM Ion Torrent est de lier aux fragments d’ADN à séquencer le couple d’adaptateurs A et P1. La taille médiane des fragments est variable et définie selon la chimie employée: 100, 200 , 300 ou 400 bases (Cf tableau ci-dessous).
Le traitement d’un échantillon d’ADN génomique débute par une étape de fragmentation mécanique ou enzymatique. Cette dernière présente l’avantage d’être considérablement plus rapide.
En amplicon-seq, la méthode pour flanquer les adaptateurs est double, par ligation ou par fusion PCR.
Par ailleurs, il est envisageable de traiter plusieurs échantillons en parallèle en utilisant des adaptateurs avec code barre ( En standard chez Life technologies: Au nombre de 96 pour les échantillons ADN et 16 pour les échantillons ARN).
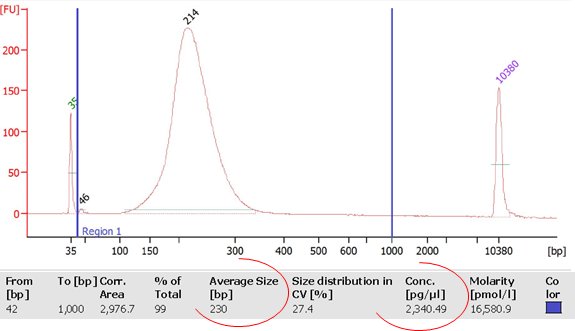
La librairie est monitorée sur puce DNA High sensitivity (Bioanalyzer, Agilent) avec comme objectifs de calculer sa concentration et d’identifier la taille moyenne des fragments qui la composent. Ces valeurs permettront ainsi de déterminer la concentration molaire de la libairie et d’y appliquer le facteur de dilution nécessaire pour favoriser le ratio idéal 1/1 (Fragment ADN de la librairie/Ion Sphere Particle) pour l’étape suivante de PCR en émulsion.
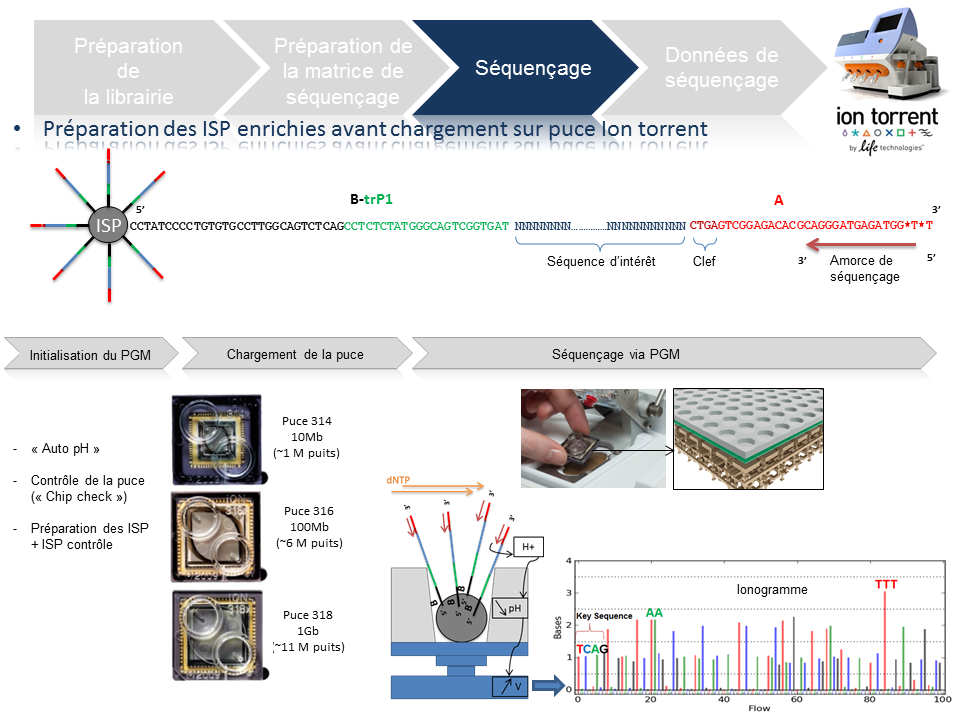
Préparation de la matrice de séquençage
Cette étape automatisée permet l’amplification clonale (OneTouch2) suivi d’un enrichissement en « Ion Sphere Particles » (ISPs) à la surface desquelles un fragment de librairie est amplifié (OneTouch ES).
L’amplification clonale est réalisée au cours de la PCR en émulsion (emPCR) et contribuera à atteindre un seuil de détection du signal nécessaire et suffisant au moment du séquençage. Malgré une optimisation du ratio 1/1 (ISP / Fragment ADN), plusieurs configurations de microréacteurs sont envisageables. Seule la configuration de monoclonalité est souhaitée car elle seule, est source de données de séquençage. Les autres configurations généreront des données qui seront filtrées lors de la primo-analyse par la « Torrent suite ».
L’amorce ePCR-A couplée à la biotine permettra l’enrichissement ultérieure par un système de capture sur billes liées à la streptavidine.
Séquençage
En amont de l’étape de séquençage, une initialisation du PGM est requise et permet notamment une homogénéisation des valeurs de pH ~ 7,8 au sein des différents réactifs de l’appareil (« Auto pH« ).
La matrice de séquençage couplée aux amorces de séquençage et à la polymérase est chargée sur la puce Ion Torrent selon un protocole bien spécifique. Les puces se déclinent selon 3 capacités de séquençage ( Chip 314 >10Mb, Chip 316 >100Mb, Chip 318 >1Gb). A noter qu’une version « v2 » pour chacune des puces précitées existe et est indispensable pour toute application de séquençage nécessitant la chimie 400. Le séquençage multiparallélisé revient donc au décryptage simultané des fragments ADN couplés aux ISP. A chaque polymérisation de nucléotides non modifiés, la libération d’ ions H+ entraine une variation de pH, elle même détectée au niveau de la couche mince (technologie des semi-conducteurs) située au fond de chaque puits. L’ensemble des données brutes générées est transcrits sous forme de ionogrammes.
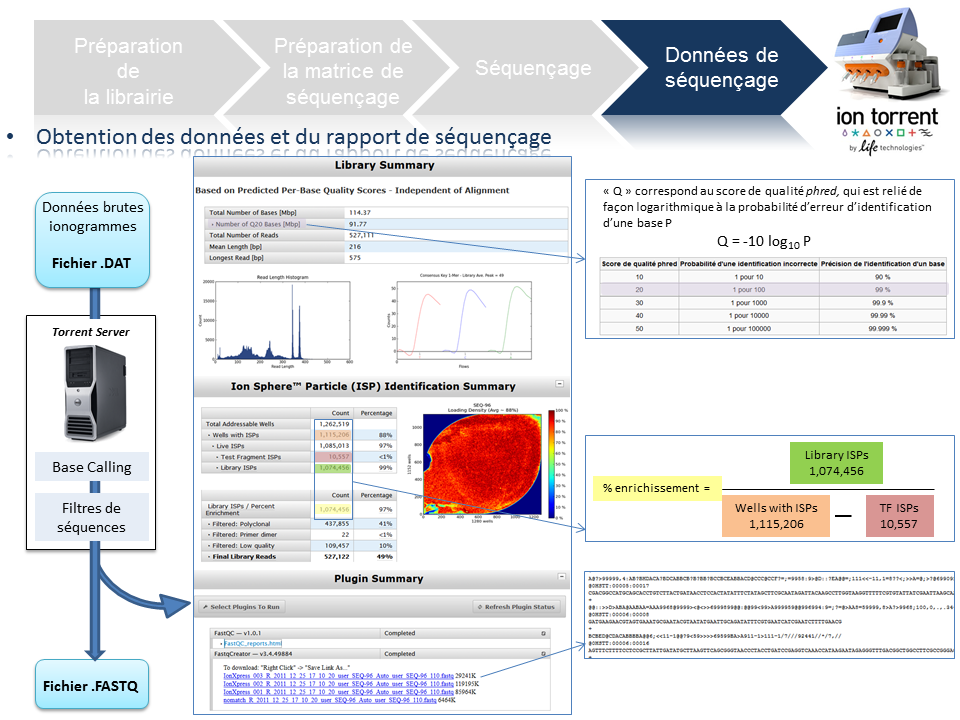
Données de séquençage
A l’issue du « run » de séquençage, le fichier .DAT regroupe l’ensemble des données brutes (ionogrammes). Ces fichiers sont transférés du PGM vers le Torrent Server. L’algorithme de « base calling » permet la conversion des données sous forme de lettres en séquences (A,T,C,G) formant le read (séquence au format fasta) associé à un score de qualité (Phred Score codé en ASCII), les deux types de données étant associés dans un fichier .FASTQ (qui tend à devenir le format de référence).
Un prétraitement est également appliqué sur la base des reads générés et qui équivaut au nombre d’ISPs vivantes ou « Live ISPs » (On parle d’ISPs vivantes pour les ISPs associées à la clef):
– trimming : élimination des adaptateurs et/ou portions de reads de mauvaise qualité
– filtres : élimination des « reads » de petites tailles, de mauvaise qualité, des polyclonaux
L’ensemble de ces informations est repris au travers du « report » généré à l’issue du séquençage et du prétraitement. Y sont également renseignés, le nombre de reads générés ainsi que leur taille moyenne.

L’Ion Torrent™ Mini Lab sera à Lille
le 20 Juin 2013
14h00-16h00
Parking du Campus Institut Pasteur de Lille
1 rue du Pr. Calmette
59000 Lille
Métro Ligne 2, Station Grand Palais
Flyer : une mini, un Ion Torrent dedans…
 Ce qui suit pourrait être tiré d’un roman d’anticipation technofuturiste où le héros s’empêtre dans un polar aux implications géopolitiques. Après la mise en application de la politique de l’enfant unique, politique lancée en 1979 par Deng Xiaoping qui a eu la fâcheuse conséquence d’induire un déséquilibre entre les sexes (116 garçons pour 100 filles, 2005), la Chine, forte de sa population de presque 1 milliard 400 millions de ressortissants, cherche à appliquer les lois de la sélection génomique sur sa propre population. Le Q.I., phénotype relativement héritable (héritabilité supérieure à 0,5) est le deuxième caractère, après le sexe, « choisi » par les autorités. Cette article développe les différents éléments qui ont permis à la Chine de devenir le premier pays transhumaniste.
Ce qui suit pourrait être tiré d’un roman d’anticipation technofuturiste où le héros s’empêtre dans un polar aux implications géopolitiques. Après la mise en application de la politique de l’enfant unique, politique lancée en 1979 par Deng Xiaoping qui a eu la fâcheuse conséquence d’induire un déséquilibre entre les sexes (116 garçons pour 100 filles, 2005), la Chine, forte de sa population de presque 1 milliard 400 millions de ressortissants, cherche à appliquer les lois de la sélection génomique sur sa propre population. Le Q.I., phénotype relativement héritable (héritabilité supérieure à 0,5) est le deuxième caractère, après le sexe, « choisi » par les autorités. Cette article développe les différents éléments qui ont permis à la Chine de devenir le premier pays transhumaniste.
Acte 1 : une politique de reconquête
La Chine possède des ports en Europe, une partie significative de la dette américaine, des milliers d’hectares agricoles en Afrique et un parc de bombes atomiques qui ne laisserait pas insensible le docteur Folamour. Malgré un budget de millions de dollars alloué à une armée aux ordres du parti unique, l’usine du monde ne semble pas avoir de volonté expansionniste impérialiste en vue de repousser ses frontières (si on omet le sujet du Tibet). Il semble que les ambitions de la Chine actuelle résident moins dans le fait de mettre à la disposition de l’occident sa main-d’œuvre bon marché que de devenir la première puissance mondiale matérielle et intellectuelle. c’est pourquoi le pays mise toujours sur sa vraie matière première : l’humain. Devenue une puissance spatiale avec ses Taikonautes, la Chine envoie dans les meilleures universités du monde, ses étudiants qui, de retour (pour ceux qui reviennent), accomplissent une recherche de tout premier plan. Dans le champ de la génomique et des biotechnologies, la Chine s’impose aujourd’hui comme un acteur de tout premier plan.
Acte 2 : une recherche efficace
En effet, la Chine compte plus d’un cinquième de la population mondiale et compte aussi sur la volonté de fer pour retrouver son lustre d’antan. Il est alors, relativement aisé d’imaginer que même si une portion faible de sa population atteint les études supérieures, le pays sélectionne des étudiants qui deviendront des chercheurs motivés et ayant émergé des tous meilleurs étudiants d’une classe d’âge donné. Il est passé le temps où les meilleurs chercheurs chinois s’exilaient inconditionnellement aux Etats-Unis pour finir par être naturalisés – le classement académique des universités mondiales (aussi appelé classement de Shanghaï, ou encore « Academic Ranking of World Universities » en anglais) est un outil parfait pour identifier les meilleures universités où envoyer ses étudiants. La Chine devient même attractive sur la scène mondiale de la recherche scientifique et technologique. Selon un rapport de l’OCDE, le pays est deuxième pour la dépense en recherche et développement, injectant 136 milliards US$ en 2006 (USA : 330 milliards US$ pour la même période).
L’Empire du milieu oriente sa politique du développement technologique dans des domaines jugés comme hautement stratégiques: l’énergie, les ressources naturelles, l’environnement, les techniques de production industrielle et les technologies de l’information, les biotechnologies, les nanotechnologies, les technologies spatiales et maritimes. La Chine est en tête des pays en nombre de brevets déposés par année, ceci est l’aboutissement de trente ans durant lesquels le pays a misé sur la recherche scientifique pour rattraper son retard technologique sur l’Occident. Depuis le début des années 2000, le gouvernement promeut une innovation purement chinoise. Concernant le niveau des étudiants chinois, ceux-ci commencent à être l’objet de discriminations dans certaines universités américaines où ils occupent les meilleurs places. La recherche scientifique, basée sur une « exportation » massive d’étudiants a permis à la Chine de rattraper son retard technologique, cette technologie aujourd’hui sert une recherche scientifique de tout premier plan et sert, en ce qui concerne la biotechnologie, un dessein qui peut faire peur, vu d’ici.
Acte 3 : des moyens puissants
L’exemple qui vient à l’esprit immédiatement est le BGI (Beijing Genomics Institute). A ce sujet, vous pouvez lire notre précédent article : Taylor + Deng Xiao Ping + Watson = la Chine marche sur les chromosomes. Rappelons que le BGI est la plus grande plateforme génomique mondiale et un contributeur scientifique de tout premier ordre. Grâce à des plateformes de haute-technologie permettant de générer des volumes de séquences et de typages à de très bas coût, la Chine aurait la possibilité d’appliquer les résultats de sa recherche scientifique à l’échelle de sa vaste population.

Le BGI développe un laboratoire de génétique cognitive avec pour ambition de répondre à ces questions : Comment fonctionne le cerveau humain ? Comment les gènes affectent la capacité cognitive ? Comment les gènes et l’environnement interagissent pour produire l’intelligence et la personnalité humaine ? Le BGI a lancé un recrutement d’une cohorte de personnes volontaires aux Q.I. élevés. L’objectif est donc de rechercher les variations génétiques associées à des variations significatives au niveau du Q.I. (notons qu’une étude cas-témoin eut nécessité une cohorte de Q.I. bas à très bas… difficile de procéder à un recrutement volontaire !). Le Q.I. étant plutôt héritable, il sera donc techniquement possible de sélectionner les porteurs des variants « Q.I. élevé ». Ainsi il est possible de procéder à deux niveaux :
– orientation / optimisation « d’accouplements » : les porteurs de ces variations « Q.I. élevé », après s’être génotypés, auront tendance à rechercher un conjoint lui aussi porteur de ces mêmes variations afin de maximiser leur chance de procréer un enfant lui même porteur de ces variations (notons ici qu’il n’y a pas de réel progrès génétique, en tant que tel)
– passons par l’étape FIV : évitant de miser sur le hasard, les gamètes des deux futurs parents produiront plusieurs dizaines d’embryons, chaque œuf sera biopsié (une petite étape de phi29, un génotypage ciblé ou une sélection génomique sur la base de l’exploitation d’un génotypage pan-génomique à l’aide d’une cartographie basée sur plusieurs millions de marqueurs SNPs humains). Fort de la connaissance du « Q.I. estimé » de ces dizaines d’œufs… il ne restera qu’à « contrecarrer » l’aléa de méiose et de ne ré-implanter que ceux présentant le meilleur score de Q.I. estimé…
 La sélection humaine pose bien évidemment des problème de bioéthique. Si l’occident sélectionne d’ores et déjà des êtres humains dans le but de limiter les enfants nés porteurs de tares génétiques majeures, l’Empire du milieu semble parti sur une autre voie (même si cette voie n’empêche pas l’autre) : améliorer significativement le Q.I. de sa population et ainsi basculer dans l’ère de la transhumanisation (selon ce mouvement intellectuel, l’Homme doit prendre en main sa propre évolution).
La sélection humaine pose bien évidemment des problème de bioéthique. Si l’occident sélectionne d’ores et déjà des êtres humains dans le but de limiter les enfants nés porteurs de tares génétiques majeures, l’Empire du milieu semble parti sur une autre voie (même si cette voie n’empêche pas l’autre) : améliorer significativement le Q.I. de sa population et ainsi basculer dans l’ère de la transhumanisation (selon ce mouvement intellectuel, l’Homme doit prendre en main sa propre évolution).
Après le Q.I., le grand jeu du moment consiste à deviner quel sera le phénotype de choix (la docilité ?, le courage ?), il faut espérer que l’on attendra quelques décennies avant d’envisager le premier humain OGM… Envisageant cela, on caricature la démarche actuelle chinoise et on s’imagine, un peu, à la place de Charlton Heston à la fin de Soleil Vert (film de Richard Fleischer, sorti en 1973) quand il découvre la matière première constitutive de l’aliment qui nourrit une planète devenue stérile.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages