Lancé en avril 2012, Coursera est une entreprise qui propose un accès gratuit à un ensemble de cours en ligne. Rien de nouveau par rapport aux nombreux MOOC (pour « massive open online course ») de plus en plus présents sur la toile? Dans cet article nous égrènerons les principales caractéristiques de Coursera : ses atouts, en quoi il se démarque du cours en ligne classique et pourquoi il pourrait constituer une petite révolution dans le monde éducatif .
– La plupart des MOOC sont très centrés autour de l’informatique (on peut le comprendre), des sciences mathématiques et de l’ingénierie. Coursera propose un panel de cours très variés, allant de la médecine à la poésie en passant par l’économie et l’histoire, sans oublier la biologie et la bioinformatique. On trouve même des cours de guitare en ligne!
– Coursera a conclu un partenariat avec des dizaines d’universités (dont de très prestigieuses) à travers le monde, offrant ainsi des cours de haute qualité dans différentes langues: fin 2012 Coursera annonce 680 000 inscrits provenant de 42 pays.
– Pour les instructeurs, Coursera est un catalyseur de propagation du savoir, certains cours dépassant les 100 000 étudiants, beaucoup plus que les 400 étudiants qu’un professeur peut espérer instruire chaque année dans son université. Un professeur de l’université de Stanford a ainsi calculé que son cours Coursera de 100 000 étudiants revenait à distiller des cours à une classe pendant 250 ans!
– Les cours sont fixés dans le temps, renouvelés, interactifs avec l’insertion de questionnaires au milieu des présentations, des exercices à rendre et à corriger, des forums pour partager. L’apprentissage est clairement maximisé si on se donne la peine de suivre assidument les cours. Coursera s’appuie sur des principes pédagogiques simples :
- Les questionnaires et exercices permettent d’ancrer le savoir
- Le partage et les corrections des exercices par ses pairs donnent une vision différente sur son travail et la façon dont l’étudiant a lui-même assimilé (se mettre à la place du correcteur en quelque sorte)
- L’organisation en classes: la présence d’un forum permet de partager son expérience et ses conseils avec ses pairs , ce qui est plus motivant.
– Pour les étudiants /autodidactes, c’ est une aubaine : participer à des cours réservés d’habitude à une élite et tout cela gratuitement, vive le e-learning!
Pour l’instant le plan d’affaire semble un peu flou. Pour plus de détails vous pouvez consulter la page Wikipedia dédiée à Coursera. Des certificats de participation authentifiés sont déjà délivrés (de l’ordre de 30 à 60$ par cours) pour permettre à l’e-étudiant de valoriser les compétences acquises. Coursera pourrait aussi vendre votre profil à des entreprises de recrutement (avec l’accord des étudiants). A terme des frais de scolarité pourraient être également appliqués.
Les nouveaux MOOC tels que Coursera, Udacity (MOOC plutôt centré sur les disciplines scientifiques) ou edX ( association à but non-lucratif ) sont-ils en train de révolutionner notre façon d’apprendre? Il nous faudra attendre quelques années avant de mesurer l’impact de cette propagation à grande échelle de l’apprentissage. Va-t-on assister à une véritable explosion du nombre d’autodidactes aussi compétents que de nouveaux diplômés? Ou cette manne de savoir va-t-elle rester marginale et utilisée par une élite déjà formée par le système universitaire « classique » et avide de nouvelles connaissances?
Pour finir, voici les principaux cours liés aux biotechnologies actuellement sur Coursera :
A plus long terme, vous retrouverez également un cours de l’université de Melbourne sur l’épigénétique qui commencera au 1er juillet et un cours d’introduction à la bioinformatique par l’université de San Diego d’ici la fin d’année 2013.
La liste complète des cours autour des sciences de la vie est disponible à cette adresse : https://www.coursera.org/courses?orderby=upcoming&cats=biology
 Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Galaxy est en fait une brique d’une collection d’outils dédiés à l’analyse et au stockage de données biologiques : GMOD ( Generic Model Organism Database )
Le lundi 14 mai a été l’occasion pour nous d’assister à une conférence sur l’utilisation de certains des outils GMOD, dont voici les principaux enseignements :
> Le projet GMOD a pour objectif de fournir à l’utilisateur biologiste un ensemble d’outils interconnectés, libre de droit (open-source), générique (pour tous types de données biologiques) et facile d’utilisation (à travers des services Web principalement)
> Les outils GMOD sont développés (et donc installés) par des bioinformaticiens pour une utilisation par des biologistes.
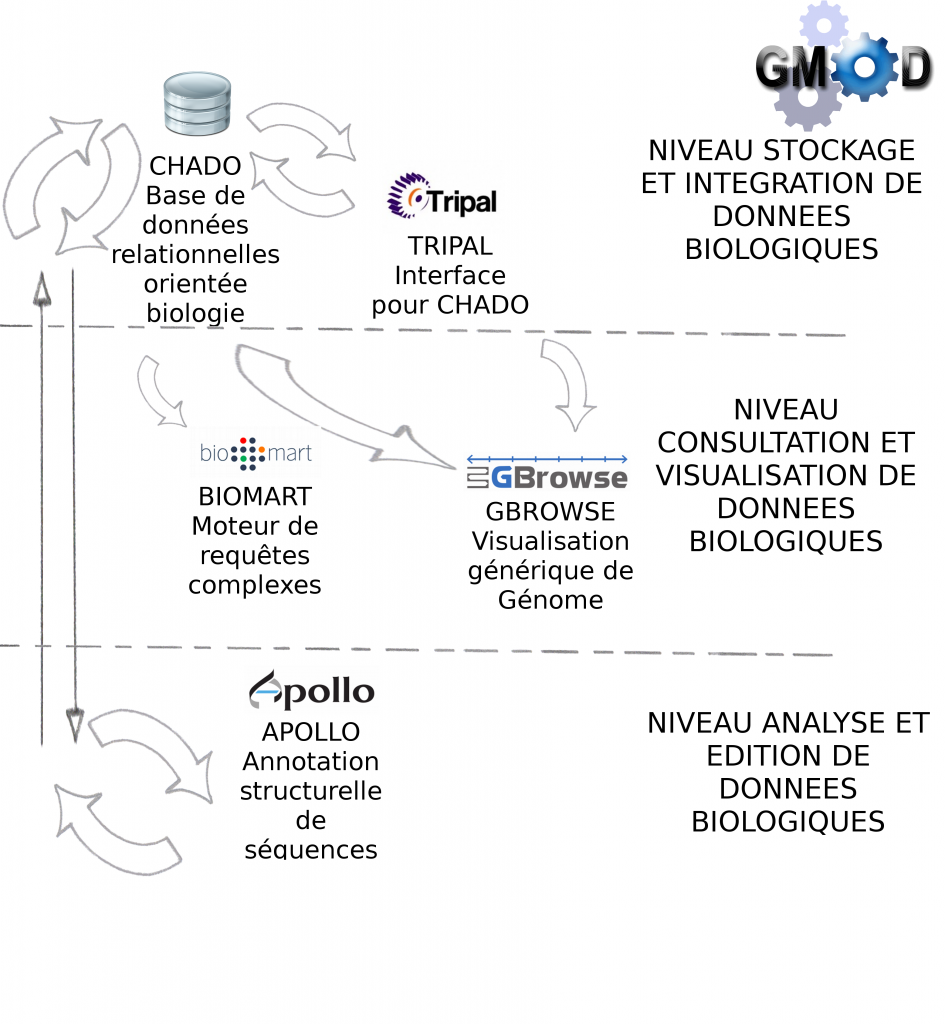
> Certains outils sont indispensables à GMOD, pour la manipulation de données génomiques, c’est le cas des outils Chado et Gbrowse qui sont respectivement les squelettes pour la manipulation et pour la visualisation des données biologiques.
Le schéma ci-dessous décrit les modules et interactions présentés lors de cette journée thématique :
> GBrowse est un outil de visualisation de données biologiques très puissant et certainement l’outil le plus populaire de la suite GMOD
Description détaillée : GMOD-GBROWSE
> Biomart est un outil de recherches avancées (ou requêtes complexes) pour la base de donnée relationnelle Chado
> Apollo est un module pour la correction manuelle d’annotation structurelle
Description détaillée : GMOD-APOLLO
> Tripal est une interface web développée en PHP pour interrogation de Chado
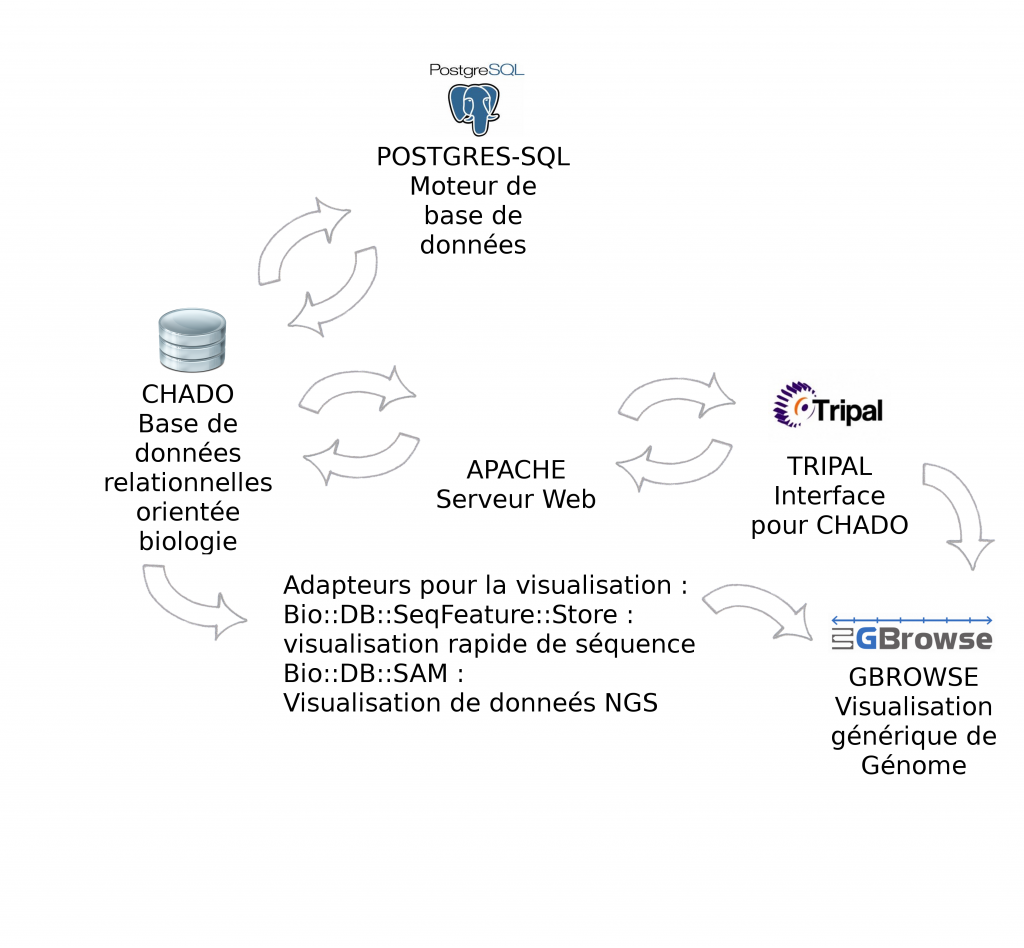
Environnement informatique : CHADO fonctionne par défaut avec Postgres-Sql. Pour l'interfaçage avec Tripal, la solution étant développée avec PHP, il est nécessaire d'installer un serveur Web Apache. Gbrowse peut s'utiliser à partir de l’interface Tripal mais également en "stand-alone". Afin d'accélérer la visualisation des données il est vivement recommandé d'utiliser les adapteurs Bio::DB::*, soit en relais entre Chado et Gbrowse (présentés dans cet article), soit en dupliquant les informations dans les deux bases de données (la visualisation ne se connecte qu'à Bio::DB sans passer par CHADO). Intuitivement, nous privilégions la première solution qui n’entraîne pas de duplication.
Pour conclure, GMOD propose un ensemble de modules pour la standardisation des processus bioinformatique : stockage et manipulation de données biologiques, visualisation et analyses avancées (assemblage, annotation…).
L’utilisation de tels outils (open-source) va dans le bon sens pour le partage scientifique et la standardisation des processus utilisés lors de l’analyse bioinformatique. Cela n’était pas le but de la conférence à laquelle nous avons assistée mais il serait également intéressant de connaître les conditions pour intégrer ces propres outils bioinformatiques en tant que brique GMOD.
GMOD est donc intéressant si :
> vous souhaitez stocker et gérer vos données biologiques
> vous cherchez des solutions d’analyses bioinformatiques déjà développées et robustes
Les biologistes sont les utilisateurs des outils GMOD, en revanche l’installation, l’administration et la formation des utilisateurs ne peuvent échapper à l’intervention, au moins ponctuelle, d’un bioinformaticien.
Si vous projetez d’utiliser ces outils, nous vous conseillons donc dans un premier temps, de regrouper l’ensemble des acteurs, installateurs comme utilisateurs de GMOD, afin de présenter les solutions offertes par l’outil et déterminer les besoins et objectifs pour votre propre utilisation.
Le réseau régional d’ingénieurs en bioinformatique de Lille et le PPF bioinformatique organisent lundi 14 Mai 2012 une conférence sur l’utilisation des outils GMOD (Generic Model Organism Database). Cette conférence se déroulera de 13h30 à 17h30 dans l’amphithéâtre de l’Institut de Biologie de Lille.
« Le projet GMOD a pour objectif de fournir une série d’outils génériques, clé en main, pour gérer et visualiser différents types de données biologiques. »
Deux interventions sont prévues :
Olivier Arnaiz du CGM Centre de Génétique Moléculaire (UPR 3404 Gif-sur-Yvette) introduira GMOD tout en se focalisant plus particulierement sur deux outils : Chado et Gbrowse. Chado est un schéma de base de données relationnelles qui permet d’intégrer des données « omiques » (génomiques, transcriptomices, séquençages…), tandis que GBrowse est un outil permettant de visualiser des annotations sur un génome.
Joelle Amselem de l’INRA – URGI (BIOGER Versailles) présentera un outil d’annotation développé à l’URGI à partir de modules GMOD. Deux exemples concrets d’utilisation seront développés.
Pour en savoir plus, vous pouvez télécharger le programme ici.
Si vous souhaitez des informations sur GMOD, je vous invite à consulter la présentation de GMOD.
La confusion entre mate-pair et paired-end, tant au niveau technologique (selon qu’on lise les notes techniques d’Illumina, de Roche ou de Life) que logiciel nous a mené à rédiger, en collaboration avec Ségolène Caboche, Bioinformaticienne à l’université de Lille2, une note technique dont le contenu est résumé ci-dessous :
– Genèse de la confusion entre mate-pair et paired-end
– Descriptions les deux approches, avec un focus sur les principales technologies de seconde génération de séquenceurs
– Traitement au niveau logiciel et conseils généralistes pour l’utilisation
Le document est consultable dans son intégralité sur notre blog :
Télécharger Paired-end versus mate-pair
Bonne lecture!
Il y a quelques semaines, nous avions discuté de l’utilisation de logiciel de Workflow pour la bioinformatique. Il est temps de passer à la pratique en vous présentant un de ses dignes représentants : Galaxy.
Le workflow Galaxy fournit un ensemble d’outils pour la manipulation et l’analyse de données génomiques. Il est très intuitif dans l’utilisation ce qui en fait une cible de choix pour le biologiste.
Il est possible d’utiliser Galaxy directement depuis le serveur. Avantage conséquent pour les bioinformaticiens il est possible d‘installer sa propre instance de serveur Galaxy, cette option fera l’objet d’un prochain post technique.
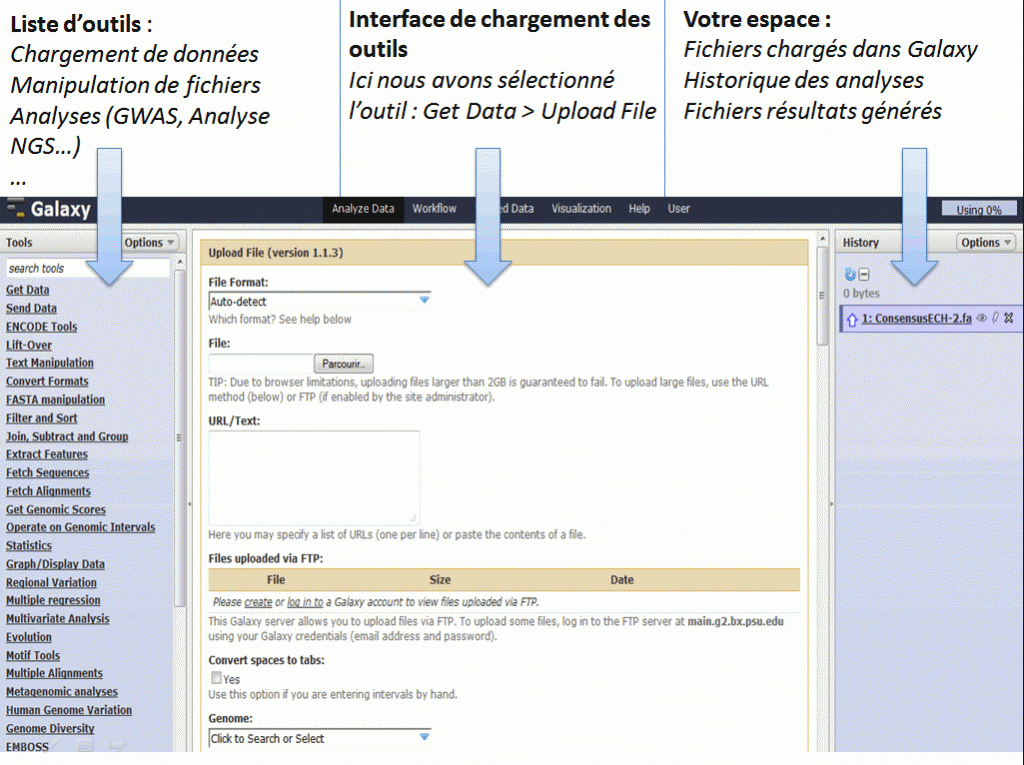
Du point de vue de l’interface graphique :
On peut également créer des workflows, les enregistrer dans un espace dédié, les partager, et les exécuter de façon automatique.
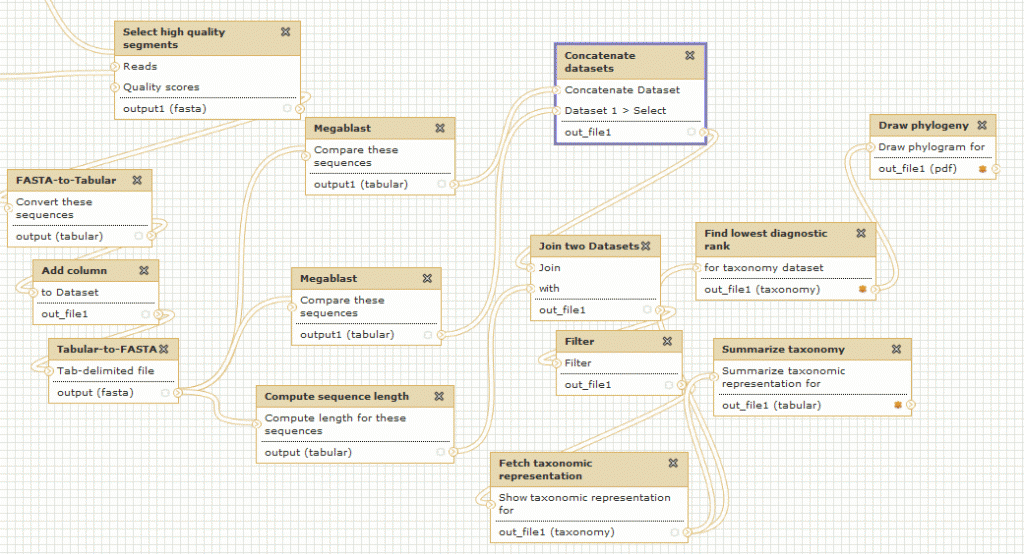
Pour exemple ce workflow de métagenomique publié gratuitement par un utilisateur de Galaxy (vous devez être connecté pour visualiser le workflow dans Galaxy)
Les outils dédiés analyse de données NGS sont régulièrement mis à jour et nul doute que d’ici peu, certains seront dédiés IonTorrent.
Les tutoriels sont également très bien faits, on apprend très vite à maitriser l’environnement grâce à des dizaines de vidéos d’aides.
Galaxy offre donc la possibilité d’exécuter des analyses bioinformatiques sans effort de programmation. La version en ligne est intéressante car elle permet de se familiariser aux logiciels et d’exécuter l’analyse depuis un portable, mais la possibilité d’intégrer ces propres outils (nous y reviendrons) est indéniablement un gros avantage de la version locale.
Si nous devions citer un inconvénient, plutôt d’actualité : l’utilisateur est obligé de charger ses données en mémoire dans Galaxy, le temps de chargement peut être très long si l’on manipule des données issues d’expériences NGS. D’autres workflows tels que Ergatis, fonctionnent en local et permettent à l’utilisateur d’utiliser directement les données présentent sur l’ordinateur.
Pour en savoir plus :
La description complète du logiciel Galaxy en Français sur PLUME :
http://www.projet-plume.org/fiche/galaxy
Le Galaxy Wiki :
http://wiki.g2.bx.psu.edu/FrontPage
La publication associée :
Constat : le séquençage, un générateur fantastique de données
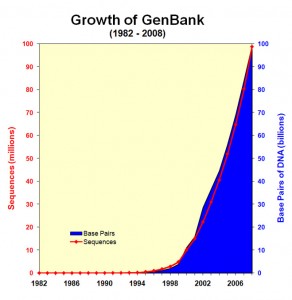
L’explosion de la génération de données génomiques (Figure 1.0) et l’hétérogénéité de ces données entraînent inévitablement un accroissement de l’écart entre les données, les connaissances et l’information que l’on peut en extraire (Figure 1.1).
Le développement d’outils bioinformatiques permet de répondre à l’analyse de données dans l’ensemble des domaines de la biologie, mais le besoin en formalisation (des formats, des processus, des architectures matérielles…) est primordial si l’on veut éviter les problèmes d’incompatibilité et de réutilisation des solutions.
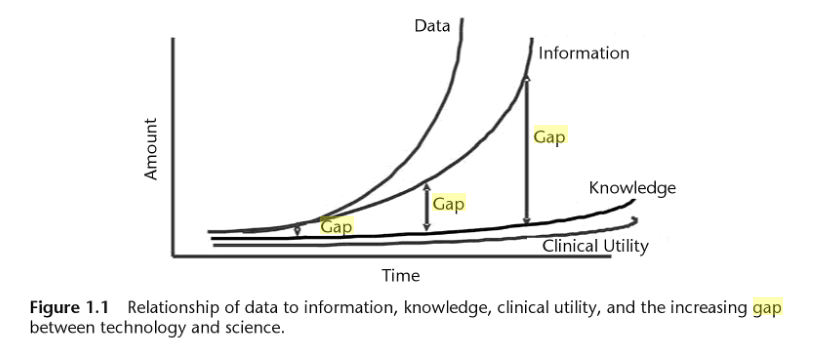
Figure 1.1 : Issu de "Biomedical informatics in translational research" Par Hai Hu,Richard J. Mural,Michael N. Liebman
En conséquence, afin d’obtenir des informations interprétables biologiquement à partir de ces données, la mise en place de processus d’analyse va de pair avec l’utilisation de logiciels à jour, efficaces pour la gestion de données à grande échelle.
En outre, les logiciels doivent aussi simplifier cette gestion de données et fournir des outils d’analyses bioinformatiques accessibles mais aussi reproductibles pour tous types d’utilisateurs – dont les biologistes eux-mêmes. C’est dans ce cadre que s’insèrent l’automatisation de processus (ou « Workflow ») et le développement de « Workflow management system », ou logiciel de Workflow, pour la bioinformatique.
Workflow ou automatisation de processus : définition
Le logiciel de Workflow s’inscrit aussi dans une logique de pérennisation des processus analytiques, qui a pour but de sortir de la logique « projet » (dans le sens ponctuel) en créant des processus d’analyses génériques.
Un logiciel de Workflow est un outil permettant d’exécuter un ensemble de processus de façon automatique. Ces « pipelines » sont très présents en bioinformatique (à défaut d’être tres utilisés) car ils permettent aux chercheurs en biologie d’analyser leurs données (issues de séquencages, génotypages) de façon relativement transparente et (quasiment) sans l’aide d’informaticiens (denrées rares dans la recherche).
Toutefois, il convient de distinguer deux sortes de logiciel de Workflow :
– Les logiciels de Workflow qui permettent aux chercheurs de manipuler leurs données et exécuter leurs analyses sans posséder de connaissances en écriture de scripts ou en bases de données. Les données sont rapatriées au sein du logiciel de Workflow, permettant l’exécution d’un ensemble de tâches, à travers des modules pré-installés. En séquençage, le Workflow permet de convertir des séquences en formats divers, les filtrer ou les assembler… Le logiciel de Workflow ISYS (2001), BioMOBY, Taverna et plus particulièrement Galaxy (qui fera l’objet d’un prochain post ici) entrent dans cette catégorie.
– Les logiciels de Workflow qui assurent un accès direct à des composants (installés sur le serveur) et/ou aux données génomiques sans passer par un rapatriement préalable des données. WildFire, Pegasys ou Ergatis (ce dernier sera décrit dans un prochain post) font partie de cette catégorie. De manière générale ces logiciels de Workflow sont plus difficiles à prendre en main mais sont évidemment plus flexibles.
Pour résumer, quel que soit le logiciel de Workflow utilisé il permet :
– D’automatiser des processus d’analyse (idéalement répétitifs) en les reliant dans un pipeline
– De lancer des analyses sur des architectures matérielles complexes telles des grilles de calculs (voir l’article sur Grisbi) ou des serveurs
– De formaliser le processus d’analyse en vue d’une publication scientifique
Philosophie générale : interaction et exemple de workflow
Pour finir cette introduction, nous vous proposons deux schémas : la place du logiciel Workflow et un exemple concret.
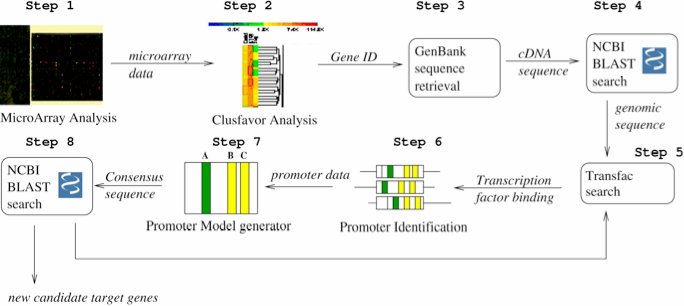
- Figure 1.3 : Exemple de conception d’un Workflow d’identification de promoteurs (extrait de « Accelerating the scientific exploration process with scientific workflows« )
 pour but, à travers la mutualisation de ressources informatiques des acteurs du projet, de proposer l’utilisation d’applications bioinformatiques déployées sur grille de calcul au plus grand nombre.
pour but, à travers la mutualisation de ressources informatiques des acteurs du projet, de proposer l’utilisation d’applications bioinformatiques déployées sur grille de calcul au plus grand nombre.
Une revue intéressante et qui se veut exhaustive sur les conséquences de la généralisation des technologies de séquençage et les solutions/adaptations possibles, on y retrouve pèle-mêle :
– Un listing à jour (2011) des différentes plateformes dédiées à la génération de données de séquençage (Illumina, Roche, Life Technologie pour ne citer qu’eux…) et leurs spécificités;
– La description de quelques stratégies de NGS : identification de variants, séquençage d’éxome, séquençage sur des régions précises…
– Les problématiques en bioinformatiques : stockage et analyse de données, développement de solutions logicielles adaptées…
– Les différentes analyses ainsi que des listes de logiciels pour répondre aux besoins: assemblage denovo et sur génome de référence, annotation et prédiction fonctionnelle, autant open-source que sous licence payante.
Il convient tout d’abord de distinguer deux types de bioinformaticiens :
-Ceux qui sont intégrés dans une unité de recherche : ils sont souvent spécialisés sur une thématique ou même un projet précis : généralement isolés, non permanent, impliqués et informés des problématiques biologiques
-Ceux qui font partie d’une unité de bioinformatique : en émulation avec d’autres bioinformaticiens, moins précaires, plus loin des problématiques biologiques
Ce document se veut une libre opinion sur la bonne intégration d’un, ou d’une équipe, de bioinformaticiens au sein d’un groupe de recherche et les clés pour la bonne réussite de celle-ci. Il est en grande partie inspiré de l’éditorial « On the organization of bioinformatics core services in biology-based research institutes » agrémenté de commentaires plus personnels.
Pour le bien-être interne et externe d’une plateforme de bioinformatique, il convient de :
-Bien séparer les tâches dîtes de prestation et les activités de recherches.
-Scinder les sujets traités par tâches bien définies : par exemple, support en développement et maintenance de base de données, analyse statistique de données haut-débit, support pour le déploiement d’applications sur environnement HPC, analyse de donnée NGS…
-Mettre en place un comité d’utilisateurs incluant des utilisateurs biologistes/bioanalystes chargés d’établir les priorités
-Mettre en place des outils pour suivre l’activité bionformatique de la façon la plus transparente possible
-Mettre en place au maximum des projets transversaux pour favoriser l’interface entre biologiste et bioinformaticiens, les interactions pluridisciplinaires sont les plus enrichissantes et souvent les plus productives. Comme toutes disciplines, la bioinformatique peut devenir endogame, particulièrement dans ou le cadre où l’on parle de bioinformatique comme une science à part entière, et non comme un outil au service de problématiques biologiques : une équipe de bioinformatique peut très bien travailler sur des sujets « porteurs » (donc plus facile à financer) sans interagir, ou seulement sporadiquement, avec des biologistes pour valider le bien-fondé du travail. C’est une des dérives du système d’évaluation actuel, pour plus d’information, lire la libre opinion publiée dans ce blog : Chemins de traverses : la recherche d’excellence ou quand les mots perdent leur sens.
-Mettre en place des formations pour les biologistes : collaborer avec des personnes connaissant un minimum les problématiques bioinformatiques est plus efficace
-Dans le même esprit, nommer un bioinformaticien responsable pour le support utilisateurs pour assister les biologistes dans leurs tâches quotidiennes de bioanalyse est très utile pour entretenir de bonne relation avec les laboratoires de recherche
-Pour les bioinformaticiens intégrés dans une unité, il est primordial de trouver des structures permettant de rencontrer et de partager avec d’autres bioinformaticiens, à défaut, passer quelques heures par semaine à effectuer de la veille technologique peut être un palliatif.
Pour conclure, une équipe de bioinformaticiens doit communiquer un maximum avec leurs partenaires biologistes afin de favoriser une saine interaction, un bioinformaticien, seul, entourer de biologistes se doit de rester à la page, idéalement en partageant et en communiquant sur ces projets à d’autres bioinformaticiens.
L’accroissement des capacités technologiques en biologie moléculaire à pour conséquence la possibilité de séquencer/génotyper mais aussi stocker toujours plus de données constituant une manne d’informations sans limite pour qui sait en extraire du sens. C’est le travail du bioinformaticien de proposer à la fois un support technique et analytique au biologiste en même temps que la capacité en terme de ressources informatiques pour répondre aux besoins.
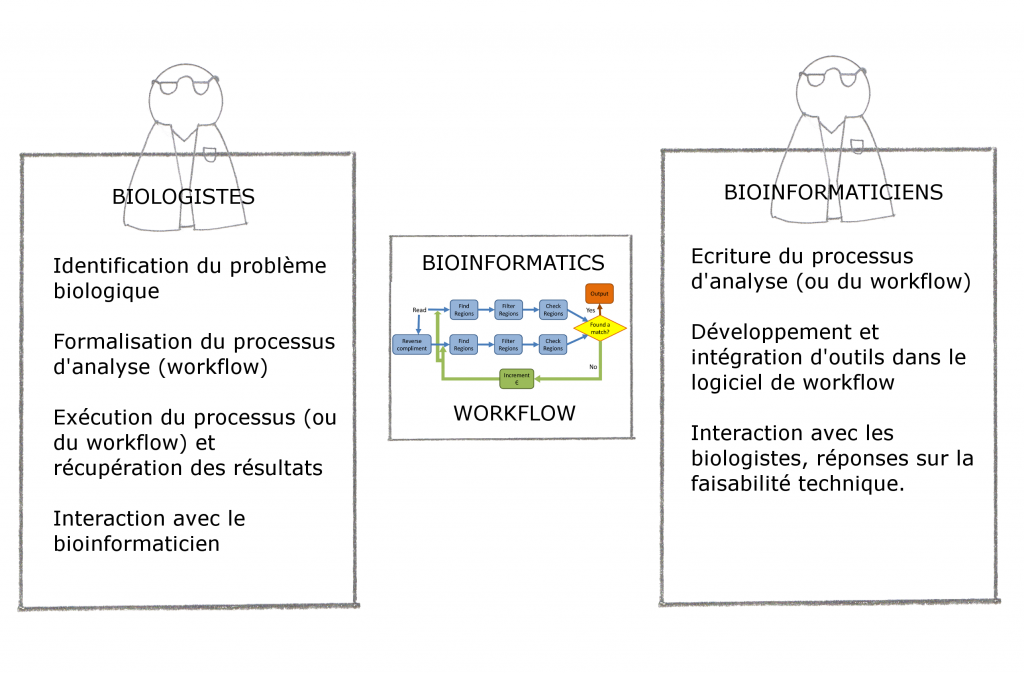
Ce premier post sur la place du bioinformaticien se focalise sur l’interaction avec le biologiste au sein d’un projet.
On peut dissocier un projet en trois phases :
– La préparation : identifier la question biologique, étudier la faisabilité et les coûts, mettre en place la méthodologie…
– La réalisation : génération des données biologiques et analyse bioinformatique
– La finalisation : interprétation des résultats, mise en production ou introduction d’une nouvelle question biologique
Le schéma ci-dessous montre un cheminement possible de l’intervention bioinformatique :
L’intervention du bioinformaticien est ici minimisée : il n’intervient qu’à partir de la phase analytique et dans l’interprétation des résultats (pour aider le biologiste à critiquer les résultats générés). Ceci pose plusieurs problèmes : les outils bioinformatiques doivent s’adapter, à tout prix, à la méthodologie mise en place précédemment. Si on constate que les méthodes d’analyses doivent être modifiées une fois la génération de données effectuées, la bioinformatique peut devenir ce qu’on appelle communément un goulot d’étranglement dans le projet…
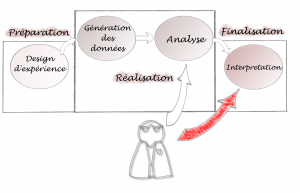
Au contraire, Le schéma ci-dessous nous parait plus judicieux :
Ici, l’intervention bioinformatique est optimisée : le bioinformaticien est totalement impliqué dans la préparation du projet. Et, au contraire, en fin de projet, les interventions sont minimisées car la méthodologie mise en place est adaptée pour une bonne compréhension par le biologiste. L’idéal et de fournir les outils nécessaires pour que le biologistes puisse lui-même exécuter ses analyses.
Pour résumer, l’ interaction doit se faire en amont et en aval de l’analyse et l’on se doit de minimiser les interventions pendant la phase d’analyse. En aval pour conseiller sur les méthodologies à utiliser, en amont pour contrôler les erreurs éventuelles rencontrées pendant la phase d’analyse (exécuter par le biologiste).
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages