Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Galaxy est en fait une brique d’une collection d’outils dédiés à l’analyse et au stockage de données biologiques : GMOD ( Generic Model Organism Database )
Le lundi 14 mai a été l’occasion pour nous d’assister à une conférence sur l’utilisation de certains des outils GMOD, dont voici les principaux enseignements :
> Le projet GMOD a pour objectif de fournir à l’utilisateur biologiste un ensemble d’outils interconnectés, libre de droit (open-source), générique (pour tous types de données biologiques) et facile d’utilisation (à travers des services Web principalement)
> Les outils GMOD sont développés (et donc installés) par des bioinformaticiens pour une utilisation par des biologistes.
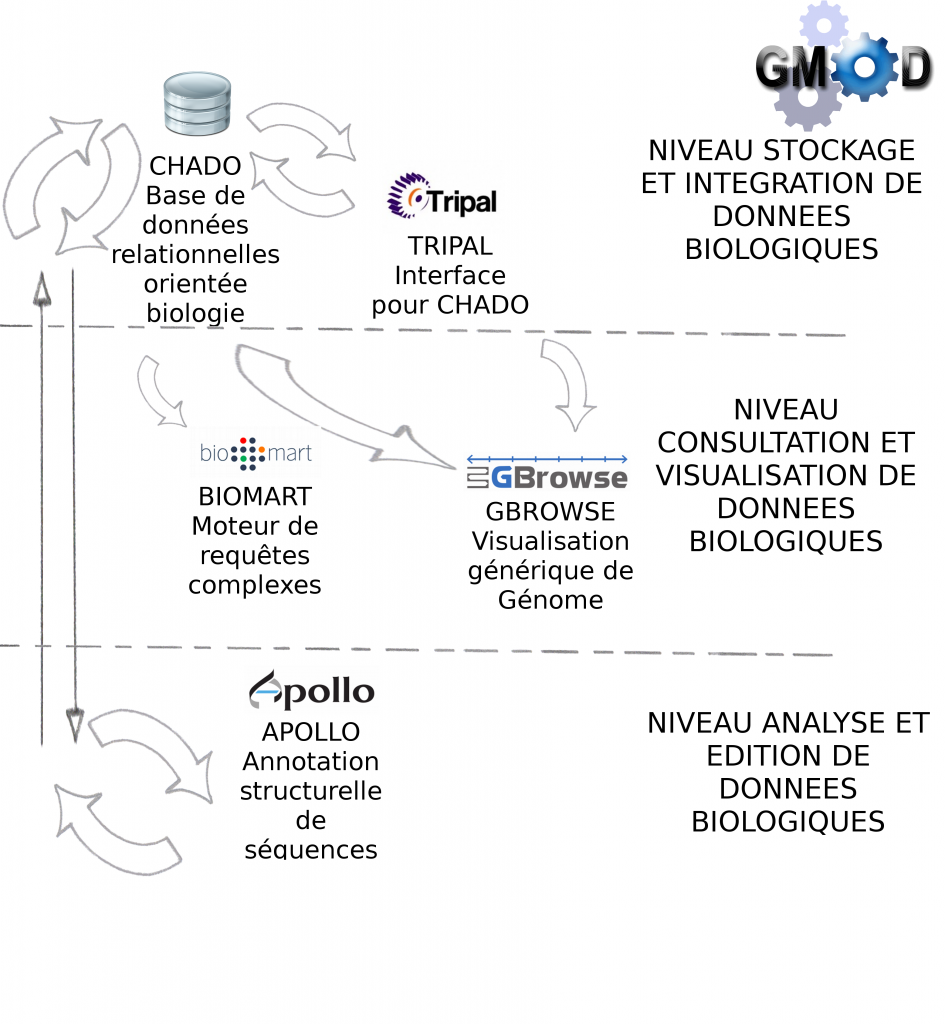
> Certains outils sont indispensables à GMOD, pour la manipulation de données génomiques, c’est le cas des outils Chado et Gbrowse qui sont respectivement les squelettes pour la manipulation et pour la visualisation des données biologiques.
Le schéma ci-dessous décrit les modules et interactions présentés lors de cette journée thématique :
> GBrowse est un outil de visualisation de données biologiques très puissant et certainement l’outil le plus populaire de la suite GMOD
Description détaillée : GMOD-GBROWSE
> Biomart est un outil de recherches avancées (ou requêtes complexes) pour la base de donnée relationnelle Chado
> Apollo est un module pour la correction manuelle d’annotation structurelle
Description détaillée : GMOD-APOLLO
> Tripal est une interface web développée en PHP pour interrogation de Chado
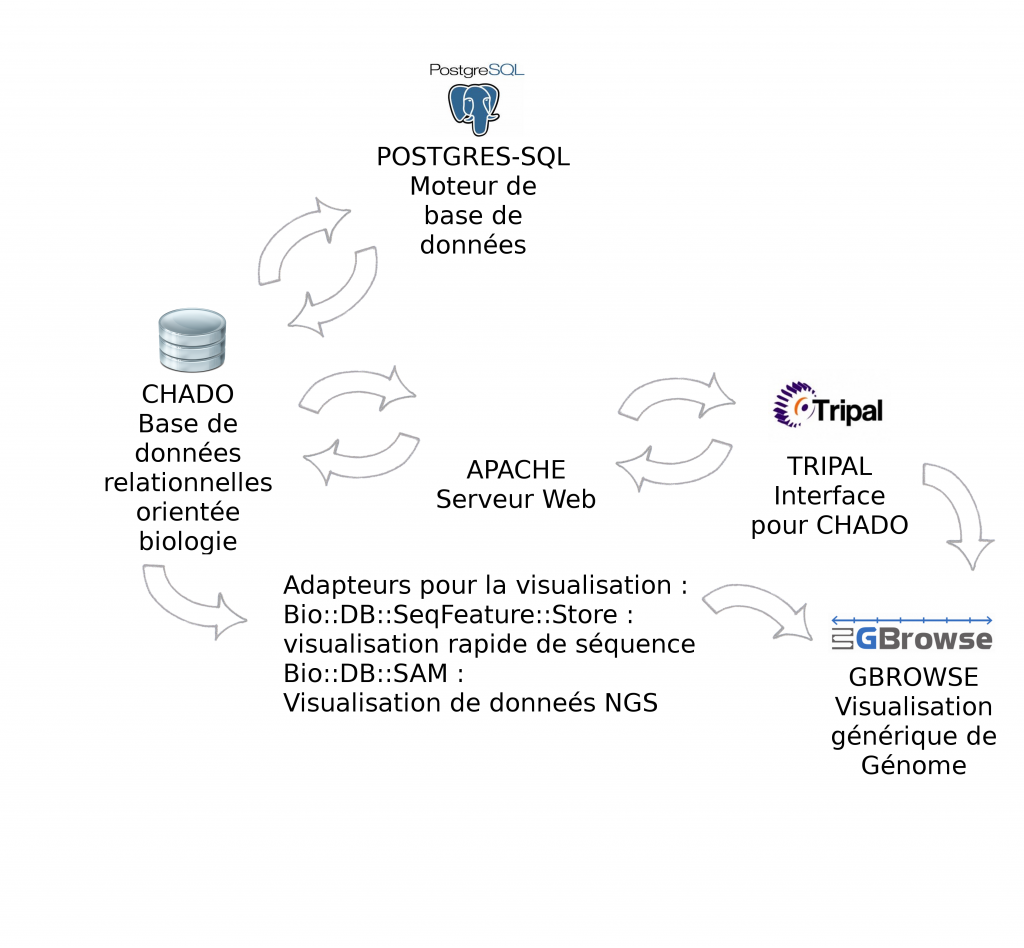
Environnement informatique : CHADO fonctionne par défaut avec Postgres-Sql. Pour l'interfaçage avec Tripal, la solution étant développée avec PHP, il est nécessaire d'installer un serveur Web Apache. Gbrowse peut s'utiliser à partir de l’interface Tripal mais également en "stand-alone". Afin d'accélérer la visualisation des données il est vivement recommandé d'utiliser les adapteurs Bio::DB::*, soit en relais entre Chado et Gbrowse (présentés dans cet article), soit en dupliquant les informations dans les deux bases de données (la visualisation ne se connecte qu'à Bio::DB sans passer par CHADO). Intuitivement, nous privilégions la première solution qui n’entraîne pas de duplication.
Pour conclure, GMOD propose un ensemble de modules pour la standardisation des processus bioinformatique : stockage et manipulation de données biologiques, visualisation et analyses avancées (assemblage, annotation…).
L’utilisation de tels outils (open-source) va dans le bon sens pour le partage scientifique et la standardisation des processus utilisés lors de l’analyse bioinformatique. Cela n’était pas le but de la conférence à laquelle nous avons assistée mais il serait également intéressant de connaître les conditions pour intégrer ces propres outils bioinformatiques en tant que brique GMOD.
GMOD est donc intéressant si :
> vous souhaitez stocker et gérer vos données biologiques
> vous cherchez des solutions d’analyses bioinformatiques déjà développées et robustes
Les biologistes sont les utilisateurs des outils GMOD, en revanche l’installation, l’administration et la formation des utilisateurs ne peuvent échapper à l’intervention, au moins ponctuelle, d’un bioinformaticien.
Si vous projetez d’utiliser ces outils, nous vous conseillons donc dans un premier temps, de regrouper l’ensemble des acteurs, installateurs comme utilisateurs de GMOD, afin de présenter les solutions offertes par l’outil et déterminer les besoins et objectifs pour votre propre utilisation.
Le réseau régional d’ingénieurs en bioinformatique de Lille et le PPF bioinformatique organisent lundi 14 Mai 2012 une conférence sur l’utilisation des outils GMOD (Generic Model Organism Database). Cette conférence se déroulera de 13h30 à 17h30 dans l’amphithéâtre de l’Institut de Biologie de Lille.
« Le projet GMOD a pour objectif de fournir une série d’outils génériques, clé en main, pour gérer et visualiser différents types de données biologiques. »
Deux interventions sont prévues :
Olivier Arnaiz du CGM Centre de Génétique Moléculaire (UPR 3404 Gif-sur-Yvette) introduira GMOD tout en se focalisant plus particulierement sur deux outils : Chado et Gbrowse. Chado est un schéma de base de données relationnelles qui permet d’intégrer des données « omiques » (génomiques, transcriptomices, séquençages…), tandis que GBrowse est un outil permettant de visualiser des annotations sur un génome.
Joelle Amselem de l’INRA – URGI (BIOGER Versailles) présentera un outil d’annotation développé à l’URGI à partir de modules GMOD. Deux exemples concrets d’utilisation seront développés.
Pour en savoir plus, vous pouvez télécharger le programme ici.
Si vous souhaitez des informations sur GMOD, je vous invite à consulter la présentation de GMOD.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages