Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?
Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?

En juillet 2017, Stockholm accueillera la 3ème conférence annuelle « Metagenomics and Metadesign of Subways and Urban Biomes (MetaSUB)« , qui rassemblera des chercheurs dédiés à la cartographie du métagénome urbain de plus de 67 villes à travers le monde (dont Paris et Marseille). Ce projet ambitieux a débuté il y a deux ans, alors que le professeur Christopher Mason et son équipe ont réalisé la première étude sur la microflore de surface, dressant le microbiome de la ville de New York. Avec ces données moléculaires essentiellement basées sur le séquençage haut-débit 16S, le projet a étudié la façon dont ce «microbiome urbain» change avec des variables telles que la météo, la propreté, les matériaux de construction et même les niveaux socio-économiques du quartier. L’équipe a cherché à établir des profils de microbiotes dans le métro, à identifier les bio-menaces potentielles et à fournir des données complémentaires qui peut être utilisé par la ville pour créer une «ville intelligente», c’est-à-dire qui agrège des données hétérogènes pour améliorer son urbanisme.
Près de la moitié (48%) de l’ADN séquencé ne correspondait pas aux organismes connus, soulignant qu’un microbiota incognita entoure les usagers du métro. Le projet du métro de New York n’était que le début, ce qui a conduit à la création d’un consortium international de laboratoires pour établir une « cartographie » mondiale de microbiomes dans les systèmes de transport en commun notamment. En cliquant sur la capture d’écran ci-dessous, on pourra se faire une idée de la communication « Metasubienne » qui n’a rien à envier à celle de Tara Oceans ou de MetaHit (qui sont toutes trois, d’excellentes communications autour d’un projet et d’un consortium).
Voici en quelques mots les promesses annoncées dans l’article Geospatial Resolution of Human and Bacterial Diversity with City-Scale Metagenomics (Cell Syst, 2016) :
« La région métropolitaine de New York City (NYC) est un endroit idéal pour entreprendre une étude métagénomique à grande échelle car c’est la plus grande et la plus dense des États-Unis; 8,2 millions de personnes vivent sur une masse continentale de seulement 755 km2. En outre, le métro de New York est le plus grand système de transport en commun dans le monde (par le nombre de stations), qui s’étend sur plus de 406 km et utilisé par 1,7 milliard de personnes par an. Ce vaste écosystème urbain est une ressource précieuse qui nécessite un suivi pour le maintenir et le sécuriser contre les actes de bioterrorisme, les perturbations environnementales ou les épidémies. Ainsi, nous avons cherché à caractériser le métagénome de NYC en examinant le matériel génétique des microorganismes et d’autres ADN présents dans, autour et au-dessous de New York, en mettant l’accent sur les métros et les zones publiques très empruntés. Nous envisageons cela comme une première étape vers l’identification des menaces biologiques potentielles, la protection de la santé des New-Yorkais et la mise à disposition de données moléculaires qui pourront être utilisée par la ville pour créer une «ville intelligente», c’est-à-dire celle qui utilise des données de grande dimension pour améliorer l’urbanisme, la gestion de l’environnement bâti, des transports en commun et de la santé humaine. »
La plate-forme de renseignement de marché de CB Insights analyse des millions de données sur le capital-risque, les start-ups, les brevets, les partenariats pour détecter les opportunités de demain qui dès lors qu’elles sont détectées deviennent celles d’hier. Triste devenir des données hautement périssables !
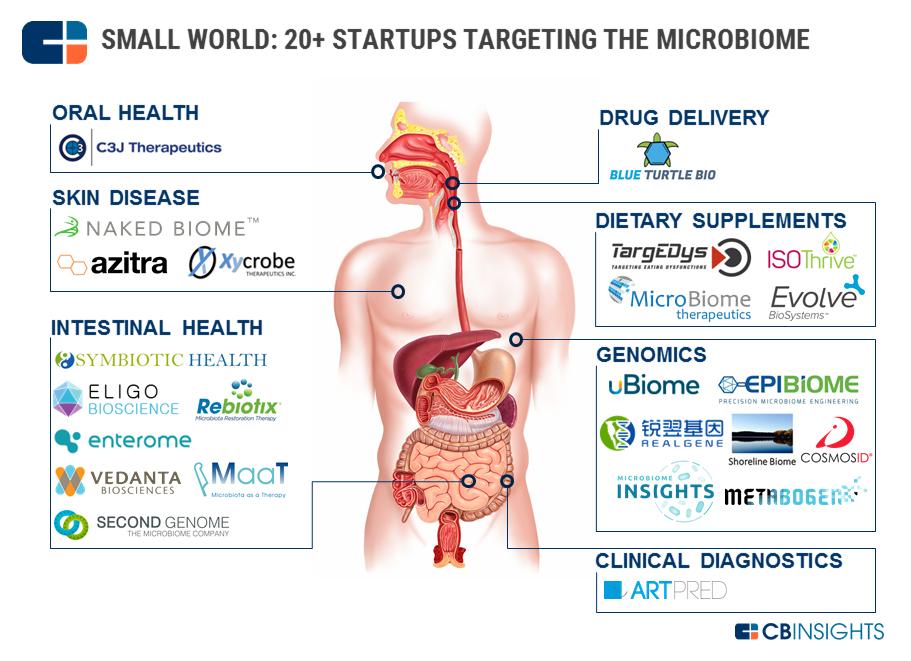
Dans son radar du jour… les start-ups s’attaquant au microbiote à des fins diagnostiques, thérapeutiques, pro- ou pré-biotiques… Beaucoup de ces sociétés telles que Blue Turtle Bio ou Microbiome therapeutics par exemple ont l’ambition de modifier « l’organe » le plus facilement (cela reste à démontrer) malléable de notre corps : le microbiome… Ces entreprises utilisent le séquençage et la métagénomique à haut-débit pour tenter de valider leurs allégations. Il y a fort à parier que nous sommes en train d’assister à une bulle qui continuera de grossir encore puisqu’en matière de métagénomique un grand nombre de projets liés à l’étude des sols vont arriver à maturité d’ici peu… (terroirs pour les vins, microbiotes sols pollués etc.).
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages