L’évolution des technologies de biologie moléculaire combinée à des systèmes de détection toujours plus sensibles, des techniques d’amplification accessibles dans des systèmes intégrés ont rendu possible l’analyse du transcriptome d’une cellule isolée. Dans de nombreux domaines tels que la cancérologie, la biologie des cellules souches, l’ingénierie des tissus, le signal moyen peut cacher la pertinence du signal noyé dans la masse des cellules d’intérêt.
Défier la loi de la moyenne, en tenant compte de l’hétérogénéité cellulaire pour tenter de capter ce signal qui échappe aux systèmes n’analysant que le mélange de cellules, est le credo du système proposé par Fluidigm : le C1™ Single-Cell Auto Prep System.
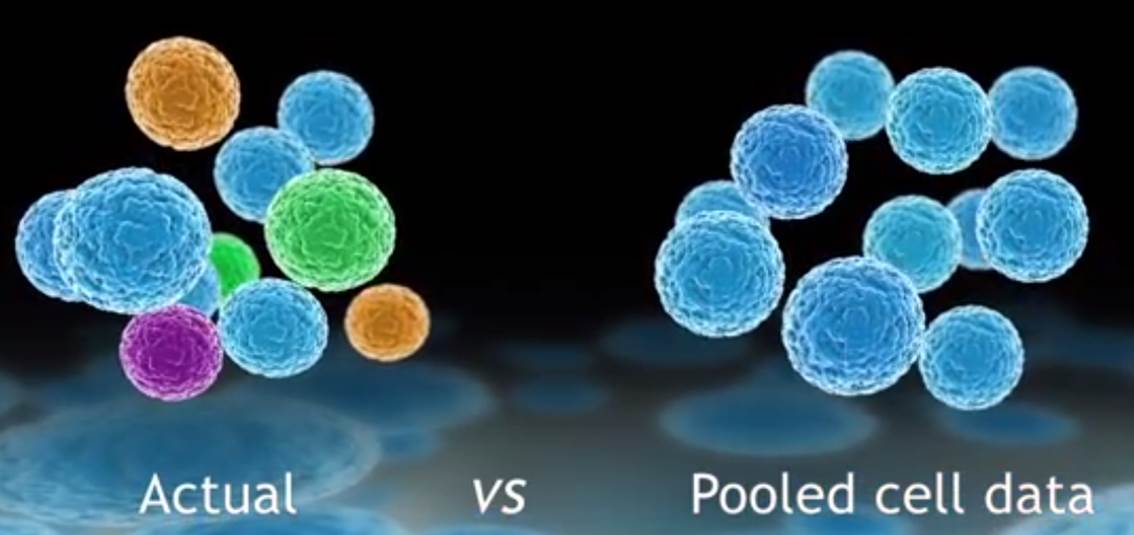
Isoler des cellules pour extraire leur ARN peut être une tâche technique ardue consommatrice de temps et très sensible aux contaminations.
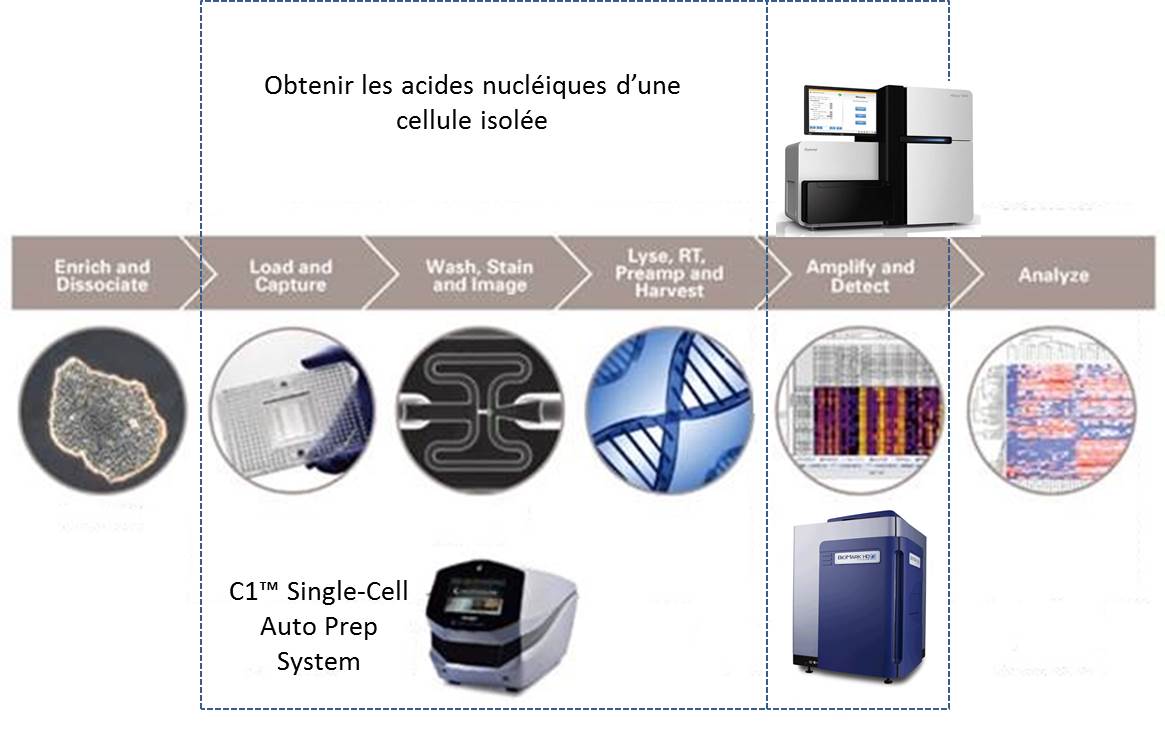
La société américaine, Fluidigm propose donc un système plutôt simple permettant de capturer quelques cellules pour en extraire leurs acides nucléiques. Le consommable est composé d’une plaque microfluidique comportant 96 pièges à cellules enchaînés. La multiplicité de ces pièges permet d’accroître les chances de capturer une cellule dans l’état escompté. Une fois la phase de capture accomplie, les divers pièges sont individualisés. Un système de by-pass permet de « pousser » chacune des cellules piégées dans une succession de « microréacteurs » isolés enchaînant phases de lyse, de transcription inverse, d’amplification des ARNs (WTA) et de collecte des acides nucléiques amplifiés.
Le schéma ci-dessous reprend les 2 phases principales permettant d’aboutir à l’analyse du transcriptome de cellules isolées :
(i) l’amplification d’ARN de cellules isolées
(ii) l’analyse de ces ARNs
Ainsi que le montre la vidéo promotionnelle ci-dessous le système est compact et se veut simple d’utilisation. Il va de soi, malgré tout, que pour optimiser vos chances de capturer un signal d’intérêt un marquage préalable cellules vivantes/mortes peut s’imposer. Ceci impose aussi d’observer la plaque microfluidique au microscope à fluorescence (en se hâtant quelque peu). Bien évidemment l’adjonction de marqueurs discriminants (anticorps couplés à un agent fluorescent) est la bienvenue.
Cette dernière vidéo aborde le « workflow » aboutissant à l’analyse du transcriptome monocellulaire par Biomark, il va de soi qu’il s’agit là d’une « suggestion de présentation » puisqu’il est possible d’analyser le transcriptome amplifié par le système C1™ Single-Cell Auto Prep System avec un grand nombre de systèmes de qPCR ou de séquençage haut-débit.
http://youtu.be/TF4NJRE4Xg4
L’analyse des biomarqueurs des cellules tumorales circulantes devient un élément majeur de la médecine personnalisée (lire L’enjeu des cellules tumorales circulantes). L’article d’Ashley A. Powell et al. (Single Cell Profiling of Circulating Tumor Cells: Transcriptional Heterogeneity and Diversity from Breast Cancer Cell Lines (Plos One, 2102)) montre l’application de ce système au niveau de l’analyse de « biopsie liquide« .
L’outil proposé par Fluidigm trouve d’ores et déjà des applications cliniques très concrètes : l’analyse de biomarqueurs des cellules tumorales circulantes à visée pronostique.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages

