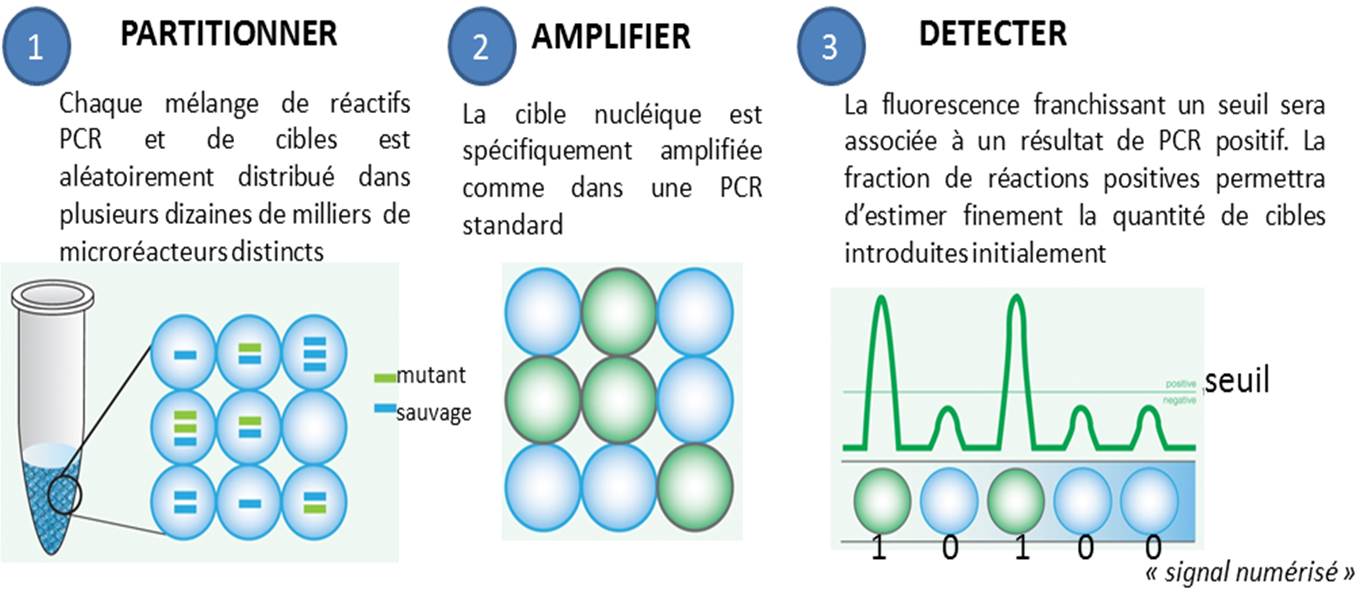
La PCR numérique : diviser pour plus de sensibilité
La PCR en point final, celle qui se termine souvent par un dépôt sur gel d’agarose, ainsi que la PCR quantitative, dans laquelle on suit l’évolution de la libération de molécules fluorescentes dont le décollage plus ou moins précoce dépend de la quantité de matrice initialement introduite, se voient déclinées en versions numériques. Par numérique est entendu ici, qu’une PCR peut prendre deux valeurs : 0 ou 1 (idéalement, 0 quand aucune cible n’est introduite au départ d’une PCR et que par voie de conséquence aucun signal issu d’amplification n’est observé, et 1 pour l’exact inverse).
Le principe de la PCR numérique est simple au niveau du concept mais techniquement beaucoup plus difficile à mettre en application. Il s’agit de multiplier le nombre de bio-réacteurs disponibles, ainsi, un échantillon sera partitionné en des milliers de compartiments distincts. Ensuite chaque compartiment sera considéré comme autant de réacteurs indépendants… donc en fin de PCR autant de réactions montrant oui ou non une amplification (autant de 0 et de 1), renvoyant à un système binaire. L’application de la Loi de Poisson permettra ensuite d’estimer très finement la quantité initiale de cibles présentes dans l’échantillon de départ.
Les avantages de la PCR numérique par rapport à la qPCR classique sont assez évidents :
– elle permet une quantification absolue sans établir une courbe standard préalable
– elle est plus sensible voire beaucoup plus sensible. Raindance annonce ainsi détecter un mutant parmi 250 000 molécules sauvages avec une limite inférieure de détection de 1 parmi 1.000.000
– elle est moins sensible aux inhibiteurs. Le fait de diluer la matrice ADN complexe de départ pour la répartir dans un nombre important de réacteurs permet de favoriser le départ de PCR
– elle est nettement plus précise. La technologie sera d’autant plus précise que le nombre de compartiments, le nombre de micro-réacteurs sera important
L’article « Digital PCR hits its stride » (La PCR numérique franchit un nouveau cap) de Monya Baker dans le Nature Methods (juin 2012) permet un aperçu de la technologie qui est passée du concept à des solutions commerciales qui se veulent de plus en plus accessibles.
Actuellement, plusieurs fournisseurs proposent des plateformes aux spécifications très différentes. Les quatre fournisseurs ci-dessous proposent des systèmes de PCR numérique basés sur de la microfluidique pour Fluidigm, des interfaces solides (type OpenArray) de Life Technologies et des micro-gouttes pour Raindance et Biorad.
Ainsi, que le mentionne Jim Huggett (chef d’équipe au LGC), cité dans la publication de Monya Baker (Digital PCR hits its stride), la PCR numérique est réservée à des utilisateurs experts et se trouve encore allouée à des applications spécialisées car encore beaucoup plus chère que la PCR quantitative (traditionnelle) qui reste adaptée à la majorité des applications.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages