L’art et la manière de développer une qPCR

La qPCR est une méthode permettant de doser la quantité d’acides nucléiques ciblés introduits dans une réaction de PCR. Pour des raisons de rapidité, de sensibilité et de coût, souvent l’option de travailler avec un agent intercalant (sans sonde) est choisie. La bonne vieille qPCR SybGreen nécessitant le seul design d’une paire d’amorces…
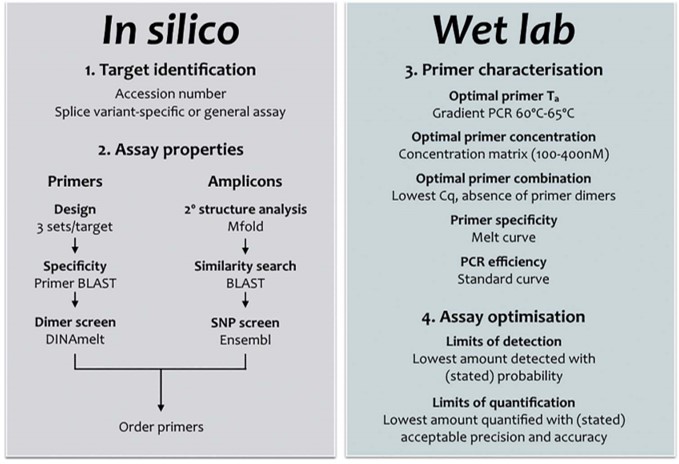
Simple ? Pas nécessairement, car cette approche, certainement plus que la version qPCR Taqman nécessite un travail in silico et de validation/optimisation expérimentales comme passages obligés. C’est ce que montre la publication de Stephen Bustin et Jim Huggett dans Biomolecular Detection and Quantification. Cette publication incontournable pour les férus de qPCR SybrGreen est un beau travail pour lequel la publication vous est mise à disposition en cliquant ci-dessous. On attend ardemment une déclinaison Taqman, HRM, MolecularBeacon de ce type de revues permettant de formaliser des procédures visant à optimiser l’approche d’optimisation.
La quantification par qPCR SybrGreen suppose une relation linéaire entre le logarithme de la quantité initiale introduite en PCR et la valeur Cq obtenue lors de l’amplification. Ceci permet de calculer l’efficacité d’amplification d’un test et de borner ses limites de détection et de quantification. Les caractéristiques d’un test qPCR (bien) optimisé sont les suivantes:
• Une excellentissime spécificité révélée par un pic unique lors de l’établissement de la courbe de fusion
• Une efficacité d’amplification élevée (95-105%)
• Une courbe étalon linéaire (R2 > 0,980)
• Une bonne répétabilité
• Peu ou prou de dimères d’amorces
Pour paraphraser la conclusion de l’article, afin de finir par convaincre de lire cet « essentiel » de la qPCR :
La conception, le design d’une PCR est souvent au cœur de tout projet de recherche visant à quantifier les acides nucléiques. Il doit être réalisé avec soin, mais peut être simplifié en suivant un flux de travail simple, comme décrit ci-dessus (cf. diagramme workflow design qPCR).
Cela signifie généralement une spécificité absolue, l’absence de structures en épingle à cheveux ou de potentielles dimérisations croisées. Une bonne conception des essais doit tenir compte de la structure de l’amplicon (paramètre souvent négligé) et veiller à ce que les cibles de l’amorce soient exempts de structure secondaire. Il existe de nombreuses opinions et lignes directrices; une recherche sur Internet pour les termes « qPCR Assay Design » renvoie 695.000 pages. Cependant, bon nombre de ceux-ci sont basés sur des mythes ou peuvent être appropriés pour la PCR mais nécessitent des modifications subtiles (ou moins subtiles) pour être utilisés pour développer une qPCR. Chaque « nouveau » dosage doit être correctement validé, la validation in silico servant de filtre initial pour éliminer des designs ne permettant pas d’aboutir à une bonne qPCR. L’optimisation et la validation empirique sont une partie essentielle, mais souvent négligée, de toute expérience qPCR. Cela s’applique aussi bien aux essais nouvellement conçus qu’aux essais obtenus en reprenant des amorces issues d’une publication, par exemple. Avec tant d’essais prêts à l’emploi, on peut se demander pourquoi quelqu’un voudrait se donner la peine de concevoir un autre essai. D’autant plus que l’on a l’impression que la conception de son propre test est beaucoup plus complexe et peu commode que de simplement l’acheter à un fournisseur commercial, qui en tout cas aura validé chacun de ses tests. Cette perception est erronée pour deux raisons:
1° il se peut que les amorces commerciales ou les conditions d’analyse n’aient pas été validées ou optimisées de façon expérimentale.
2° on ne peut pas présumer qu’un ensemble d’amorces produira les mêmes résultats dans des conditions expérimentales différentes puisque la performance du dosage peut varier selon les méthodes d’extraction utilisées pour purifier les acides nucléiques.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages